-検索条件
-検索結果
検索 (著者・登録者: nakanishi & h)の結果174件中、1から50件目までを表示しています

EMDB-36794: 
Cryo-EM structure of Na+,K+-ATPase alpha2 from Artemia salina in cation-free E2P form

PDB-8k1l: 
Cryo-EM structure of Na+,K+-ATPase alpha2 from Artemia salina in cation-free E2P form

EMDB-29033: 
Inactivate state of Maribacter polysiphoniae Argonuate (short pAgo system)

EMDB-29043: 
Structure of tetramerized MapSPARTA upon guide RNA-mediated target DNA binding

EMDB-29219: 
Raw map for structure of tetramerized short MapSPARTA (short prokaryotic Agronuate) system upon guide RNA-mediated target ssDNA binding

EMDB-29222: 
Focused refined map of TIR domain for MapSPARTA

EMDB-29223: 
Focused map for corner area4 of MapSPARTA

EMDB-29224: 
Local refined map of corner area 3 for the MapSPARTA

EMDB-29225: 
Focused refinement of corner area2 for MapSPARTA

EMDB-29226: 
Focused refinement for corner area1 of MapSPARTA

EMDB-40672: 
Symmetric dimer of MapSPARTA bound with gRNA/tDNA hybrid

EMDB-40673: 
Asymmetric dimer of MapSPARTA bound with gRNA/tDNA hybrid

EMDB-40679: 
Incomplete map of Maribacter polysiphoniae Argonaute (MapSPARTA) bound with guide RNA and target DNA duplex.

EMDB-40680: 
Tetramerized activation of MapSPARTA bound with NAD+

EMDB-40713: 
Monomeric MapSPARTA bound with guide RNA and target DNA hybrid

PDB-8fex: 
Inactivate state of Maribacter polysiphoniae Argonuate (short pAgo system)

PDB-8ffi: 
Structure of tetramerized MapSPARTA upon guide RNA-mediated target DNA binding

PDB-8sp0: 
Symmetric dimer of MapSPARTA bound with gRNA/tDNA hybrid

PDB-8sp3: 
Asymmetric dimer of MapSPARTA bound with gRNA/tDNA hybrid

PDB-8spo: 
Tetramerized activation of MapSPARTA bound with NAD+

PDB-8squ: 
Monomeric MapSPARTA bound with guide RNA and target DNA hybrid

EMDB-34752: 
F1 domain of FoF1-ATPase from Bacillus PS3,120 degrees,highATP

EMDB-34753: 
F1 domain of FoF1-ATPase from Bacillus PS3,step waiting,highATP

EMDB-34754: 
F1 domain of FoF1-ATPase from Bacillus PS3, 81 degrees, lowATP

EMDB-34755: 
F1 domain of FoF1-ATPase from Bacillus PS3,post-hyd,lowATP

EMDB-34770: 
FoF1-ATPase from Bacillus PS3,100 degrees,state3,highATP

EMDB-34771: 
FoF1-ATPase from Bacillus PS3,120 degrees,state1,highATP

EMDB-34772: 
FoF1-ATPase from Bacillus PS3,120 degrees,state2,highATP

EMDB-34773: 
FoF1-ATPase from Bacillus PS3,120 degrees,state3,highATP

EMDB-34774: 
FoF1-ATPase from Bacillus PS3,step waiting, state1,highATP

EMDB-34775: 
FoF1-ATPase from Bacillus PS3,step waiting,state2,highATP

EMDB-34776: 
FoF1-ATPase from Bacillus PS3,step waiting,state3,highATP

EMDB-34777: 
FoF1-ATPase from Bacillus PS3, 81 degrees, state1,lowATP

EMDB-34778: 
FoF1-ATPase from Bacillus PS3,post-hyd,state1,lowATP

EMDB-34779: 
FoF1-ATPase from Bacillus PS3,81 degrees,state2,lowATP

EMDB-34780: 
FoF1-ATPase from Bacillus PS3,81 degrees,state3,lowATP

EMDB-34762: 
FoF1-ATPase from Bacillus PS3,post-hyd,state1,highATP

EMDB-34362: 
1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

EMDB-34363: 
2 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

EMDB-34364: 
3 nucleotide-bound V1EG of V/A-ATPase from Thermus thermophilus.

EMDB-34365: 
2 sulfate-bound V1EG of V/A-ATPase from Thermus thermophilus.

EMDB-34366: 
1 sulfate and 1 ATP bound V1EG of V/A-ATPase from Thermus thermophilus.

PDB-8gxu: 
1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

PDB-8gxw: 
2 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus

PDB-8gxx: 
3 nucleotide-bound V1EG of V/A-ATPase from Thermus thermophilus.

PDB-8gxy: 
2 sulfate-bound V1EG of V/A-ATPase from Thermus thermophilus.

PDB-8gxz: 
1 sulfate and 1 ATP bound V1EG of V/A-ATPase from Thermus thermophilus.

EMDB-33200: 
Cryo-EM structure of human DNMT1 (aa:351-1616) in complex with ubiquitinated H3 and hemimethylated DNA analog (CXXC-ordered form)
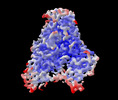
EMDB-33201: 
Cryo-EM structure of human DNMT1 (aa:351-1616) in complex with ubiquitinated H3 and hemimethylated DNA analog (CXXC-disordered form)
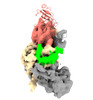
EMDB-33298: 
Cryo-EM map of apo-DNMT1 (aa:351-1616)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
