-検索条件
-検索結果
検索 (著者・登録者: de & carlo & s)の結果436件中、1から50件目までを表示しています
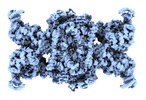
EMDB-19492: 
Trypanosoma brucei 3-methylcrotonyl-CoA carboxylase

PDB-8rth: 
Trypanosoma brucei 3-methylcrotonyl-CoA carboxylase

EMDB-42628: 
Structure of the insect gustatory receptor Gr9 from Bombyx mori

EMDB-42629: 
Structure of the insect gustatory receptor Gr9 from Bombyx mori in complex with D-fructose

EMDB-43548: 
Structure of the insect gustatory receptor Gr9 from Bombyx mori in complex with L-sorbose

PDB-8uvt: 
Structure of the insect gustatory receptor Gr9 from Bombyx mori

PDB-8uvu: 
Structure of the insect gustatory receptor Gr9 from Bombyx mori in complex with D-fructose

PDB-8vv3: 
Structure of the insect gustatory receptor Gr9 from Bombyx mori in complex with L-sorbose

EMDB-41423: 
Cryo-EM structure of DDB1dB:CRBN:Pomalidomide:SD40

EMDB-41424: 
Cryo-EM structure of DDB1dB:CRBN:PT-179:SD40, conformation 1

EMDB-41425: 
Cryo-EM structure of DDB1dB:CRBN:PT-179:SD40, conformation 2

EMDB-41777: 
Map from local refinement (focused on CRBN) of DDB1dB:CRBN:Pomalidomide:SD40

EMDB-41778: 
Map from local refinement (focused on CRBN) of DDB1dB:CRBN:PT-179:SD40, conformation 1

EMDB-41779: 
Map from local refinement (focused on CRBN) of DDB1dB:CRBN:PT-179:SD40, conformation 2

PDB-8tnp: 
Cryo-EM structure of DDB1dB:CRBN:Pomalidomide:SD40

PDB-8tnq: 
Cryo-EM structure of DDB1dB:CRBN:PT-179:SD40, conformation 1

PDB-8tnr: 
Cryo-EM structure of DDB1dB:CRBN:PT-179:SD40, conformation 2
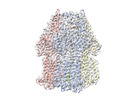
EMDB-17350: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

PDB-8p1i: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

EMDB-16453: 
SARS-CoV-2 Omicron Variant Spike Trimer in complex with three 17T2 Fabs

EMDB-16473: 
SARS-CoV-2 spike in complex with the 17T2 neutralizing antibody Fab fragment (local refinement of RBD and Fab)

PDB-8c89: 
SARS-CoV-2 spike in complex with the 17T2 neutralizing antibody Fab fragment (local refinement of RBD and Fab)

EMDB-42480: 
Cryo-EM reconstruction of Staphylococcus aureus Oleate hydratase (OhyA) dimer with an ordered C-terminal membrane-association domain

EMDB-42484: 
Cryo-EM reconstruction of Staphylococcus aureus oleate hydratase (OhyA) dimer with a disordered C-terminal membrane-association domain

PDB-8ur3: 
Cryo-EM reconstruction of Staphylococcus aureus Oleate hydratase (OhyA) dimer with an ordered C-terminal membrane-association domain

PDB-8ur6: 
Cryo-EM reconstruction of Staphylococcus aureus oleate hydratase (OhyA) dimer with a disordered C-terminal membrane-association domain

EMDB-40856: 
Single particle reconstruction of the human LINE-1 ORF2p without substrate (apo)
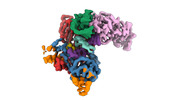
EMDB-40858: 
Structure of LINE-1 ORF2p with template:primer hybrid
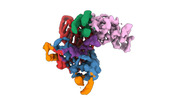
EMDB-40859: 
Structure of LINE-1 ORF2p with an oligo(A) template

PDB-8sxt: 
Structure of LINE-1 ORF2p with template:primer hybrid

PDB-8sxu: 
Structure of LINE-1 ORF2p with an oligo(A) template

EMDB-15413: 
Architecture of the ESCPE-1 membrane coat

PDB-8afz: 
Architecture of the ESCPE-1 membrane coat

EMDB-16799: 
Cryo-EM structure of the NINJ1 filament

PDB-8cqr: 
Cryo-EM structure of the NINJ1 filament

EMDB-40554: 
Cryo-EM Consensus map of the E. coli transcription-translation complex (RNAP in an anti-swiveled conformation)

EMDB-40178: 
Cryo-EM composited map of the E. coli transcription-translation complex (RNAP in an anti-swiveled conformation)

EMDB-14421: 
Structure of yeast RNA Polymerase III-Ty1 integrase complex at 2.6 A (focus subunit AC40).

EMDB-14468: 
Structure of yeast RNA Polymerase III-DNA-Ty1 integrase complex (Pol III-DNA-IN1) at 3.1 A

EMDB-14469: 
Structure of yeast RNA Polymerase III-Ty1 integrase complex at 2.9 A (focus subunit C11 terminal Zn-ribbon in the funnel pore).

EMDB-14470: 
Structure of yeast RNA Polymerase III-Ty1 integrase complex at 2.7 A (focus subunit C11, no C11 C-terminal Zn-ribbon in the funnel pore).

EMDB-16299: 
Structure of yeast RNA Polymerase III elongation complex at 3.3 A

PDB-7z0h: 
Structure of yeast RNA Polymerase III-Ty1 integrase complex at 2.6 A (focus subunit AC40).

PDB-7z2z: 
Structure of yeast RNA Polymerase III-DNA-Ty1 integrase complex (Pol III-DNA-IN1) at 3.1 A

PDB-7z30: 
Structure of yeast RNA Polymerase III-Ty1 integrase complex at 2.9 A (focus subunit C11 terminal Zn-ribbon in the funnel pore).

PDB-7z31: 
Structure of yeast RNA Polymerase III-Ty1 integrase complex at 2.7 A (focus subunit C11, no C11 C-terminal Zn-ribbon in the funnel pore).

PDB-8bws: 
Structure of yeast RNA Polymerase III elongation complex at 3.3 A
EMDB-29212: 
Cryo-EM structure of E. coli RNA polymerase backtracked elongation complex harboring a terminal mismatch

EMDB-29213: 
Cryo-EM structure of E. coli RNA polymerase Elongation complex in the Transcription-Translation Complex (RNAP in an anti-swiveled conformation)
EMDB-29214: 
Cryo-EM structure of E. coli 70S Ribosome containing mRNA and tRNA (in the transcription-translation complex)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
