+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6700 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Complex of Snf2-Nucleosome complex with Snf2 bound to SHL2 of the nucleosome | |||||||||
 マップデータ マップデータ | Density map of Snf2-nucleosome complex with Snf2 binding to SHL2 of the nucleosome. The map was sharpened by RELION post-processing, and the resolution was reported at 4.69A. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Snf2 / nucleosome / chromatin remodeling / STRUCTURAL PROTEIN-HYDROLASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of cell adhesion involved in single-species biofilm formation / positive regulation of mating type switching / positive regulation of invasive growth in response to glucose limitation / aggrephagy / DNA strand invasion / rDNA binding / SWI/SNF complex / ATP-dependent chromatin remodeler activity / ATP-dependent activity, acting on DNA / nucleosomal DNA binding ...positive regulation of cell adhesion involved in single-species biofilm formation / positive regulation of mating type switching / positive regulation of invasive growth in response to glucose limitation / aggrephagy / DNA strand invasion / rDNA binding / SWI/SNF complex / ATP-dependent chromatin remodeler activity / ATP-dependent activity, acting on DNA / nucleosomal DNA binding / cellular response to amino acid starvation / histone reader activity / transcription initiation-coupled chromatin remodeling / helicase activity / lysine-acetylated histone binding / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / DNA-templated DNA replication / structural constituent of chromatin / nucleosome / double-strand break repair / nucleosome assembly / RNA polymerase II-specific DNA-binding transcription factor binding / hydrolase activity / chromatin remodeling / protein heterodimerization activity / chromatin / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA binding / nucleoplasm / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.69 Å | |||||||||
 データ登録者 データ登録者 | Li M / Liu X / Xia X / Cheng Z / Li X | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: Mechanism of chromatin remodelling revealed by the Snf2-nucleosome structure. 著者: Xiaoyu Liu / Meijing Li / Xian Xia / Xueming Li / Zhucheng Chen /  要旨: Chromatin remodellers are helicase-like, ATP-dependent enzymes that alter chromatin structure and nucleosome positions to allow regulatory proteins access to DNA. Here we report the cryo-electron ...Chromatin remodellers are helicase-like, ATP-dependent enzymes that alter chromatin structure and nucleosome positions to allow regulatory proteins access to DNA. Here we report the cryo-electron microscopy structure of chromatin remodeller Switch/sucrose non-fermentable (SWI2/SNF2) from Saccharomyces cerevisiae bound to the nucleosome. The structure shows that the two core domains of Snf2 are realigned upon nucleosome binding, suggesting activation of the enzyme. The core domains contact each other through two induced Brace helices, which are crucial for coupling ATP hydrolysis to chromatin remodelling. Snf2 binds to the phosphate backbones of one DNA gyre of the nucleosome mainly through its helicase motifs within the major domain cleft, suggesting a conserved mechanism of substrate engagement across different remodellers. Snf2 contacts the second DNA gyre via a positively charged surface, providing a mechanism to anchor the remodeller at a fixed position of the nucleosome. Snf2 locally deforms nucleosomal DNA at the site of binding, priming the substrate for the remodelling reaction. Together, these findings provide mechanistic insights into chromatin remodelling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6700.map.gz emd_6700.map.gz | 4.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6700-v30.xml emd-6700-v30.xml emd-6700.xml emd-6700.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6700.png emd_6700.png | 139.5 KB | ||
| Filedesc metadata |  emd-6700.cif.gz emd-6700.cif.gz | 7 KB | ||
| その他 |  emd_6700_additional.map.gz emd_6700_additional.map.gz | 49.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6700 http://ftp.pdbj.org/pub/emdb/structures/EMD-6700 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6700 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6700 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6700_validation.pdf.gz emd_6700_validation.pdf.gz | 354.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6700_full_validation.pdf.gz emd_6700_full_validation.pdf.gz | 354.3 KB | 表示 | |
| XML形式データ |  emd_6700_validation.xml.gz emd_6700_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_6700_validation.cif.gz emd_6700_validation.cif.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6700 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6700 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6700 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6700 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6700.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6700.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density map of Snf2-nucleosome complex with Snf2 binding to SHL2 of the nucleosome. The map was sharpened by RELION post-processing, and the resolution was reported at 4.69A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
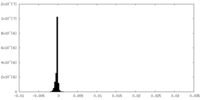
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened density map of Snf2-nucleosome complex with Snf2...
| ファイル | emd_6700_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened density map of Snf2-nucleosome complex with Snf2 binding to SHL2 of the nucleosome. The resolution was reported at 7.18A. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SHL2 complex
| 全体 | 名称: SHL2 complex |
|---|---|
| 要素 |
|
-超分子 #1: SHL2 complex
| 超分子 | 名称: SHL2 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Histone H3.2
| 分子 | 名称: Histone H3.2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 15.289904 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVGLFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA UniProtKB: Histone H3.2 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 11.263231 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG UniProtKB: Histone H4 |
-分子 #3: Histone H2A
| 分子 | 名称: Histone H2A / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.978241 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKT RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAVRN DEELNKLLGR VTIAQGGVLP NIQSVLLPKK TESSKSAKSK UniProtKB: Histone H2A |
-分子 #4: Histone H2B 1.1
| 分子 | 名称: Histone H2B 1.1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 13.524752 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AKSAPAPKKG SKKAVTKTQK KDGKKRRKTR KESYAIYVYK VLKQVHPDTG ISSKAMSIMN SFVNDVFERI AGEASRLAHY NKRSTITSR EIQTAVRLLL PGELAKHAVS EGTKAVTKYT SAK UniProtKB: Histone H2B 1.1 |
-分子 #7: Transcription regulatory protein SNF2
| 分子 | 名称: Transcription regulatory protein SNF2 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 85.802805 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AYIKLLDQTK DTRITHLLRQ TNAFLDSLTR AVKDQQKYTK EMIDSHIKEA SEEVDDLSMV PKMKDEEYDD DDDNSNVDYY NVAHRIKED IKKQPSILVG GTLKDYQIKG LQWMVSLFNN HLNGILADEM GLGKTIQTIS LLTYLYEMKN IRGPYLVIVP L STLSNWSS ...文字列: AYIKLLDQTK DTRITHLLRQ TNAFLDSLTR AVKDQQKYTK EMIDSHIKEA SEEVDDLSMV PKMKDEEYDD DDDNSNVDYY NVAHRIKED IKKQPSILVG GTLKDYQIKG LQWMVSLFNN HLNGILADEM GLGKTIQTIS LLTYLYEMKN IRGPYLVIVP L STLSNWSS EFAKWAPTLR TISFKGSPNE RKAKQAKIRA GEFDVVLTTF EYIIKERALL SKVKWVHMII DEGHRMKNAQ SK LSLTLNT HYHADYRLIL TGTPLQNNLP ELWALLNFVL PKIFNSVKSF DEWFNTPFAN TGGQDKIELS EEETLLVIRR LHK VLRPFL LRRLKKDVEK ELPDKVEKVV KCKMSALQQI MYQQMLKYRR LFIGDQNNKK MVGLRGFNNQ IMQLKKICNH PFVF EEVED QINPTRETND DIWRVAGKFE LLDRILPKLK ATGHRVLIFF QMTQIMDIME DFLRYINIKY LRLDGHTKSD ERSEL LRLF NAPDSEYLCF ILSTRAGGLG LNLQTADTVI IFDTDWNPHQ DLQAQDRAHR IGQKNEVRIL RLITTNSVEE VILERA YKK LDIDGKVIQA GKFDNKSTSE EQEALLRSLL DAEEERRKKR ESGVEEEEEL KDSEINEILA RNDEEMAVLT RMDEDRS KK EEELGVKSRL LEKSELPDIY SRDIGAELKR EESESAAVYN GRGARERKTA TYNDNMSEEQ WLRQFEVSDD EKNDKQAR K QRTKKEDKSE AIDG UniProtKB: Transcription regulatory protein SNF2 |
-分子 #5: DNA (167-MER)
| 分子 | 名称: DNA (167-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 51.381758 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC) ...文字列: (DA)(DT)(DC)(DG)(DA)(DG)(DA)(DA)(DT)(DC) (DC)(DC)(DG)(DG)(DT)(DG)(DC)(DC)(DG)(DA) (DG)(DG)(DC)(DC)(DG)(DC)(DT)(DC)(DA) (DA)(DT)(DT)(DG)(DG)(DT)(DC)(DG)(DT)(DA) (DG) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT) (DA)(DG)(DC)(DA)(DC)(DC)(DG)(DC)(DT)(DT) (DA)(DA) (DA)(DC)(DG)(DC)(DA)(DC)(DG) (DT)(DA)(DC)(DG)(DC)(DG)(DC)(DT)(DG)(DT) (DC)(DC)(DC) (DC)(DC)(DG)(DC)(DG)(DT) (DT)(DT)(DT)(DA)(DA)(DC)(DC)(DG)(DC)(DC) (DA)(DA)(DG)(DG) (DG)(DG)(DA)(DT)(DT) (DA)(DC)(DT)(DC)(DC)(DC)(DT)(DA)(DG)(DT) (DC)(DT)(DC)(DC)(DA) (DG)(DG)(DC)(DA) (DC)(DG)(DT)(DG)(DT)(DC)(DA)(DG)(DA)(DT) (DA)(DT)(DA)(DT)(DA)(DC) (DA)(DT)(DC) (DC)(DG)(DA)(DT)(DA)(DG)(DC)(DT)(DT)(DG) (DT)(DC)(DG)(DA)(DG)(DA)(DA) (DG)(DT) (DA)(DC)(DG)(DA)(DT) |
-分子 #6: DNA (167-MER)
| 分子 | 名称: DNA (167-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 51.723949 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DT)(DA)(DC)(DT)(DT)(DC) (DT)(DC)(DG)(DA)(DC)(DA)(DA)(DG)(DC)(DT) (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT) (DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA) (DC) (DA)(DC)(DG)(DT)(DG) ...文字列: (DA)(DT)(DC)(DG)(DT)(DA)(DC)(DT)(DT)(DC) (DT)(DC)(DG)(DA)(DC)(DA)(DA)(DG)(DC)(DT) (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT) (DA)(DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA) (DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT) (DG)(DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG) (DG)(DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC) (DC)(DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT) (DT)(DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG) (DG)(DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG) (DC)(DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC) (DG)(DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG) (DT)(DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC) (DT)(DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC) (DC)(DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC) (DG)(DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA) (DC)(DC)(DG)(DG)(DG)(DA)(DT) (DT)(DC) (DT)(DC)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil holey carbon grid / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 8.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER 詳細: A cylinder initial model generated with SPIDER and the negative staining model |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.69 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 1.4) / 使用した粒子像数: 90725 |
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)