+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of AHIR determined by cryoEM at 100 keV | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | OXIDOREDUCTASE | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ketol-acid reductoisomerase (NADP+) / ketol-acid reductoisomerase activity / L-valine biosynthetic process / isoleucine biosynthetic process / NADP binding / magnesium ion binding / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) | |||||||||||||||||||||
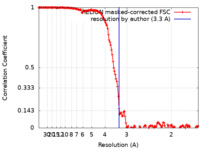
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | McMullan G / Naydenova K / Mihaylov D / Peet MJ / Wilson H / Yamashita K / Dickerson JL / Chen S / Cannone G / Lee Y ...McMullan G / Naydenova K / Mihaylov D / Peet MJ / Wilson H / Yamashita K / Dickerson JL / Chen S / Cannone G / Lee Y / Hutchings KA / Gittins O / Sobhy M / Wells T / El-Gomati MM / Dalby J / Meffert M / Schulze-Briese C / Henderson R / Russo CJ | |||||||||||||||||||||
| 資金援助 |  英国, 6件 英国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Structure determination by cryoEM at 100 keV. 著者: Greg McMullan / Katerina Naydenova / Daniel Mihaylov / Keitaro Yamashita / Mathew J Peet / Hugh Wilson / Joshua L Dickerson / Shaoxia Chen / Giuseppe Cannone / Yang Lee / Katherine A ...著者: Greg McMullan / Katerina Naydenova / Daniel Mihaylov / Keitaro Yamashita / Mathew J Peet / Hugh Wilson / Joshua L Dickerson / Shaoxia Chen / Giuseppe Cannone / Yang Lee / Katherine A Hutchings / Olivia Gittins / Mohamed A Sobhy / Torquil Wells / Mohamed M El-Gomati / Jason Dalby / Matthias Meffert / Clemens Schulze-Briese / Richard Henderson / Christopher J Russo /    要旨: Electron cryomicroscopy can, in principle, determine the structures of most biological molecules but is currently limited by access, specimen preparation difficulties, and cost. We describe a purpose- ...Electron cryomicroscopy can, in principle, determine the structures of most biological molecules but is currently limited by access, specimen preparation difficulties, and cost. We describe a purpose-built instrument operating at 100 keV-including advances in electron optics, detection, and processing-that makes structure determination fast and simple at a fraction of current costs. The instrument attains its theoretical performance limits, allowing atomic resolution imaging of gold test specimens and biological molecular structure determination in hours. We demonstrate its capabilities by determining the structures of eleven different specimens, ranging in size from 140 kDa to 2 MDa, using a fraction of the data normally required. CryoEM with a microscope designed specifically for high-efficiency, on-the-spot imaging of biological molecules will expand structural biology to a wide range of previously intractable problems. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17963.map.gz emd_17963.map.gz | 19.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17963-v30.xml emd-17963-v30.xml emd-17963.xml emd-17963.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17963_fsc.xml emd_17963_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17963.png emd_17963.png | 97.8 KB | ||
| マスクデータ |  emd_17963_msk_1.map emd_17963_msk_1.map | 282.6 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17963.cif.gz emd-17963.cif.gz | 6 KB | ||
| その他 |  emd_17963_half_map_1.map.gz emd_17963_half_map_1.map.gz emd_17963_half_map_2.map.gz emd_17963_half_map_2.map.gz | 225.4 MB 225.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17963 http://ftp.pdbj.org/pub/emdb/structures/EMD-17963 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17963 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17963 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17963_validation.pdf.gz emd_17963_validation.pdf.gz | 806.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17963_full_validation.pdf.gz emd_17963_full_validation.pdf.gz | 805.7 KB | 表示 | |
| XML形式データ |  emd_17963_validation.xml.gz emd_17963_validation.xml.gz | 22.4 KB | 表示 | |
| CIF形式データ |  emd_17963_validation.cif.gz emd_17963_validation.cif.gz | 29.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17963 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17963 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17963 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17963 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pveMC  8pv9C  8pvaC  8pvbC  8pvcC  8pvdC  8pvfC  8pvgC  8pvhC  8pviC  8pvjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17963.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17963.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8315 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17963_msk_1.map emd_17963_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17963_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17963_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Acetohydroxy acid isomeroreductase
| 全体 | 名称: Acetohydroxy acid isomeroreductase |
|---|---|
| 要素 |
|
-超分子 #1: Acetohydroxy acid isomeroreductase
| 超分子 | 名称: Acetohydroxy acid isomeroreductase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
-分子 #1: Ketol-acid reductoisomerase (NADP(+))
| 分子 | 名称: Ketol-acid reductoisomerase (NADP(+)) / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: ketol-acid reductoisomerase (NADP+) |
|---|---|
| 由来(天然) | 生物種:   Thermus thermophilus (バクテリア) Thermus thermophilus (バクテリア) |
| 分子量 | 理論値: 37.994246 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTGTMKIYYE HDADLGFILG KKVAVLGFGS QGHAHAHNLK DSGVDVRVGL RKGSRSWEKA EAAGLRVLPV AEAVREADVV MVLLPDEKQ AQVYREEVEP NLKEGGALAF AHGFNVHFGQ IKPRKDLDVW MVAPKGPGHL VRSEYGGGSN WSHPQFEKRP P GGSGVPAL ...文字列: MTGTMKIYYE HDADLGFILG KKVAVLGFGS QGHAHAHNLK DSGVDVRVGL RKGSRSWEKA EAAGLRVLPV AEAVREADVV MVLLPDEKQ AQVYREEVEP NLKEGGALAF AHGFNVHFGQ IKPRKDLDVW MVAPKGPGHL VRSEYGGGSN WSHPQFEKRP P GGSGVPAL VAVHQDASGS AFPTALAYAK AIGAARAGVI ATTFKDETET DLFGEQAVLC GGLTRLIRAG FETLVEAGYP PE MAYFETV HEVKLIVDLI YEAGLKGMRY SISNTAEYGD YTRGDLAVPL EETKRRMREI LRQIQSGEFA REWMLENQVG SPV LEANRK RWAAHPIEEV GSRLRAMMRS UniProtKB: Ketol-acid reductoisomerase (NADP(+)) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 1400/HR + YPS FEG |
|---|---|
| 撮影 | フィルム・検出器のモデル: DECTRIS SINGLA (1k x 1k) 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 100 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL |
|---|---|
| 得られたモデル |  PDB-8pve: |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)