+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3016 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM single particle 3D reconstruction of the native conformation of E. coli alpha-2-macroglobulin (ECAM) | |||||||||
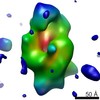
 マップデータ マップデータ | Reconstruction of the native conformation of the E.coli alpha-2-macroglobulin (ECAM) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  alpha-2-macroglobulin (Α2-マクログロブリン) / ECAM / alpha-2-macroglobulin (Α2-マクログロブリン) / ECAM /  peptidase inhibitor (プロテアーゼ阻害剤 (生物学)) peptidase inhibitor (プロテアーゼ阻害剤 (生物学)) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 endopeptidase inhibitor activity / protein homodimerization activity / endopeptidase inhibitor activity / protein homodimerization activity /  extracellular space / extracellular space /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 16.0 Å クライオ電子顕微鏡法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Garcia Ferrer I / Arede P / Gomez Blanco J / Luque D / Duquerroy S / Caston JR / Goulas T / Gomis Ruth FX | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2015 ジャーナル: Proc Natl Acad Sci U S A / 年: 2015タイトル: Structural and functional insights into Escherichia coli α2-macroglobulin endopeptidase snap-trap inhibition. 著者: Irene Garcia-Ferrer / Pedro Arêde / Josué Gómez-Blanco / Daniel Luque / Stephane Duquerroy / José R Castón / Theodoros Goulas / F Xavier Gomis-Rüth /   要旨: The survival of commensal bacteria requires them to evade host peptidases. Gram-negative bacteria from the human gut microbiome encode a relative of the human endopeptidase inhibitor, α2- ...The survival of commensal bacteria requires them to evade host peptidases. Gram-negative bacteria from the human gut microbiome encode a relative of the human endopeptidase inhibitor, α2-macroglobulin (α2M). Escherichia coli α2M (ECAM) is a ∼ 180-kDa multidomain membrane-anchored pan-peptidase inhibitor, which is cleaved by host endopeptidases in an accessible bait region. Structural studies by electron microscopy and crystallography reveal that this cleavage causes major structural rearrangement of more than half the 13-domain structure from a native to a compact induced form. It also exposes a reactive thioester bond, which covalently traps the peptidase. Subsequently, peptidase-laden ECAM is shed from the membrane and may dimerize. Trapped peptidases are still active except against very large substrates, so inhibition potentially prevents damage of large cell envelope components, but not host digestion. Mechanistically, these results document a novel monomeric "snap trap." | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3016.map.gz emd_3016.map.gz | 1.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3016-v30.xml emd-3016-v30.xml emd-3016.xml emd-3016.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-3016image500gris.png EMD-3016image500gris.png | 37.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3016 http://ftp.pdbj.org/pub/emdb/structures/EMD-3016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3016 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3016.map.gz / 形式: CCP4 / 大きさ: 1.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3016.map.gz / 形式: CCP4 / 大きさ: 1.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the native conformation of the E.coli alpha-2-macroglobulin (ECAM) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Native conformation of the E. coli alpha-2-macroglobulin (ECAM).
| 全体 | 名称: Native conformation of the E. coli alpha-2-macroglobulin (ECAM). |
|---|---|
| 要素 |
|
-超分子 #1000: Native conformation of the E. coli alpha-2-macroglobulin (ECAM).
| 超分子 | 名称: Native conformation of the E. coli alpha-2-macroglobulin (ECAM). タイプ: sample / ID: 1000 / 集合状態: Monomeric / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 180 KDa / 理論値: 180 KDa / 手法: Size exclusion chromatography. |
-分子 #1: alpha-2-macroglobulin
| 分子 | 名称: alpha-2-macroglobulin / タイプ: protein_or_peptide / ID: 1 / Name.synonym: ECAM 詳細: The expressed and purified sample contained the sequence GPM-A40 to P1653. コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 細胞中の位置: Periplasm Escherichia coli K-12 (大腸菌) / 細胞中の位置: Periplasm |
| 分子量 | 実験値: 180 KDa / 理論値: 180 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pCRI8b Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pCRI8b |
| 配列 | UniProtKB:  Α2-マクログロブリン Α2-マクログロブリン |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 10mM Tris-HCL, 75mM NaCl |
| グリッド | 詳細: Glow discharged carbon coated Cu/Rh 300 mesh Quantifoil R 1.2/1.3um grids. |
| 凍結 | 凍結剤: ETHANE / 装置: LEICA EM CPC 手法: Blotted for 1min before plunging with 5ul purified sample. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | OTHER |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 84269 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 日付 | 2014年6月10日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 実像数: 130 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each image |
|---|---|
| 最終 2次元分類 | クラス数: 64 |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: XMIPP3, EMAN2 / 使用した粒子像数: 46842 |
| 詳細 | The XMIPP automatic picking routine was used to select particles. |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An homology model based on the experimental coordinates of native ECAM fragments (PDB codes: 4ZJG, 4ZIU, 4ZJH) induced iECAM (PDB code: 4ZIQ) and the coordinates of native Salmonella enterica serovar typhimurium alpha-2-macroglobulin (PDB code: 4U48) was fitted as a rigid body into the density. Several rounds of manual flexible fitting for MG0-NIE domains were performed to improve the fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-5a42: |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An homology model based on the experimental coordinates of native ECAM fragments (PDB codes: 4ZJG, 4ZIU, 4ZJH) induced iECAM (PDB code: 4ZIQ) and the coordinates of native Salmonella enterica serovar typhimurium alpha-2-macroglobulin (PDB code: 4U48) was fitted as a rigid body into the density. Several rounds of manual flexible fitting for MG0-NIE domains were performed to improve the fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-5a42: |
-原子モデル構築 3
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An homology model based on the experimental coordinates of native ECAM fragments (PDB codes: 4ZJG, 4ZIU, 4ZJH) induced iECAM (PDB code: 4ZIQ) and the coordinates of native Salmonella enterica serovar typhimurium alpha-2-macroglobulin (PDB code: 4U48) was fitted as a rigid body into the density. Several rounds of manual flexible fitting for MG0-NIE domains were performed to improve the fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-5a42: |
-原子モデル構築 4
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An homology model based on the experimental coordinates of native ECAM fragments (PDB codes: 4ZJG, 4ZIU, 4ZJH) induced iECAM (PDB code: 4ZIQ) and the coordinates of native Salmonella enterica serovar typhimurium alpha-2-macroglobulin (PDB code: 4U48) was fitted as a rigid body into the density. Several rounds of manual flexible fitting for MG0-NIE domains were performed to improve the fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-5a42: |
-原子モデル構築 5
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An homology model based on the experimental coordinates of native ECAM fragments (PDB codes: 4ZJG, 4ZIU, 4ZJH) induced iECAM (PDB code: 4ZIQ) and the coordinates of native Salmonella enterica serovar typhimurium alpha-2-macroglobulin (PDB code: 4U48) was fitted as a rigid body into the density. Several rounds of manual flexible fitting for MG0-NIE domains were performed to improve the fitting. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC |
| 得られたモデル |  PDB-5a42: |
 ムービー
ムービー コントローラー
コントローラー