| 登録情報 | データベース: PDB / ID: 2rqu
|
|---|
| タイトル | Solution structure of the complex between the DDEF1 SH3 domain and the APC SAMP1 motif |
|---|
 要素 要素 | - 19-mer from Adenomatous polyposis coli protein
- DDEF1_SH3
|
|---|
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  SH3 domain (SH3ドメイン) / GAP / SAMP motif / SH3 domain (SH3ドメイン) / GAP / SAMP motif /  Tumor suppressor (がん抑制遺伝子) / Tumor suppressor (がん抑制遺伝子) /  Cell junction (細胞結合) / Disease mutation / Cell junction (細胞結合) / Disease mutation /  Phosphoprotein / Phosphoprotein /  Wnt signaling pathway (Wntシグナル経路) Wnt signaling pathway (Wntシグナル経路) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of membrane tubulation / APC truncation mutants are not K63 polyubiquitinated / regulation of microtubule-based movement / negative regulation of cell cycle G1/S phase transition / negative regulation of dendritic spine development / gamma-catenin binding / VxPx cargo-targeting to cilium / regulation of attachment of spindle microtubules to kinetochore / positive regulation of pseudopodium assembly / positive regulation of protein localization to centrosome ...positive regulation of membrane tubulation / APC truncation mutants are not K63 polyubiquitinated / regulation of microtubule-based movement / negative regulation of cell cycle G1/S phase transition / negative regulation of dendritic spine development / gamma-catenin binding / VxPx cargo-targeting to cilium / regulation of attachment of spindle microtubules to kinetochore / positive regulation of pseudopodium assembly / positive regulation of protein localization to centrosome / pattern specification process / bicellular tight junction assembly / negative regulation of cyclin-dependent protein serine/threonine kinase activity / negative regulation of microtubule depolymerization / protein localization to cilium / APC truncation mutants have impaired AXIN binding / AXIN missense mutants destabilize the destruction complex / Truncations of AMER1 destabilize the destruction complex / beta-catenin destruction complex / microtubule plus-end binding / heart valve development / regulation of microtubule-based process / protein kinase regulator activity / Beta-catenin phosphorylation cascade / Signaling by GSK3beta mutants / CTNNB1 S33 mutants aren't phosphorylated / CTNNB1 S37 mutants aren't phosphorylated / CTNNB1 S45 mutants aren't phosphorylated / CTNNB1 T41 mutants aren't phosphorylated /  catenin complex / catenin complex /  podosome / Wnt signalosome / Disassembly of the destruction complex and recruitment of AXIN to the membrane / cell fate specification / regulation of postsynapse organization / endocardial cushion morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / dynein complex binding / mitotic spindle assembly checkpoint signaling / podosome / Wnt signalosome / Disassembly of the destruction complex and recruitment of AXIN to the membrane / cell fate specification / regulation of postsynapse organization / endocardial cushion morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / dynein complex binding / mitotic spindle assembly checkpoint signaling /  phosphatidylserine binding / Apoptotic cleavage of cellular proteins / phosphatidylserine binding / Apoptotic cleavage of cellular proteins /  regulation of cell differentiation / phosphatidylinositol-3,4,5-trisphosphate binding / mitotic cytokinesis / regulation of cell differentiation / phosphatidylinositol-3,4,5-trisphosphate binding / mitotic cytokinesis /  cilium assembly / lateral plasma membrane / bicellular tight junction / cilium assembly / lateral plasma membrane / bicellular tight junction /  phosphatidylinositol-4,5-bisphosphate binding / phosphatidylinositol-4,5-bisphosphate binding /  GTPase activator activity / trans-Golgi network membrane / Deactivation of the beta-catenin transactivating complex / GTPase activator activity / trans-Golgi network membrane / Deactivation of the beta-catenin transactivating complex /  接着結合 / Degradation of beta-catenin by the destruction complex / negative regulation of canonical Wnt signaling pathway / 接着結合 / Degradation of beta-catenin by the destruction complex / negative regulation of canonical Wnt signaling pathway /  動原体 / 動原体 /  beta-catenin binding / beta-catenin binding /  Wntシグナル経路 / ruffle membrane / positive regulation of protein catabolic process / Ovarian tumor domain proteases / Wntシグナル経路 / ruffle membrane / positive regulation of protein catabolic process / Ovarian tumor domain proteases /  遊走 / insulin receptor signaling pathway / 遊走 / insulin receptor signaling pathway /  lamellipodium / lamellipodium /  nervous system development / positive regulation of cold-induced thermogenesis / nervous system development / positive regulation of cold-induced thermogenesis /  microtubule binding / proteasome-mediated ubiquitin-dependent protein catabolic process / protein-containing complex assembly / microtubule binding / proteasome-mediated ubiquitin-dependent protein catabolic process / protein-containing complex assembly /  微小管 / 微小管 /  樹状突起スパイン / 樹状突起スパイン /  細胞接着 / positive regulation of cell migration / 細胞接着 / positive regulation of cell migration /  cadherin binding / positive regulation of apoptotic process / negative regulation of cell population proliferation / cadherin binding / positive regulation of apoptotic process / negative regulation of cell population proliferation /  ゴルジ体 / ゴルジ体 /  中心体 / glutamatergic synapse / DNA damage response / 中心体 / glutamatergic synapse / DNA damage response /  ubiquitin protein ligase binding / ubiquitin protein ligase binding /  protein kinase binding / perinuclear region of cytoplasm / protein kinase binding / perinuclear region of cytoplasm /  ゴルジ体 / ゴルジ体 /  核質 / 核質 /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 ASAP1, BAR domain / ASAP1, SH3 domain /  ASAP, PH domain / Arf-GAP with SH3 domain, ANK repeat and PH domain-containing protein / BAR domain of APPL family / Adenomatous polyposis coli tumour suppressor protein / Armadillo-associated region on APC / Unstructured region on APC between 1st and 2nd catenin-bdg motifs / Unstructured region on APC between 1st two creatine-rich regions / Unstructured region on APC between APC_crr and SAMP ...ASAP1, BAR domain / ASAP1, SH3 domain / ASAP, PH domain / Arf-GAP with SH3 domain, ANK repeat and PH domain-containing protein / BAR domain of APPL family / Adenomatous polyposis coli tumour suppressor protein / Armadillo-associated region on APC / Unstructured region on APC between 1st and 2nd catenin-bdg motifs / Unstructured region on APC between 1st two creatine-rich regions / Unstructured region on APC between APC_crr and SAMP ...ASAP1, BAR domain / ASAP1, SH3 domain /  ASAP, PH domain / Arf-GAP with SH3 domain, ANK repeat and PH domain-containing protein / BAR domain of APPL family / Adenomatous polyposis coli tumour suppressor protein / Armadillo-associated region on APC / Unstructured region on APC between 1st and 2nd catenin-bdg motifs / Unstructured region on APC between 1st two creatine-rich regions / Unstructured region on APC between APC_crr and SAMP / Unstructured region on APC between SAMP and APC_crr / Unstructured region on APC between APC_crr regions 5 and 6 / ASAP, PH domain / Arf-GAP with SH3 domain, ANK repeat and PH domain-containing protein / BAR domain of APPL family / Adenomatous polyposis coli tumour suppressor protein / Armadillo-associated region on APC / Unstructured region on APC between 1st and 2nd catenin-bdg motifs / Unstructured region on APC between 1st two creatine-rich regions / Unstructured region on APC between APC_crr and SAMP / Unstructured region on APC between SAMP and APC_crr / Unstructured region on APC between APC_crr regions 5 and 6 /  Adenomatous polyposis coli protein repeat / Adenomatous polyposis coli protein repeat /  SAMP / EB-1 binding / SAMP / EB-1 binding /  Adenomatous polyposis coli protein basic domain / Adenomatous polyposis coli protein basic domain /  Adenomatous polyposis coli protein, 15 residue repeat / Adenomatous polyposis coli (APC) family / Adenomatous polyposis coli protein, 15 residue repeat / Adenomatous polyposis coli (APC) family /  家族性大腸腺腫症 / 家族性大腸腺腫症 /  Adenomatous polyposis coli, N-terminal dimerisation domain / APC, N-terminal coiled-coil domain superfamily / Adenomatous polyposis coli (APC) repeat / APC repeat / SAMP Motif / EB-1 Binding Domain / APC basic domain / APC 15 residue motif / Coiled-coil N-terminus of APC, dimerisation domain / Adenomatous polyposis coli (APC) repeat / Arf GTPase activating protein / ArfGAP domain superfamily / Putative GTPase activating protein for Arf / ARF GTPase-activating proteins domain profile. / Putative GTP-ase activating proteins for the small GTPase, ARF / Adenomatous polyposis coli, N-terminal dimerisation domain / APC, N-terminal coiled-coil domain superfamily / Adenomatous polyposis coli (APC) repeat / APC repeat / SAMP Motif / EB-1 Binding Domain / APC basic domain / APC 15 residue motif / Coiled-coil N-terminus of APC, dimerisation domain / Adenomatous polyposis coli (APC) repeat / Arf GTPase activating protein / ArfGAP domain superfamily / Putative GTPase activating protein for Arf / ARF GTPase-activating proteins domain profile. / Putative GTP-ase activating proteins for the small GTPase, ARF /  BAR domain / ARFGAP/RecO-like zinc finger / AH/BAR domain superfamily / Variant SH3 domain / Armadillo/plakoglobin ARM repeat profile. / Armadillo/beta-catenin-like repeat / Armadillo/beta-catenin-like repeats / BAR domain / ARFGAP/RecO-like zinc finger / AH/BAR domain superfamily / Variant SH3 domain / Armadillo/plakoglobin ARM repeat profile. / Armadillo/beta-catenin-like repeat / Armadillo/beta-catenin-like repeats /  Armadillo / SH3 Domains / Armadillo / SH3 Domains /  PH domain / PH domain profile. / PH domain / PH domain profile. /  Pleckstrin homology domain. / Pleckstrin homology domain. /  Pleckstrin homology domain / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats / Pleckstrin homology domain / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats /  Ankyrin repeat / Ankyrin repeat-containing domain superfamily / SH3 type barrels. / Src homology 3 domains / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. / Ankyrin repeat / Ankyrin repeat-containing domain superfamily / SH3 type barrels. / Src homology 3 domains / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. /  SH3ドメイン / Armadillo-like helical / PH-like domain superfamily / Armadillo-type fold / Roll / Mainly Beta類似検索 - ドメイン・相同性 SH3ドメイン / Armadillo-like helical / PH-like domain superfamily / Armadillo-type fold / Roll / Mainly Beta類似検索 - ドメイン・相同性  家族性大腸腺腫症 / Arf-GAP with SH3 domain, ANK repeat and PH domain-containing protein 1類似検索 - 構成要素 家族性大腸腺腫症 / Arf-GAP with SH3 domain, ANK repeat and PH domain-containing protein 1類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  溶液NMR / torsion angle dynamics, 溶液NMR / torsion angle dynamics,  simulated annealing simulated annealing |
|---|
| Model details | lowest energy, model 1 |
|---|
 データ登録者 データ登録者 | Kaieda, S. / Matsui, C. / Mimori-Kiyosue, Y. / Ikegami, T. |
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2010 ジャーナル: Biochemistry / 年: 2010
タイトル: Structural basis of the recognition of the SAMP motif of adenomatous polyposis coli by the Src-homology 3 domain.
著者: Kaieda, S. / Matsui, C. / Mimori-Kiyosue, Y. / Ikegami, T. |
|---|
| 履歴 | | 登録 | 2009年12月14日 | 登録サイト: BMRB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2010年7月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2024年5月1日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード SIGNALING PROTEIN /
SIGNALING PROTEIN /  SH3 domain (SH3ドメイン) / GAP / SAMP motif /
SH3 domain (SH3ドメイン) / GAP / SAMP motif /  Tumor suppressor (がん抑制遺伝子) /
Tumor suppressor (がん抑制遺伝子) /  Cell junction (細胞結合) / Disease mutation /
Cell junction (細胞結合) / Disease mutation /  Phosphoprotein /
Phosphoprotein /  Wnt signaling pathway (Wntシグナル経路)
Wnt signaling pathway (Wntシグナル経路) 機能・相同性情報
機能・相同性情報 catenin complex /
catenin complex /  podosome / Wnt signalosome / Disassembly of the destruction complex and recruitment of AXIN to the membrane / cell fate specification / regulation of postsynapse organization / endocardial cushion morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / dynein complex binding / mitotic spindle assembly checkpoint signaling /
podosome / Wnt signalosome / Disassembly of the destruction complex and recruitment of AXIN to the membrane / cell fate specification / regulation of postsynapse organization / endocardial cushion morphogenesis / negative regulation of G1/S transition of mitotic cell cycle / dynein complex binding / mitotic spindle assembly checkpoint signaling /  phosphatidylserine binding / Apoptotic cleavage of cellular proteins /
phosphatidylserine binding / Apoptotic cleavage of cellular proteins /  regulation of cell differentiation / phosphatidylinositol-3,4,5-trisphosphate binding / mitotic cytokinesis /
regulation of cell differentiation / phosphatidylinositol-3,4,5-trisphosphate binding / mitotic cytokinesis /  cilium assembly / lateral plasma membrane / bicellular tight junction /
cilium assembly / lateral plasma membrane / bicellular tight junction /  phosphatidylinositol-4,5-bisphosphate binding /
phosphatidylinositol-4,5-bisphosphate binding /  GTPase activator activity / trans-Golgi network membrane / Deactivation of the beta-catenin transactivating complex /
GTPase activator activity / trans-Golgi network membrane / Deactivation of the beta-catenin transactivating complex /  接着結合 / Degradation of beta-catenin by the destruction complex / negative regulation of canonical Wnt signaling pathway /
接着結合 / Degradation of beta-catenin by the destruction complex / negative regulation of canonical Wnt signaling pathway /  動原体 /
動原体 /  beta-catenin binding /
beta-catenin binding /  Wntシグナル経路 / ruffle membrane / positive regulation of protein catabolic process / Ovarian tumor domain proteases /
Wntシグナル経路 / ruffle membrane / positive regulation of protein catabolic process / Ovarian tumor domain proteases /  遊走 / insulin receptor signaling pathway /
遊走 / insulin receptor signaling pathway /  lamellipodium /
lamellipodium /  nervous system development / positive regulation of cold-induced thermogenesis /
nervous system development / positive regulation of cold-induced thermogenesis /  microtubule binding / proteasome-mediated ubiquitin-dependent protein catabolic process / protein-containing complex assembly /
microtubule binding / proteasome-mediated ubiquitin-dependent protein catabolic process / protein-containing complex assembly /  微小管 /
微小管 /  樹状突起スパイン /
樹状突起スパイン /  細胞接着 / positive regulation of cell migration /
細胞接着 / positive regulation of cell migration /  cadherin binding / positive regulation of apoptotic process / negative regulation of cell population proliferation /
cadherin binding / positive regulation of apoptotic process / negative regulation of cell population proliferation /  ゴルジ体 /
ゴルジ体 /  中心体 / glutamatergic synapse / DNA damage response /
中心体 / glutamatergic synapse / DNA damage response /  ubiquitin protein ligase binding /
ubiquitin protein ligase binding /  protein kinase binding / perinuclear region of cytoplasm /
protein kinase binding / perinuclear region of cytoplasm /  ゴルジ体 /
ゴルジ体 /  核質 /
核質 /  metal ion binding /
metal ion binding /  細胞核 /
細胞核 /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 溶液NMR / torsion angle dynamics,
溶液NMR / torsion angle dynamics,  simulated annealing
simulated annealing  データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2010
ジャーナル: Biochemistry / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2rqu.cif.gz
2rqu.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2rqu.ent.gz
pdb2rqu.ent.gz PDB形式
PDB形式 2rqu.json.gz
2rqu.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/rq/2rqu
https://data.pdbj.org/pub/pdb/validation_reports/rq/2rqu ftp://data.pdbj.org/pub/pdb/validation_reports/rq/2rqu
ftp://data.pdbj.org/pub/pdb/validation_reports/rq/2rqu リンク
リンク 集合体
集合体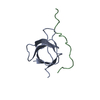
 要素
要素
 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: Q9ULH1
Escherichia coli (大腸菌) / 参照: UniProt: Q9ULH1
 Homo sapiens (ヒト) / 遺伝子: APC, DP2.5 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: APC, DP2.5 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P25054
Escherichia coli (大腸菌) / 参照: UniProt: P25054 溶液NMR
溶液NMR 試料調製
試料調製 解析
解析 simulated annealing / ソフトェア番号: 1
simulated annealing / ソフトェア番号: 1  ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj






