+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8718 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Domain identification of Penicillium Chrysogenum UGGT by labeling the protein with monovalent streptavidin | |||||||||
 マップデータ マップデータ | The negative stain EM map of Penicillium Chrysogenum UDP-glucose: glycoprotein glucosyltransferase (UGGT) | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報UDP-glucose:glycoprotein glucosyltransferase activity /  胞子 / anatomical structure development / protein glycosylation / 胞子 / anatomical structure development / protein glycosylation /  細胞分化 / 細胞分化 /  小胞体 小胞体類似検索 - 分子機能 | |||||||||
| 生物種 |   Penicillium chrysogenum (菌類) Penicillium chrysogenum (菌類) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 33.0 Å ネガティブ染色法 / 解像度: 33.0 Å | |||||||||
 データ登録者 データ登録者 | Calles-Garcia D / Yang M / Vargas J / Melero R / Soya N / Menade M / Ito Y / Lukacs G / Kollman J / Kozlov G / Gehring K | |||||||||
| 資金援助 |  カナダ, 1件 カナダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2017 ジャーナル: J Biol Chem / 年: 2017タイトル: Single-particle electron microscopy structure of UDP-glucose:glycoprotein glucosyltransferase suggests a selectivity mechanism for misfolded proteins. 著者: Daniel Calles-Garcia / Meng Yang / Naoto Soya / Roberto Melero / Marie Ménade / Yukishige Ito / Javier Vargas / Gergely L Lukacs / Justin M Kollman / Guennadi Kozlov / Kalle Gehring /     要旨: The enzyme UDP-glucose:glycoprotein glucosyltransferase (UGGT) mediates quality control of glycoproteins in the endoplasmic reticulum by attaching glucose to -linked glycan of misfolded proteins. As ...The enzyme UDP-glucose:glycoprotein glucosyltransferase (UGGT) mediates quality control of glycoproteins in the endoplasmic reticulum by attaching glucose to -linked glycan of misfolded proteins. As a sensor, UGGT ensures that misfolded proteins are recognized by the lectin chaperones and do not leave the secretory pathway. The structure of UGGT and the mechanism of its selectivity for misfolded proteins have been unknown for 25 years. Here, we used negative-stain electron microscopy and small-angle X-ray scattering to determine the structure of UGGT from at 18-Å resolution. Three-dimensional reconstructions revealed a cage-like structure with a large central cavity. Particle classification revealed flexibility that precluded determination of a high-resolution structure. Introduction of biotinylation sites into a fungal UGGT expressed in allowed identification of the catalytic and first thioredoxin-like domains. We also used hydrogen-deuterium exchange mass spectrometry to map the binding site of an accessory protein, Sep15, to the first thioredoxin-like domain. The UGGT structural features identified suggest that the central cavity contains the catalytic site and is lined with hydrophobic surfaces. This enhances the binding of misfolded substrates with exposed hydrophobic residues and excludes folded proteins with hydrophilic surfaces. In conclusion, we have determined the UGGT structure, which enabled us to develop a plausible functional model of the mechanism for UGGT's selectivity for misfolded glycoproteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8718.map.gz emd_8718.map.gz | 2.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8718-v30.xml emd-8718-v30.xml emd-8718.xml emd-8718.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8718_fsc.xml emd_8718_fsc.xml emd_8718_fsc_1.xml emd_8718_fsc_1.xml emd_8718_fsc_2.xml emd_8718_fsc_2.xml | 5.8 KB 5.8 KB 5.8 KB | 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8718.png emd_8718.png emd_8718_1.png emd_8718_1.png emd_8718_2.png emd_8718_2.png emd_8718_3.png emd_8718_3.png | 32.2 KB 32.2 KB 22.3 KB 27.1 KB | ||
| その他 |  emd_8718_additional_1.map.gz emd_8718_additional_1.map.gz emd_8718_additional_2.map.gz emd_8718_additional_2.map.gz | 10 MB 2.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8718 http://ftp.pdbj.org/pub/emdb/structures/EMD-8718 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8718 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8718 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8718.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8718.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The negative stain EM map of Penicillium Chrysogenum UDP-glucose: glycoprotein glucosyltransferase (UGGT) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: The negative stain EM map of Penicillium Chrysogenum...
| ファイル | emd_8718_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The negative stain EM map of Penicillium Chrysogenum UDP-glucose: glycoprotein glucosyltransferase (UGGT) with a biotinylation site inserted after lysine 261 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
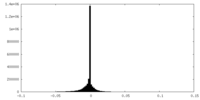
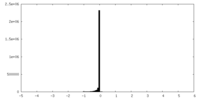
| 密度ヒストグラム |
-追加マップ: The negative stain EM map of Penicillium Chrysogenum...
| ファイル | emd_8718_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The negative stain EM map of Penicillium Chrysogenum UDP-glucose: glycoprotein glucosyltransferase (UGGT) with a biotinylation site inserted after aspartic acid 1377 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
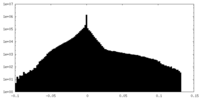
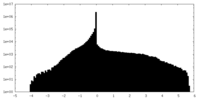
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : UDP-glucose:glycoprotein glucosyltransferase (UGGT)
| 全体 | 名称: UDP-glucose:glycoprotein glucosyltransferase (UGGT) |
|---|---|
| 要素 |
|
-超分子 #1: UDP-glucose:glycoprotein glucosyltransferase (UGGT)
| 超分子 | 名称: UDP-glucose:glycoprotein glucosyltransferase (UGGT) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Penicillium chrysogenum (菌類) Penicillium chrysogenum (菌類) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)組換細胞: SF9 |
| 分子量 | 実験値: 175 KDa |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.0075 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Buffer was filtered through a 0.22um filter | |||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: Freshly prepared uranyl formate at 0.75% was applied on the grids for 60s and then blotted | |||||||||
| グリッド | モデル: EMS / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 10.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 62000 Bright-field microscopy / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 62000 |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X