+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8288 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Putative tetramer of human CPAP (residues 897-1338) | |||||||||
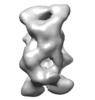
 マップデータ マップデータ | Contour level of 0.031 using Chimera | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 23.0 Å ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Alvarez-Cabrera AL / Delgado S / Gil D / Mortuza G / Montoya G / Sorzano CO / Tang TK / Carazo JM | |||||||||
 引用 引用 |  ジャーナル: Front Mol Biosci / 年: 2017 ジャーナル: Front Mol Biosci / 年: 2017タイトル: Electron Microscopy Structural Insights into CPAP Oligomeric Behavior: A Plausible Assembly Process of a Supramolecular Scaffold of the Centrosome. 著者: Ana L Alvarez-Cabrera / Sandra Delgado / David Gil-Carton / Gulnahar B Mortuza / Guillermo Montoya / Carlos O S Sorzano / Tang K Tang / Jose M Carazo /     要旨: Centrosomal P4.1-associated protein (CPAP) is a cell cycle regulated protein fundamental for centrosome assembly and centriole elongation. In humans, the region between residues 897-1338 of CPAP ...Centrosomal P4.1-associated protein (CPAP) is a cell cycle regulated protein fundamental for centrosome assembly and centriole elongation. In humans, the region between residues 897-1338 of CPAP mediates interactions with other proteins and includes a homodimerization domain. CPAP mutations cause primary autosomal recessive microcephaly and Seckel syndrome. Despite of the biological/clinical relevance of CPAP, its mechanistic behavior remains unclear and its C-terminus (the G-box/TCP domain) is the only part whose structure has been solved. This situation is perhaps due in part to the challenges that represent obtaining the protein in a soluble, homogeneous state for structural studies. Our work constitutes a systematic structural analysis on multiple oligomers of , using single-particle electron microscopy (EM) of negatively stained (NS) samples. Based on image classification into clearly different regular 3D maps (putatively corresponding to dimers and tetramers) and direct observation of individual images representing other complexes of CPAP (i.e., putative flexible monomers and higher-order multimers), we report a dynamic oligomeric behavior of this protein, where different homo-oligomers coexist in variable proportions. We propose that dimerization of the putative homodimer forms a putative tetramer which could be the structural unit for the scaffold that either tethers the pericentriolar material to centrioles or promotes procentriole elongation. A coarse fitting of atomic models into the NS 3D maps at resolutions around 20 Å is performed only to complement our experimental data, allowing us to hypothesize on the oligomeric composition of the different complexes. In this way, the current EM work represents an initial step toward the structural characterization of different oligomers of CPAP, suggesting further insights to understand how this protein works, contributing to the elucidation of control mechanisms for centriole biogenesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8288.map.gz emd_8288.map.gz | 126.4 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8288-v30.xml emd-8288-v30.xml emd-8288.xml emd-8288.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8288.png emd_8288.png | 32.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8288 http://ftp.pdbj.org/pub/emdb/structures/EMD-8288 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8288 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8288 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8288.map.gz / 形式: CCP4 / 大きさ: 844.7 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8288.map.gz / 形式: CCP4 / 大きさ: 844.7 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Contour level of 0.031 using Chimera | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.42 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Putative homotetramer of human CPAP (residues 897-1338)
| 全体 | 名称: Putative homotetramer of human CPAP (residues 897-1338) |
|---|---|
| 要素 |
|
-超分子 #1: Putative homotetramer of human CPAP (residues 897-1338)
| 超分子 | 名称: Putative homotetramer of human CPAP (residues 897-1338) タイプ: complex / ID: 1 / 親要素: 0 詳細: Human CPAP (residues 897-1338) expresed in E. coli and purified by Immobilized Metal Affinity Chromatography (IMAC) and Size Exclusion Chromatography (SEC) |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌)組換プラスミド: pST66Trc2-His (is a pET3a modified plasmid) |
| 分子量 | 実験値: 208 KDa |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
詳細: TCEP added to the buffer was prepared fresh | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate (0.75%) | |||||||||||||||
| グリッド | モデル: Quantifoil. Formvar/Carbon / 材質: COPPER / メッシュ: 400 / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: CONTINUOUS / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: FORMVAR / 支持フィルム - #1 - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | |||||||||||||||
| 詳細 | This human CPAP construct includes residues 897-1338 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.26 mm Bright-field microscopy / Cs: 2.26 mm |
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 9.0 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 3048 |
|---|---|
| CTF補正 | ソフトウェア - 名称: Xmipp (ver. 3.1) |
| 初期モデル | モデルのタイプ: OTHER 詳細: RANSAC algorithm (Bioinformatics. 2014 Oct 15;30(20):2891-8) |
| 初期 角度割当 | タイプ: OTHER / ソフトウェア - 名称: Xmipp (ver. 3.1) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 1.2) ソフトウェア - 詳細: Relion (accessed from Xmipp3.1) |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION (ver. 1.2) ソフトウェア - 詳細: Relion (accessed from Xmipp3.1) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: Xmipp (ver. 3.1) ソフトウェア - 詳細: Reconstruct Significant algorithm 使用した粒子像数: 3048 |
 ムービー
ムービー コントローラー
コントローラー