+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7wfw | ||||||
|---|---|---|---|---|---|---|---|
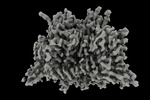
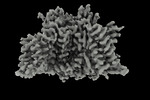
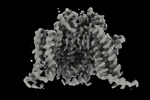
| タイトル | Apo human Nav1.8 | ||||||
 要素 要素 | Sodium channel protein type 10 subunit alpha ナトリウムチャネル ナトリウムチャネル | ||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  Voltage-gated sodium channel (ナトリウムチャネル) / Nav / Voltage-gated sodium channel (ナトリウムチャネル) / Nav /  activation (活性化) / selectivity activation (活性化) / selectivity | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bundle of His cell action potential / AV node cell action potential /  clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization / clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization /  知覚 / 知覚 /  voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity / voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity /  voltage-gated sodium channel activity ...bundle of His cell action potential / AV node cell action potential / voltage-gated sodium channel activity ...bundle of His cell action potential / AV node cell action potential /  clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization / clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization /  知覚 / 知覚 /  voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity / voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity /  voltage-gated sodium channel activity / regulation of monoatomic ion transmembrane transport / Interaction between L1 and Ankyrins / voltage-gated sodium channel activity / regulation of monoatomic ion transmembrane transport / Interaction between L1 and Ankyrins /  voltage-gated calcium channel complex / Phase 0 - rapid depolarisation / odontogenesis of dentin-containing tooth / calcium ion import across plasma membrane / sodium ion transmembrane transport / regulation of cardiac muscle contraction / voltage-gated calcium channel complex / Phase 0 - rapid depolarisation / odontogenesis of dentin-containing tooth / calcium ion import across plasma membrane / sodium ion transmembrane transport / regulation of cardiac muscle contraction /  regulation of heart rate / regulation of heart rate /  presynaptic membrane / transmembrane transporter binding / presynaptic membrane / transmembrane transporter binding /  神経繊維 / glutamatergic synapse / extracellular exosome / 神経繊維 / glutamatergic synapse / extracellular exosome /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | ||||||
 データ登録者 データ登録者 | Yan, N. / Pan, X.J. / Huang, X.S. / Huang, G.X. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structural basis for high-voltage activation and subtype-specific inhibition of human Na1.8. 著者: Xiaoshuang Huang / Xueqin Jin / Gaoxingyu Huang / Jian Huang / Tong Wu / Zhangqiang Li / Jiaofeng Chen / Fang Kong / Xiaojing Pan / Nieng Yan /  要旨: The dorsal root ganglia-localized voltage-gated sodium (Na) channel Na1.8 represents a promising target for developing next-generation analgesics. A prominent characteristic of Na1.8 is the ...The dorsal root ganglia-localized voltage-gated sodium (Na) channel Na1.8 represents a promising target for developing next-generation analgesics. A prominent characteristic of Na1.8 is the requirement of more depolarized membrane potential for activation. Here we present the cryogenic electron microscopy structures of human Na1.8 alone and bound to a selective pore blocker, A-803467, at overall resolutions of 2.7 to 3.2 Å. The first voltage-sensing domain (VSD) displays three different conformations. Structure-guided mutagenesis identified the extracellular interface between VSD and the pore domain (PD) to be a determinant for the high-voltage dependence of activation. A-803467 was clearly resolved in the central cavity of the PD, clenching S6. Our structure-guided functional characterizations show that two nonligand binding residues, Thr397 on S6 and Gly1406 on S6, allosterically modulate the channel's sensitivity to A-803467. Comparison of available structures of human Na channels suggests the extracellular loop region to be a potential site for developing subtype-specific pore-blocking biologics. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7wfw.cif.gz 7wfw.cif.gz | 215.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7wfw.ent.gz pdb7wfw.ent.gz | 164.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7wfw.json.gz 7wfw.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/wf/7wfw https://data.pdbj.org/pub/pdb/validation_reports/wf/7wfw ftp://data.pdbj.org/pub/pdb/validation_reports/wf/7wfw ftp://data.pdbj.org/pub/pdb/validation_reports/wf/7wfw | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  ナトリウムチャネル / Peripheral nerve sodium channel 3 / PN3 / hPN3 / Sodium channel protein type X subunit alpha / ...Peripheral nerve sodium channel 3 / PN3 / hPN3 / Sodium channel protein type X subunit alpha / Voltage-gated sodium channel subunit alpha Nav1.8 ナトリウムチャネル / Peripheral nerve sodium channel 3 / PN3 / hPN3 / Sodium channel protein type X subunit alpha / ...Peripheral nerve sodium channel 3 / PN3 / hPN3 / Sodium channel protein type X subunit alpha / Voltage-gated sodium channel subunit alpha Nav1.8分子量: 220903.500 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SCN10A / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SCN10A / 発現宿主:   Homo sapiens (ヒト) / 参照: UniProt: Q9Y5Y9 Homo sapiens (ヒト) / 参照: UniProt: Q9Y5Y9 |
|---|
-糖 , 2種, 5分子 
| #2: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose オリゴ糖 / 分子量: 424.401 Da / 分子数: 1 / 由来タイプ: 組換発現 オリゴ糖 / 分子量: 424.401 Da / 分子数: 1 / 由来タイプ: 組換発現 |
|---|---|
| #3: 糖 | ChemComp-NAG /  N-アセチルグルコサミン N-アセチルグルコサミン |
-非ポリマー , 4種, 17分子 






| #4: 化合物 | ChemComp-CLR /  コレステロール コレステロール#5: 化合物 | #6: 化合物 | ChemComp-LPE / #7: 化合物 |  ホスファチジルセリン ホスファチジルセリン |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: sodium channel II ナトリウムチャネル / タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT ナトリウムチャネル / タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1800 nm / 最小 デフォーカス(公称値): 1500 nm Bright-field microscopy / 最大 デフォーカス(公称値): 1800 nm / 最小 デフォーカス(公称値): 1500 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.18_3855: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
3次元再構成 | 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 410478 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj














