+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7tkf | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
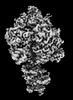
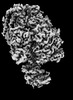
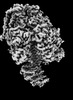
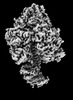
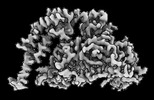
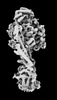
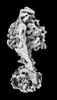
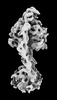
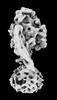
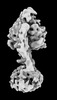
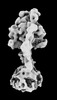
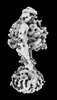
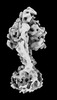
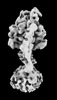
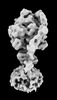
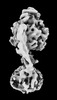
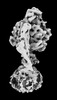
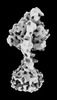
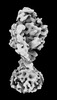
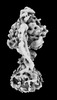
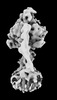
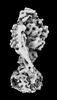
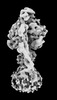
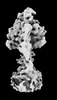
| タイトル | Yeast ATP synthase State 2binding(b) with 10 mM ATP backbone model | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / F1-ATPase / HYDROLASE (加水分解酵素) / F1-ATPase /  ATP Synthase (ATP合成酵素) / ATP Synthase (ATP合成酵素) /  Nanomotor Nanomotor | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cristae formation / mitochondrial proton-transporting ATP synthase, central stalk / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex / mitochondrial proton-transporting ATP synthase complex, catalytic sector F(1) / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis ...cristae formation / mitochondrial proton-transporting ATP synthase, central stalk / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex / mitochondrial proton-transporting ATP synthase complex, catalytic sector F(1) / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton motive force-driven ATP synthesis / proton transmembrane transporter activity / proton-transporting ATP synthase complex, catalytic core F(1) /  ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton transmembrane transport / proton-transporting ATP synthase activity, rotational mechanism / ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton transmembrane transport / proton-transporting ATP synthase activity, rotational mechanism /  ADP binding / ADP binding /  ミトコンドリア / protein-containing complex assembly / ミトコンドリア / protein-containing complex assembly /  ミトコンドリア内膜 / ミトコンドリア内膜 /  lipid binding / lipid binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.1 Å クライオ電子顕微鏡法 / 解像度: 7.1 Å | ||||||||||||
 データ登録者 データ登録者 | Guo, H. / Rubinstein, J.L. | ||||||||||||
| 資金援助 |  カナダ, 3件 カナダ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of ATP synthase under strain during catalysis. 著者: Hui Guo / John L Rubinstein /  要旨: ATP synthases are macromolecular machines consisting of an ATP-hydrolysis-driven F motor and a proton-translocation-driven F motor. The F and F motors oppose each other's action on a shared rotor ...ATP synthases are macromolecular machines consisting of an ATP-hydrolysis-driven F motor and a proton-translocation-driven F motor. The F and F motors oppose each other's action on a shared rotor subcomplex and are held stationary relative to each other by a peripheral stalk. Structures of resting mitochondrial ATP synthases revealed a left-handed curvature of the peripheral stalk even though rotation of the rotor, driven by either ATP hydrolysis in F or proton translocation through F, would apply a right-handed bending force to the stalk. We used cryoEM to image yeast mitochondrial ATP synthase under strain during ATP-hydrolysis-driven rotary catalysis, revealing a large deformation of the peripheral stalk. The structures show how the peripheral stalk opposes the bending force and suggests that during ATP synthesis proton translocation causes accumulation of strain in the stalk, which relaxes by driving the relative rotation of the rotor through six sub-steps within F, leading to catalysis. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7tkf.cif.gz 7tkf.cif.gz | 600 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7tkf.ent.gz pdb7tkf.ent.gz | 371.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7tkf.json.gz 7tkf.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tk/7tkf https://data.pdbj.org/pub/pdb/validation_reports/tk/7tkf ftp://data.pdbj.org/pub/pdb/validation_reports/tk/7tkf ftp://data.pdbj.org/pub/pdb/validation_reports/tk/7tkf | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | 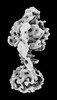 25967MC  7tjsC  7tjtC  7tjuC  7tjvC  7tjwC  7tjxC  7tjyC  7tjzC  7tk0C  7tk1C  7tk2C  7tk3C  7tk4C  7tk5C  7tk6C  7tk7C  7tk8C  7tk9C  7tkaC  7tkbC  7tkcC  7tkdC  7tkeC  7tkgC  7tkhC  7tkiC  7tkjC  7tkkC  7tklC  7tkmC  7tknC  7tkoC  7tkpC  7tkqC  7tkrC  7tksC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP synthase subunit ... , 13種, 26分子 0123456789ABCDEFGHIOTUVWXY
| #1: タンパク質 |  ATP合成酵素 / Lipid-binding protein / Oligomycin resistance protein 1 ATP合成酵素 / Lipid-binding protein / Oligomycin resistance protein 1分子量: 7762.375 Da / 分子数: 10 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P61829 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P61829#2: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 55007.402 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P07251 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P07251#3: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 51181.082 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00830, Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00830,  ATP合成酵素 ATP合成酵素#4: タンパク質 | |  ATP合成酵素 / F-ATPase gamma subunit ATP合成酵素 / F-ATPase gamma subunit分子量: 30657.160 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P38077 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P38077#5: タンパク質 | |  ATP合成酵素 / F-ATPase delta subunit ATP合成酵素 / F-ATPase delta subunit分子量: 14565.385 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: Q12165 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: Q12165#6: タンパク質 | |  ATP合成酵素 / ATPase subunit epsilon ATP合成酵素 / ATPase subunit epsilon分子量: 6618.359 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P21306 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P21306#7: タンパク質 | |  ATP合成酵素 / ATP synthase chain 5 / Oligomycin sensitivity conferral protein / OSCP ATP合成酵素 / ATP synthase chain 5 / Oligomycin sensitivity conferral protein / OSCP分子量: 20901.139 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P09457 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P09457#8: タンパク質 | |  ATP合成酵素 / F-ATPase protein 6 ATP合成酵素 / F-ATPase protein 6分子量: 27900.430 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P00854 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P00854#9: タンパク質 | |  ATP合成酵素 ATP合成酵素分子量: 23194.498 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P05626 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P05626#10: タンパク質 | |  ATP合成酵素 ATP合成酵素分子量: 19709.424 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P30902 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P30902#11: タンパク質 | |  ATP合成酵素 ATP合成酵素分子量: 10584.166 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: Q06405 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: Q06405#12: タンパク質 | |  ATP合成酵素 ATP合成酵素分子量: 10417.298 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: Q12349 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: Q12349#13: タンパク質 | |  ATP合成酵素 ATP合成酵素分子量: 6696.771 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P81450 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P81450 |
|---|
-タンパク質・ペプチド , 1種, 1分子 Z
| #14: タンパク質・ペプチド |  ATP合成酵素 / A6L / ATP-associated protein 1 / F-ATPase subunit 8 ATP合成酵素 / A6L / ATP-associated protein 1 / F-ATPase subunit 8分子量: 5825.215 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P00856 Saccharomyces cerevisiae (パン酵母) / 株: USY006 / 参照: UniProt: P00856 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Yeast ATP synthase State 2binding(b) with 10 mM ATPv backbone model タイプ: COMPLEX / Entity ID: all / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.61 MDa / 実験値: NO |
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: USY006 Saccharomyces cerevisiae (パン酵母) / 株: USY006 |
| 緩衝液 | pH: 7.4 |
| 試料 | 濃度: 15 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | グリッドの材料: COPPER/RHODIUM / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Homemade |
急速凍結 | 装置: LEICA EM GP / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 59000 X / 倍率(補正後): 103896 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1100 nm / Cs Bright-field microscopy / 倍率(公称値): 59000 X / 倍率(補正後): 103896 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1100 nm / Cs : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: ZEMLIN TABLEAU : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: ZEMLIN TABLEAU |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 11.9 sec. / 電子線照射量: 40 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 10037 |
| 画像スキャン | 横: 4096 / 縦: 4096 |
- 解析
解析
| ソフトウェア | 名称: UCSF ChimeraX / バージョン: 1.2/v9 / 分類: モデル構築 / URL: https://www.rbvi.ucsf.edu/chimerax/ / Os: macOS / タイプ: package | ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2534488 | ||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 7.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 9157 / アルゴリズム: BACK PROJECTION / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 2HLD Accession code: 2HLD / Source name: PDB / タイプ: experimental model |
 ムービー
ムービー コントローラー
コントローラー





























 PDBj
PDBj



