+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7b4g | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structural basis of reactivation of oncogenic p53 mutants by a small molecule: methylene quinuclidinone (MQ). Human p53DBD-R282W mutant bound to DNA: R282W-MQ (II) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  P53 (P53遺伝子) / P53 (P53遺伝子) /  TUMOR SUPPRESSOR (がん抑制遺伝子) / TUMOR SUPPRESSOR (がん抑制遺伝子) /  DNA BINDING PROTEIN (DNA結合タンパク質) / PROTEIN DNA COMPLEX / DNA BINDING PROTEIN (DNA結合タンパク質) / PROTEIN DNA COMPLEX /  MICHAEL ACCEPTOR / MICHAEL ACCEPTOR /  MICHAEL REACTION / PROTEIN-DRUG COMPLEX / PROTEIN-DNA-DRUG COMPLEX / LOOP-SHEET-HELIX MOTIF / DNA TARGET / ACTIVATOR / HOOGSTEEN BASE-PAIRING MICHAEL REACTION / PROTEIN-DRUG COMPLEX / PROTEIN-DNA-DRUG COMPLEX / LOOP-SHEET-HELIX MOTIF / DNA TARGET / ACTIVATOR / HOOGSTEEN BASE-PAIRING | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity ...Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity / negative regulation of helicase activity / regulation of cell cycle G2/M phase transition / intrinsic apoptotic signaling pathway in response to hypoxia / regulation of fibroblast apoptotic process / oxidative stress-induced premature senescence / oligodendrocyte apoptotic process / negative regulation of miRNA processing / positive regulation of thymocyte apoptotic process / glucose catabolic process to lactate via pyruvate /  regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death / regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death /  転写 (生物学) / bone marrow development / circadian behavior / T cell proliferation involved in immune response / regulation of mitochondrial membrane permeability involved in apoptotic process / histone deacetylase regulator activity / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of neuroblast proliferation / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / Regulation of TP53 Activity through Association with Co-factors / positive regulation of execution phase of apoptosis / mitochondrial DNA repair / T cell lineage commitment / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / positive regulation of cardiac muscle cell apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / entrainment of circadian clock by photoperiod / cardiac septum morphogenesis / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding / 転写 (生物学) / bone marrow development / circadian behavior / T cell proliferation involved in immune response / regulation of mitochondrial membrane permeability involved in apoptotic process / histone deacetylase regulator activity / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of neuroblast proliferation / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / Regulation of TP53 Activity through Association with Co-factors / positive regulation of execution phase of apoptosis / mitochondrial DNA repair / T cell lineage commitment / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / positive regulation of cardiac muscle cell apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / entrainment of circadian clock by photoperiod / cardiac septum morphogenesis / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding /  マイトファジー / negative regulation of telomere maintenance via telomerase / SUMOylation of transcription factors / intrinsic apoptotic signaling pathway by p53 class mediator / neuroblast proliferation / general transcription initiation factor binding / cellular response to actinomycin D / Transcriptional Regulation by VENTX / DNA damage response, signal transduction by p53 class mediator / response to X-ray / マイトファジー / negative regulation of telomere maintenance via telomerase / SUMOylation of transcription factors / intrinsic apoptotic signaling pathway by p53 class mediator / neuroblast proliferation / general transcription initiation factor binding / cellular response to actinomycin D / Transcriptional Regulation by VENTX / DNA damage response, signal transduction by p53 class mediator / response to X-ray /  ヘイフリック限界 / chromosome organization / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / ヘイフリック限界 / chromosome organization / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress /  gastrulation / cellular response to UV-C / response to inorganic substance / hematopoietic stem cell differentiation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of reactive oxygen species metabolic process / MDM2/MDM4 family protein binding / embryonic organ development / glial cell proliferation / gastrulation / cellular response to UV-C / response to inorganic substance / hematopoietic stem cell differentiation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of reactive oxygen species metabolic process / MDM2/MDM4 family protein binding / embryonic organ development / glial cell proliferation /  Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest / Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest /  somitogenesis / type II interferon-mediated signaling pathway / DNA damage response, signal transduction by p53 class mediator resulting in cell cycle arrest / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cardiac muscle cell apoptotic process / response to salt stress / transcription initiation-coupled chromatin remodeling / mitotic G1 DNA damage checkpoint signaling / Regulation of TP53 Activity through Acetylation somitogenesis / type II interferon-mediated signaling pathway / DNA damage response, signal transduction by p53 class mediator resulting in cell cycle arrest / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cardiac muscle cell apoptotic process / response to salt stress / transcription initiation-coupled chromatin remodeling / mitotic G1 DNA damage checkpoint signaling / Regulation of TP53 Activity through Acetylation類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.86 Å 分子置換 / 解像度: 1.86 Å | ||||||
 データ登録者 データ登録者 | Degtjarik, O. / Rozenberg, H. / Shakked, Z. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural basis of reactivation of oncogenic p53 mutants by a small molecule: methylene quinuclidinone (MQ). 著者: Degtjarik, O. / Golovenko, D. / Diskin-Posner, Y. / Abrahmsen, L. / Rozenberg, H. / Shakked, Z. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7b4g.cif.gz 7b4g.cif.gz | 145.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7b4g.ent.gz pdb7b4g.ent.gz | 90.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7b4g.json.gz 7b4g.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b4/7b4g https://data.pdbj.org/pub/pdb/validation_reports/b4/7b4g ftp://data.pdbj.org/pub/pdb/validation_reports/b4/7b4g ftp://data.pdbj.org/pub/pdb/validation_reports/b4/7b4g | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  6zncC  7b46C  7b47C  7b48C  7b49C  7b4aC  7b4bC  7b4cC  7b4dC  7b4eC  7b4fC  7b4hC  7b4nC  2ac0S C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 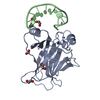
| ||||||||||||
| 単位格子 |
| ||||||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 / DNA鎖 , 2種, 2分子 AB
| #1: タンパク質 |  P53遺伝子 / Antigen NY-CO-13 / Phosphoprotein p53 / Tumor suppressor p53 P53遺伝子 / Antigen NY-CO-13 / Phosphoprotein p53 / Tumor suppressor p53分子量: 22534.598 Da / 分子数: 1 / 断片: p53 human DNA binding domain / 変異: R282W / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: TP53, P53 / プラスミド: pET-27-b / 発現宿主: Homo sapiens (ヒト) / 遺伝子: TP53, P53 / プラスミド: pET-27-b / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P04637 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: P04637 |
|---|---|
| #2: DNA鎖 | 分子量: 3664.380 Da / 分子数: 1 / 断片: p53 DNA target / 変異: ' / 由来タイプ: 合成 / 詳細: IDT: Integrated DNA Technology / 由来: (合成) synthetic construct (人工物) |
-非ポリマー , 4種, 324分子 






| #3: 化合物 | ChemComp-ZN / | ||||
|---|---|---|---|---|---|
| #4: 化合物 | ChemComp-EDO /  エチレングリコール エチレングリコール#5: 化合物 | ChemComp-PEG / |  ジエチレングリコール ジエチレングリコール#6: 水 | ChemComp-HOH / |  水 水 |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.22 Å3/Da / 溶媒含有率: 44.55 % / Mosaicity: 0.675 ° |
|---|---|
結晶化 | 温度: 292 K / 手法: 蒸発脱水法 詳細: Protein/DNA ratio 1:1.5, 0.2M Sodium Acetate, 20% w/v PEG 3350 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54178 Å 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54178 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: RIGAKU RAXIS IV++ / 検出器: IMAGE PLATE / 日付: 2009年3月10日 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: VariMax Optics / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 1.54178 Å / 相対比: 1 : 1.54178 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.86→40 Å / Num. obs: 18811 / % possible obs: 96.2 % / 冗長度: 3.5 % / Rmerge(I) obs: 0.088 / Rpim(I) all: 0.055 / Rrim(I) all: 0.104 / Χ2: 0.91 / Net I/σ(I): 7.7 / Num. measured all: 66152 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 2AC0 解像度: 1.86→34.29 Å / SU ML: 0.1977 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 19.6241 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 21.67 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.86→34.29 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Refine-ID: X-RAY DIFFRACTION
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





















































