+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6y8i | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Fragment KCL_I013 in complex with IL-1-beta | |||||||||
 要素 要素 | Interleukin-1 beta インターロイキン-1β インターロイキン-1β | |||||||||
 キーワード キーワード |  SIGNALING PROTEIN / FBDD / FRAGMENT BASED DRUG DESIGN / XCHEM / SIGNALING PROTEIN / FBDD / FRAGMENT BASED DRUG DESIGN / XCHEM /  IL1B (インターロイキン-1β) IL1B (インターロイキン-1β) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報smooth muscle adaptation / positive regulation of T cell mediated immunity / hyaluronan biosynthetic process / positive regulation of myosin light chain kinase activity / negative regulation of adiponectin secretion / negative regulation of lipid metabolic process / positive regulation of lipid catabolic process / negative regulation of glucose transmembrane transport / positive regulation of cell adhesion molecule production / positive regulation of T-helper 1 cell cytokine production ...smooth muscle adaptation / positive regulation of T cell mediated immunity / hyaluronan biosynthetic process / positive regulation of myosin light chain kinase activity / negative regulation of adiponectin secretion / negative regulation of lipid metabolic process / positive regulation of lipid catabolic process / negative regulation of glucose transmembrane transport / positive regulation of cell adhesion molecule production / positive regulation of T-helper 1 cell cytokine production / positive regulation of complement activation / positive regulation of calcidiol 1-monooxygenase activity / cellular response to interleukin-17 / sequestering of triglyceride / positive regulation of RNA biosynthetic process / monocyte aggregation / negative regulation of gap junction assembly / positive regulation of prostaglandin secretion / positive regulation of prostaglandin biosynthetic process / positive regulation of immature T cell proliferation in thymus / vascular endothelial growth factor production / positive regulation of neuroinflammatory response / fever generation / regulation of defense response to virus by host / positive regulation of fever generation / CLEC7A/inflammasome pathway / regulation of establishment of endothelial barrier / positive regulation of membrane protein ectodomain proteolysis / Interleukin-1 processing / response to carbohydrate /  interleukin-1 receptor binding / positive regulation of monocyte chemotactic protein-1 production / positive regulation of vascular endothelial growth factor receptor signaling pathway / positive regulation of granulocyte macrophage colony-stimulating factor production / negative regulation of synaptic transmission / regulation of canonical NF-kappaB signal transduction / positive regulation of heterotypic cell-cell adhesion / interleukin-1-mediated signaling pathway / positive regulation of p38MAPK cascade / interleukin-1 receptor binding / positive regulation of monocyte chemotactic protein-1 production / positive regulation of vascular endothelial growth factor receptor signaling pathway / positive regulation of granulocyte macrophage colony-stimulating factor production / negative regulation of synaptic transmission / regulation of canonical NF-kappaB signal transduction / positive regulation of heterotypic cell-cell adhesion / interleukin-1-mediated signaling pathway / positive regulation of p38MAPK cascade /  regulation of nitric-oxide synthase activity / cellular response to organic substance / response to ATP / Interleukin-10 signaling / regulation of insulin secretion / positive regulation of cell division / regulation of nitric-oxide synthase activity / cellular response to organic substance / response to ATP / Interleukin-10 signaling / regulation of insulin secretion / positive regulation of cell division /  regulation of neurogenesis / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / positive regulation of vascular endothelial growth factor production / positive regulation of epithelial to mesenchymal transition / ectopic germ cell programmed cell death / regulation of neurogenesis / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / positive regulation of vascular endothelial growth factor production / positive regulation of epithelial to mesenchymal transition / ectopic germ cell programmed cell death /  Pyroptosis / Pyroptosis /  Purinergic signaling in leishmaniasis infection / negative regulation of lipid catabolic process / positive regulation of glial cell proliferation / JNK cascade / Purinergic signaling in leishmaniasis infection / negative regulation of lipid catabolic process / positive regulation of glial cell proliferation / JNK cascade /  着床 / negative regulation of insulin receptor signaling pathway / extrinsic apoptotic signaling pathway in absence of ligand / positive regulation of T cell proliferation / positive regulation of interleukin-2 production / response to interleukin-1 / regulation of ERK1 and ERK2 cascade / 着床 / negative regulation of insulin receptor signaling pathway / extrinsic apoptotic signaling pathway in absence of ligand / positive regulation of T cell proliferation / positive regulation of interleukin-2 production / response to interleukin-1 / regulation of ERK1 and ERK2 cascade /  好中球 / negative regulation of MAP kinase activity / positive regulation of mitotic nuclear division / positive regulation of protein export from nucleus / 好中球 / negative regulation of MAP kinase activity / positive regulation of mitotic nuclear division / positive regulation of protein export from nucleus /  分泌 / 分泌 /  cytokine activity / positive regulation of interleukin-8 production / astrocyte activation / positive regulation of JNK cascade / positive regulation of MAP kinase activity / positive regulation of inflammatory response / negative regulation of neurogenesis / positive regulation of interleukin-6 production / Interleukin-1 signaling / cytokine-mediated signaling pathway / positive regulation of non-canonical NF-kappaB signal transduction / cellular response to mechanical stimulus / positive regulation of angiogenesis / positive regulation of nitric oxide biosynthetic process / positive regulation of DNA-binding transcription factor activity / positive regulation of type II interferon production / cellular response to xenobiotic stimulus / cytokine activity / positive regulation of interleukin-8 production / astrocyte activation / positive regulation of JNK cascade / positive regulation of MAP kinase activity / positive regulation of inflammatory response / negative regulation of neurogenesis / positive regulation of interleukin-6 production / Interleukin-1 signaling / cytokine-mediated signaling pathway / positive regulation of non-canonical NF-kappaB signal transduction / cellular response to mechanical stimulus / positive regulation of angiogenesis / positive regulation of nitric oxide biosynthetic process / positive regulation of DNA-binding transcription factor activity / positive regulation of type II interferon production / cellular response to xenobiotic stimulus /  integrin binding / cell-cell signaling / positive regulation of NF-kappaB transcription factor activity / positive regulation of canonical NF-kappaB signal transduction / Interleukin-4 and Interleukin-13 signaling / cellular response to lipopolysaccharide / response to lipopolysaccharide / integrin binding / cell-cell signaling / positive regulation of NF-kappaB transcription factor activity / positive regulation of canonical NF-kappaB signal transduction / Interleukin-4 and Interleukin-13 signaling / cellular response to lipopolysaccharide / response to lipopolysaccharide /  リソソーム / positive regulation of cell migration / defense response to Gram-positive bacterium / リソソーム / positive regulation of cell migration / defense response to Gram-positive bacterium /  免疫応答 / 免疫応答 /  炎症 / positive regulation of protein phosphorylation / protein domain specific binding / negative regulation of cell population proliferation / apoptotic process 炎症 / positive regulation of protein phosphorylation / protein domain specific binding / negative regulation of cell population proliferation / apoptotic process類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.46 Å 分子置換 / 解像度: 1.46 Å | |||||||||
 データ登録者 データ登録者 | De Nicola, G.F. / Nichols, C.E. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Fragment KCL_I013 in complex with IL-1-beta 著者: De Nicola, G.F. / Nichols, C.E. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6y8i.cif.gz 6y8i.cif.gz | 51.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6y8i.ent.gz pdb6y8i.ent.gz | 29.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6y8i.json.gz 6y8i.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/y8/6y8i https://data.pdbj.org/pub/pdb/validation_reports/y8/6y8i ftp://data.pdbj.org/pub/pdb/validation_reports/y8/6y8i ftp://data.pdbj.org/pub/pdb/validation_reports/y8/6y8i | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  5r85S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 |  インターロイキン-1β / IL-1 beta / Catabolin インターロイキン-1β / IL-1 beta / Catabolin分子量: 17395.832 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: IL1B, IL1F2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: IL1B, IL1F2 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P01584 Escherichia coli (大腸菌) / 参照: UniProt: P01584 |
|---|---|
| #2: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩 |
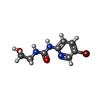
| #3: 化合物 | ChemComp-OGE / |
| #4: 水 | ChemComp-HOH /  水 水 |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.26 Å3/Da / 溶媒含有率: 62.27 % |
|---|---|
結晶化 | 温度: 291 K / 手法: 蒸気拡散法, シッティングドロップ法 / 詳細: 2.8M ammonium sulphate and 0.1M Tris pH7.9 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I03 / 波長: 0.9763 Å / ビームライン: I03 / 波長: 0.9763 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2019年9月17日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9763 Å / 相対比: 1 : 0.9763 Å / 相対比: 1 |
| 反射 | 解像度: 1.46→37.79 Å / Num. obs: 37604 / % possible obs: 97 % / 冗長度: 6.8 % / Biso Wilson estimate: 31.27 Å2 / CC1/2: 1 / Net I/σ(I): 16.9 |
| 反射 シェル | 解像度: 1.46→1.49 Å / Mean I/σ(I) obs: 1.1 / Num. unique obs: 1878 / CC1/2: 0.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5R85 解像度: 1.46→24.5 Å / SU ML: 0.2014 / 交差検証法: FREE R-VALUE / σ(F): 0.31 / 位相誤差: 26.7472
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 37.51 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.46→24.5 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj