| 登録情報 | データベース: PDB / ID: 6rx7
|
|---|
| タイトル | Structure of the KIV type 2 (KIV-2) domain of lipoprotein (a) |
|---|
 要素 要素 | Apolipoprotein(a) |
|---|
 キーワード キーワード | LIPID TRANSPORT / Lipoprotein (a) / kringle IV type 2 / KIV-2 / Lp(a). |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 リポタンパク質 / LDL remodeling / リポタンパク質 / LDL remodeling /  循環器 / lipid transport / 循環器 / lipid transport /  endopeptidase inhibitor activity / endopeptidase inhibitor activity /  apolipoprotein binding / apolipoprotein binding /  fibronectin binding / fibronectin binding /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process /  heparin binding ... heparin binding ... リポタンパク質 / LDL remodeling / リポタンパク質 / LDL remodeling /  循環器 / lipid transport / 循環器 / lipid transport /  endopeptidase inhibitor activity / endopeptidase inhibitor activity /  apolipoprotein binding / apolipoprotein binding /  fibronectin binding / fibronectin binding /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process /  heparin binding / heparin binding /  endopeptidase activity / endopeptidase activity /  signaling receptor binding / serine-type endopeptidase activity / signaling receptor binding / serine-type endopeptidase activity /  タンパク質分解 / タンパク質分解 /  extracellular space / extracellular region類似検索 - 分子機能 extracellular space / extracellular region類似検索 - 分子機能Plasminogen Kringle 4 / Plasminogen Kringle 4 /  Kringle domain / Kringle domain /  Kringle / Kringle /  Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. / Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. /  Kringle domain / Kringle-like fold ...Plasminogen Kringle 4 / Plasminogen Kringle 4 / Kringle domain / Kringle-like fold ...Plasminogen Kringle 4 / Plasminogen Kringle 4 /  Kringle domain / Kringle domain /  Kringle / Kringle /  Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. / Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. /  Kringle domain / Kringle-like fold / Kringle domain / Kringle-like fold /  Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, histidine active site /  Serine proteases, trypsin family, serine active site / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site / Peptidase S1A, chymotrypsin family /  Serine proteases, trypsin family, histidine active site. / Serine proteases, trypsin family, histidine active site. /  Serine proteases, trypsin domain profile. / Serine proteases, trypsin domain profile. /  Serine proteases, trypsin family, serine active site. / Trypsin-like serine protease / Serine proteases, trypsin family, serine active site. / Trypsin-like serine protease /  Serine proteases, trypsin domain / Serine proteases, trypsin domain /  トリプシン / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan / トリプシン / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan /  Βバレル / Mainly Beta類似検索 - ドメイン・相同性 Βバレル / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.63 Å 分子置換 / 解像度: 1.63 Å |
|---|
 データ登録者 データ登録者 | Santonastaso, A. / Maggi, M. / Scotti, C. |
|---|
| 資金援助 |  イタリア, 1件 イタリア, 1件 | 組織 | 認可番号 | 国 |
|---|
| Fondazione CARIPLO | 42657763 (2013-0380) |  イタリア イタリア |
|
|---|
 引用 引用 |  ジャーナル: J.Lipid Res. / 年: 2020 ジャーナル: J.Lipid Res. / 年: 2020
タイトル: High resolution structure of human apolipoprotein (a) kringle IV type 2: beyond the lysine binding site.
著者: Santonastaso, A. / Maggi, M. / De Jonge, H. / Scotti, C. |
|---|
| 履歴 | | 登録 | 2019年6月7日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2020年7月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年11月11日 | Group: Derived calculations
カテゴリ: pdbx_struct_assembly / pdbx_struct_assembly_gen / pdbx_struct_assembly_prop |
|---|
| 改定 1.2 | 2021年1月20日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.3 | 2024年1月24日 | Group: Advisory / Data collection ...Advisory / Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_unobs_or_zero_occ_atoms / pdbx_unobs_or_zero_occ_residues
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 リポタンパク質 / LDL remodeling /
リポタンパク質 / LDL remodeling /  循環器 / lipid transport /
循環器 / lipid transport /  endopeptidase inhibitor activity /
endopeptidase inhibitor activity /  apolipoprotein binding /
apolipoprotein binding /  fibronectin binding /
fibronectin binding /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process /
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process /  heparin binding ...
heparin binding ... リポタンパク質 / LDL remodeling /
リポタンパク質 / LDL remodeling /  循環器 / lipid transport /
循環器 / lipid transport /  endopeptidase inhibitor activity /
endopeptidase inhibitor activity /  apolipoprotein binding /
apolipoprotein binding /  fibronectin binding /
fibronectin binding /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process /
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ / lipid metabolic process /  heparin binding /
heparin binding /  endopeptidase activity /
endopeptidase activity /  signaling receptor binding / serine-type endopeptidase activity /
signaling receptor binding / serine-type endopeptidase activity /  タンパク質分解 /
タンパク質分解 /  extracellular space / extracellular region
extracellular space / extracellular region
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.63 Å
分子置換 / 解像度: 1.63 Å  データ登録者
データ登録者 イタリア, 1件
イタリア, 1件  引用
引用 ジャーナル: J.Lipid Res. / 年: 2020
ジャーナル: J.Lipid Res. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6rx7.cif.gz
6rx7.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6rx7.ent.gz
pdb6rx7.ent.gz PDB形式
PDB形式 6rx7.json.gz
6rx7.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/rx/6rx7
https://data.pdbj.org/pub/pdb/validation_reports/rx/6rx7 ftp://data.pdbj.org/pub/pdb/validation_reports/rx/6rx7
ftp://data.pdbj.org/pub/pdb/validation_reports/rx/6rx7 リンク
リンク 集合体
集合体
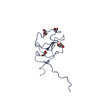
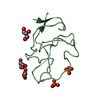

 要素
要素
 Homo sapiens (ヒト) / 遺伝子: LPA / プラスミド: pET45b(+) / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: LPA / プラスミド: pET45b(+) / 発現宿主: 
 Escherichia coli BL21(DE3) (大腸菌)
Escherichia coli BL21(DE3) (大腸菌) 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; セリンエンドペプチターゼ グリセリン
グリセリン 硫酸塩
硫酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: MASSIF-1 / 波長: 0.966 Å
/ ビームライン: MASSIF-1 / 波長: 0.966 Å : 0.966 Å / 相対比: 1
: 0.966 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー





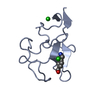








 PDBj
PDBj






