+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6kpc | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of an agonist bound GPCR | |||||||||
 要素 要素 | Cannabinoid receptor 2,Endolysin | |||||||||
 キーワード キーワード | MEMBRANE PROTEIN / GPCR / LCP / agonist | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cannabinoid receptor activity / negative regulation of mast cell activation / negative regulation of synaptic transmission, GABAergic / negative regulation of action potential / Class A/1 (Rhodopsin-like receptors) / regulation of metabolic process / leukocyte chemotaxis / extrinsic component of cytoplasmic side of plasma membrane / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / viral release from host cell by cytolysis ...cannabinoid receptor activity / negative regulation of mast cell activation / negative regulation of synaptic transmission, GABAergic / negative regulation of action potential / Class A/1 (Rhodopsin-like receptors) / regulation of metabolic process / leukocyte chemotaxis / extrinsic component of cytoplasmic side of plasma membrane / G protein-coupled receptor signaling pathway, coupled to cyclic nucleotide second messenger / viral release from host cell by cytolysis / response to amphetamine / peptidoglycan catabolic process / adenylate cyclase-activating G protein-coupled receptor signaling pathway / cell wall macromolecule catabolic process / lysozyme / lysozyme activity / G alpha (i) signalling events / perikaryon / host cell cytoplasm / response to lipopolysaccharide / defense response to bacterium / inflammatory response / immune response / dendrite / endoplasmic reticulum / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) Enterobacteria phage RB59 (ファージ) Enterobacteria phage RB59 (ファージ) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.2 Å 分子置換 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Li, X.T. / Hua, T. / Wu, L.J. / Makriyannis, A. / Wu, M. / Liu, Z.J. | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2020 ジャーナル: Cell / 年: 2020タイトル: Activation and Signaling Mechanism Revealed by Cannabinoid Receptor-G Complex Structures. 著者: Tian Hua / Xiaoting Li / Lijie Wu / Christos Iliopoulos-Tsoutsouvas / Yuxia Wang / Meng Wu / Ling Shen / Christina A Brust / Spyros P Nikas / Feng Song / Xiyong Song / Shuguang Yuan / ...著者: Tian Hua / Xiaoting Li / Lijie Wu / Christos Iliopoulos-Tsoutsouvas / Yuxia Wang / Meng Wu / Ling Shen / Christina A Brust / Spyros P Nikas / Feng Song / Xiyong Song / Shuguang Yuan / Qianqian Sun / Yiran Wu / Shan Jiang / Travis W Grim / Othman Benchama / Edward L Stahl / Nikolai Zvonok / Suwen Zhao / Laura M Bohn / Alexandros Makriyannis / Zhi-Jie Liu /   要旨: Human endocannabinoid systems modulate multiple physiological processes mainly through the activation of cannabinoid receptors CB1 and CB2. Their high sequence similarity, low agonist selectivity, ...Human endocannabinoid systems modulate multiple physiological processes mainly through the activation of cannabinoid receptors CB1 and CB2. Their high sequence similarity, low agonist selectivity, and lack of activation and G protein-coupling knowledge have hindered the development of therapeutic applications. Importantly, missing structural information has significantly held back the development of promising CB2-selective agonist drugs for treating inflammatory and neuropathic pain without the psychoactivity of CB1. Here, we report the cryoelectron microscopy structures of synthetic cannabinoid-bound CB2 and CB1 in complex with G, as well as agonist-bound CB2 crystal structure. Of important scientific and therapeutic benefit, our results reveal a diverse activation and signaling mechanism, the structural basis of CB2-selective agonists design, and the unexpected interaction of cholesterol with CB1, suggestive of its endogenous allosteric modulating role. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6kpc.cif.gz 6kpc.cif.gz | 197.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6kpc.ent.gz pdb6kpc.ent.gz | 154.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6kpc.json.gz 6kpc.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6kpc_validation.pdf.gz 6kpc_validation.pdf.gz | 671.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6kpc_full_validation.pdf.gz 6kpc_full_validation.pdf.gz | 677.3 KB | 表示 | |
| XML形式データ |  6kpc_validation.xml.gz 6kpc_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  6kpc_validation.cif.gz 6kpc_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kp/6kpc https://data.pdbj.org/pub/pdb/validation_reports/kp/6kpc ftp://data.pdbj.org/pub/pdb/validation_reports/kp/6kpc ftp://data.pdbj.org/pub/pdb/validation_reports/kp/6kpc | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 56209.934 Da / 分子数: 1 / 変異: G78L,T127A,T153L,G210A,C54T,C97A,R242E,G304E / 由来タイプ: 組換発現 詳細: CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, ...詳細: CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion,CB2, T4 lysozyme, CB2 fusion 由来: (組換発現)  Homo sapiens (ヒト), (組換発現) Homo sapiens (ヒト), (組換発現)  Enterobacteria phage RB59 (ファージ) Enterobacteria phage RB59 (ファージ)遺伝子: CNR2, CB2A, CB2B, e, RB59_126 発現宿主:  参照: UniProt: P34972, UniProt: A0A097J809, lysozyme |
|---|---|
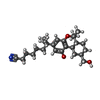
| #2: 化合物 | ChemComp-E3R / |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.31 Å3/Da / 溶媒含有率: 67.19 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 詳細: 100 mM HEPES sodium pH 7.0, 25% PEG 400, 220 mM Sodium sulfate decahydrate |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: Y | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL41XU / 波長: 1 Å / ビームライン: BL41XU / 波長: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2018年10月19日 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 1 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 3.2→44.78 Å / Num. obs: 11476 / % possible obs: 87.6 % / 冗長度: 2.847 % / Biso Wilson estimate: 96.93 Å2 / CC1/2: 0.986 / Rmerge(I) obs: 0.171 / Rrim(I) all: 0.2 / Χ2: 0.994 / Net I/σ(I): 4.26 / Num. measured all: 32673 / Scaling rejects: 21 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Serial crystallography sample delivery | 手法: fixed target |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 5zty 解像度: 3.2→44.78 Å / Cor.coef. Fo:Fc: 0.905 / Cor.coef. Fo:Fc free: 0.871 / 交差検証法: THROUGHOUT / σ(F): 0 / SU Rfree Blow DPI: 0.48
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 262.08 Å2 / Biso mean: 118.21 Å2 / Biso min: 11.04 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.53 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 3.2→44.78 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 3.2→3.5 Å / Rfactor Rfree error: 0 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj