+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5uja | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of bovine multidrug resistance protein 1 (MRP1) bound to leukotriene C4 | ||||||||||||
 要素 要素 | bovine multidrug resistance protein 1 (MRP1),Multidrug resistance-associated protein 1 | ||||||||||||
 キーワード キーワード |  PROTEIN TRANSPORT / PROTEIN TRANSPORT /  ABC transporter / ABC transporter /  multidrug resistance (多剤耐性) / multidrug resistance (多剤耐性) /  leukotriene C4 / LTC4 leukotriene C4 / LTC4 | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Heme degradation / Synthesis of Leukotrienes (LT) and Eoxins (EX) / cyclic nucleotide transport / Transport of RCbl within the body / Paracetamol ADME / leukotriene transport / glutathione transmembrane transporter activity / glutathione transmembrane transport / ABC-type glutathione-S-conjugate transporter / ABC-type glutathione S-conjugate transporter activity ...Heme degradation / Synthesis of Leukotrienes (LT) and Eoxins (EX) / cyclic nucleotide transport / Transport of RCbl within the body / Paracetamol ADME / leukotriene transport / glutathione transmembrane transporter activity / glutathione transmembrane transport / ABC-type glutathione-S-conjugate transporter / ABC-type glutathione S-conjugate transporter activity / ABC-family proteins mediated transport / Cytoprotection by HMOX1 / ABC-type xenobiotic transporter / ABC-type xenobiotic transporter activity / xenobiotic transport / lipid transport / xenobiotic transmembrane transporter activity / ABC-type transporter activity / positive regulation of inflammatory response / basolateral plasma membrane / response to xenobiotic stimulus /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding ATP binding類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Bos taurus (ウシ) Bos taurus (ウシ) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.34 Å クライオ電子顕微鏡法 / 解像度: 3.34 Å | ||||||||||||
 データ登録者 データ登録者 | Johnson, Z.L. / Chen, J. | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Structural Basis of Substrate Recognition by the Multidrug Resistance Protein MRP1. 著者: Zachary Lee Johnson / Jue Chen /  要旨: The multidrug resistance protein MRP1 is an ATP-binding cassette (ABC) transporter that confers resistance to many anticancer drugs and plays a role in the disposition and efficacy of several ...The multidrug resistance protein MRP1 is an ATP-binding cassette (ABC) transporter that confers resistance to many anticancer drugs and plays a role in the disposition and efficacy of several opiates, antidepressants, statins, and antibiotics. In addition, MRP1 regulates redox homeostasis, inflammation, and hormone secretion. Using electron cryomicroscopy, we determined the molecular structures of bovine MRP1 in two conformations: an apo form at 3.5 Å without any added substrate and a complex form at 3.3 Å with one of its physiological substrates, leukotriene C. These structures show that by forming a single bipartite binding site, MRP1 can recognize a spectrum of substrates with different chemical structures. We also observed large conformational changes induced by leukotriene C, explaining how substrate binding primes the transporter for ATP hydrolysis. Structural comparison of MRP1 and P-glycoprotein advances our understanding of the common and unique properties of these two important molecules in multidrug resistance to chemotherapy. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5uja.cif.gz 5uja.cif.gz | 265.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5uja.ent.gz pdb5uja.ent.gz | 203.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5uja.json.gz 5uja.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/uj/5uja https://data.pdbj.org/pub/pdb/validation_reports/uj/5uja ftp://data.pdbj.org/pub/pdb/validation_reports/uj/5uja ftp://data.pdbj.org/pub/pdb/validation_reports/uj/5uja | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  8560MC  8559C  5uj9C C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10813 (タイトル: Cryo-electron microscopy reconstruction of leukotriene C4 bound bovine MRP1 EMPIAR-10813 (タイトル: Cryo-electron microscopy reconstruction of leukotriene C4 bound bovine MRP1Data size: 938.2 Data #1: Unaligned and uncorrected multiframe movies of bovine leukotriene C4 bound MRP1 [micrographs - multiframe]) |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 159701.922 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Bos taurus (ウシ) / 遺伝子: ABCC1, ABCC1, MRP1 / 細胞株 (発現宿主): HEK293S GntI- / 発現宿主: Bos taurus (ウシ) / 遺伝子: ABCC1, ABCC1, MRP1 / 細胞株 (発現宿主): HEK293S GntI- / 発現宿主:   Homo sapiens (ヒト) / 参照: UniProt: Q8HXQ5 Homo sapiens (ヒト) / 参照: UniProt: Q8HXQ5 |
|---|---|
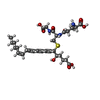
| #2: 化合物 | ChemComp-LTX / ( Leukotriene C4 Leukotriene C4 |
| 配列の詳細 | Residues 31-194, corresponding to TMD0, are modeled as poly-alanine. The register in this region ...Residues 31-194, corresponding to TMD0, are modeled as poly-alanine. The register in this region could not be confidently established and thus the numbering assigned to the residues is putative. The poly-alanine regions have been renamed as unknown |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: bovine multidrug resistance protein 1 (MRP1) complexed with leukotriene C4 タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.17 MDa / 実験値: NO | ||||||||||||||||||||||||||||||||||||
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) | ||||||||||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:   Homo sapiens (ヒト) / 細胞: HEK293S GntI- / プラスミド Homo sapiens (ヒト) / 細胞: HEK293S GntI- / プラスミド : Baculovirus : Baculovirus | ||||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 8 | ||||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 4.65 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: GOLD グリッドのタイプ: Quantifoil R1.2/1.3 400-mesh Au Holey Carbon Grids | ||||||||||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 295 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 37000 X / 最大 デフォーカス(公称値): 2400 nm / 最小 デフォーカス(公称値): 700 nm / Cs Bright-field microscopy / 倍率(公称値): 37000 X / 最大 デフォーカス(公称値): 2400 nm / 最小 デフォーカス(公称値): 700 nm / Cs : 2.7 mm / C2レンズ絞り径: 70 µm : 2.7 mm / C2レンズ絞り径: 70 µm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER 最高温度: 100 K / 最低温度: 80 K |
| 撮影 | 平均露光時間: 7 sec. / 電子線照射量: 84 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2302 |
| 画像スキャン | 横: 3710 / 縦: 3838 / 動画フレーム数/画像: 50 / 利用したフレーム数/画像: 1-50 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0158 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 203732 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.34 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 203732 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL / 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.34→146 Å / Cor.coef. Fo:Fc: 0.973 / ESU R: 0.423 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 285.432 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 9900 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj