+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5j0c | ||||||
|---|---|---|---|---|---|---|---|
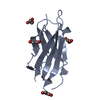
| タイトル | Monomeric Human Cu,Zn Superoxide dismutase, loops IV and VII deleted, apo form, circular permutant P2/3 | ||||||
 要素 要素 | Superoxide dismutase [Cu-Zn],Superoxide dismutase [Cu-Zn],OXIDOREDUCTASE,Superoxide dismutase [Cu-Zn] | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  SOD1 SOD1 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報action potential initiation / neurofilament cytoskeleton organization / anterograde axonal transport / retrograde axonal transport / positive regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / protein phosphatase 2B binding / regulation of organ growth / relaxation of vascular associated smooth muscle / response to superoxide /  peripheral nervous system myelin maintenance ...action potential initiation / neurofilament cytoskeleton organization / anterograde axonal transport / retrograde axonal transport / positive regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / protein phosphatase 2B binding / regulation of organ growth / relaxation of vascular associated smooth muscle / response to superoxide / peripheral nervous system myelin maintenance ...action potential initiation / neurofilament cytoskeleton organization / anterograde axonal transport / retrograde axonal transport / positive regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / protein phosphatase 2B binding / regulation of organ growth / relaxation of vascular associated smooth muscle / response to superoxide /  peripheral nervous system myelin maintenance / regulation of T cell differentiation in thymus / superoxide anion generation / retina homeostasis / negative regulation of cholesterol biosynthetic process / hydrogen peroxide biosynthetic process / auditory receptor cell stereocilium organization / peripheral nervous system myelin maintenance / regulation of T cell differentiation in thymus / superoxide anion generation / retina homeostasis / negative regulation of cholesterol biosynthetic process / hydrogen peroxide biosynthetic process / auditory receptor cell stereocilium organization /  regulation of protein kinase activity / myeloid cell homeostasis / muscle cell cellular homeostasis / regulation of protein kinase activity / myeloid cell homeostasis / muscle cell cellular homeostasis /  regulation of GTPase activity / superoxide metabolic process / regulation of GTPase activity / superoxide metabolic process /  heart contraction / positive regulation of catalytic activity / heart contraction / positive regulation of catalytic activity /  スーパーオキシドディスムターゼ / Detoxification of Reactive Oxygen Species / transmission of nerve impulse / negative regulation of reproductive process / negative regulation of developmental process / スーパーオキシドディスムターゼ / Detoxification of Reactive Oxygen Species / transmission of nerve impulse / negative regulation of reproductive process / negative regulation of developmental process /  superoxide dismutase activity / neuronal action potential / regulation of multicellular organism growth / response to axon injury / ectopic germ cell programmed cell death / glutathione metabolic process / positive regulation of phagocytosis / ovarian follicle development / axon cytoplasm / superoxide dismutase activity / neuronal action potential / regulation of multicellular organism growth / response to axon injury / ectopic germ cell programmed cell death / glutathione metabolic process / positive regulation of phagocytosis / ovarian follicle development / axon cytoplasm /  着床 / reactive oxygen species metabolic process / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / dendrite cytoplasm / removal of superoxide radicals / positive regulation of superoxide anion generation / regulation of mitochondrial membrane potential / thymus development / locomotory behavior / placenta development / response to organic substance / determination of adult lifespan / positive regulation of cytokine production / sensory perception of sound / response to hydrogen peroxide / 着床 / reactive oxygen species metabolic process / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / dendrite cytoplasm / removal of superoxide radicals / positive regulation of superoxide anion generation / regulation of mitochondrial membrane potential / thymus development / locomotory behavior / placenta development / response to organic substance / determination of adult lifespan / positive regulation of cytokine production / sensory perception of sound / response to hydrogen peroxide /  ミトコンドリア / ミトコンドリア /  small GTPase binding / small GTPase binding /  血圧 / negative regulation of inflammatory response / 血圧 / negative regulation of inflammatory response /  ペルオキシソーム / Platelet degranulation / ペルオキシソーム / Platelet degranulation /  遺伝子発現 / response to heat / protein-folding chaperone binding / cytoplasmic vesicle / 遺伝子発現 / response to heat / protein-folding chaperone binding / cytoplasmic vesicle /  精子形成 / response to ethanol / intracellular iron ion homeostasis / negative regulation of neuron apoptotic process / positive regulation of MAPK cascade / 精子形成 / response to ethanol / intracellular iron ion homeostasis / negative regulation of neuron apoptotic process / positive regulation of MAPK cascade /  ミトコンドリアマトリックス / response to xenobiotic stimulus / positive regulation of apoptotic process / copper ion binding / neuronal cell body / apoptotic process / protein-containing complex / ミトコンドリアマトリックス / response to xenobiotic stimulus / positive regulation of apoptotic process / copper ion binding / neuronal cell body / apoptotic process / protein-containing complex /  ミトコンドリア / ミトコンドリア /  extracellular space / extracellular exosome / zinc ion binding / extracellular region / extracellular space / extracellular exosome / zinc ion binding / extracellular region /  核質 / identical protein binding / 核質 / identical protein binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.6 Å 分子置換 / 解像度: 1.6 Å | ||||||
 データ登録者 データ登録者 | Wang, H. / Lang, L. / Logan, D. / Danielsson, J. / Oliveberg, M. | ||||||
 引用 引用 |  ジャーナル: J. Am. Chem. Soc. / 年: 2016 ジャーナル: J. Am. Chem. Soc. / 年: 2016タイトル: Tricking a Protein To Swap Strands. 著者: Wang, H. / Lang, L. / Logan, D.T. / Danielsson, J. / Oliveberg, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5j0c.cif.gz 5j0c.cif.gz | 99.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5j0c.ent.gz pdb5j0c.ent.gz | 76.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5j0c.json.gz 5j0c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/j0/5j0c https://data.pdbj.org/pub/pdb/validation_reports/j0/5j0c ftp://data.pdbj.org/pub/pdb/validation_reports/j0/5j0c ftp://data.pdbj.org/pub/pdb/validation_reports/j0/5j0c | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 11277.669 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SOD1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SOD1 / 発現宿主:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌)参照: UniProt: P00441,  スーパーオキシドディスムターゼ スーパーオキシドディスムターゼ#2: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.36 Å3/Da / 溶媒含有率: 47.93 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸発脱水法 / pH: 5 / 詳細: 0.1 M MIB buffer pH 5.0 25 % w/v PEG 1500 / PH範囲: 5 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  MAX II MAX II  / ビームライン: I911-3 / 波長: 1 Å / ビームライン: I911-3 / 波長: 1 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2014年9月11日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1 Å / 相対比: 1 : 1 Å / 相対比: 1 |
| 反射 | 解像度: 1.6→24 Å / Num. obs: 27738 / % possible obs: 100 % / 冗長度: 6.2 % / Rmerge(I) obs: 0.078 / Net I/σ(I): 12.9 |
| 反射 シェル | 解像度: 1.6→1.63 Å / Rmerge(I) obs: 0.63 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 4BCZ 解像度: 1.6→23.32 Å / Cor.coef. Fo:Fc: 0.971 / Cor.coef. Fo:Fc free: 0.958 / SU B: 2.913 / SU ML: 0.047 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.097 / ESU R Free: 0.079 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : REFINED INDIVIDUALLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 90.31 Å2 / Biso mean: 23.238 Å2 / Biso min: 8.87 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.6→23.32 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.6→1.642 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj







