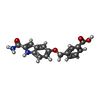
登録情報 データベース : PDB / ID : 5fbiタイトル COMPLEMENT FACTOR D IN COMPLEX WITH COMPOUND 3b Complement factor D キーワード 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 1.47 Å データ登録者 Ostermann, N. / Zink, F. ジャーナル : Nat.Chem.Biol. / 年 : 2016タイトル : Small-molecule factor D inhibitors targeting the alternative complement pathway.著者: Maibaum, J. / Liao, S.M. / Vulpetti, A. / Ostermann, N. / Randl, S. / Rudisser, S. / Lorthiois, E. / Erbel, P. / Kinzel, B. / Kolb, F.A. / Barbieri, S. / Wagner, J. / Durand, C. / Fettis, K. ... 著者 : Maibaum, J. / Liao, S.M. / Vulpetti, A. / Ostermann, N. / Randl, S. / Rudisser, S. / Lorthiois, E. / Erbel, P. / Kinzel, B. / Kolb, F.A. / Barbieri, S. / Wagner, J. / Durand, C. / Fettis, K. / Dussauge, S. / Hughes, N. / Delgado, O. / Hommel, U. / Gould, T. / Mac Sweeney, A. / Gerhartz, B. / Cumin, F. / Flohr, S. / Schubart, A. / Jaffee, B. / Harrison, R. / Risitano, A.M. / Eder, J. / Anderson, K. 履歴 登録 2015年12月14日 登録サイト / 処理サイト 改定 1.0 2016年10月26日 Provider / タイプ 改定 1.1 2016年11月2日 Group 改定 1.2 2016年11月23日 Group 改定 1.3 2024年1月10日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 Factor D
Factor D  キーワード
キーワード HYDROLASE (加水分解酵素)
HYDROLASE (加水分解酵素) 機能・相同性情報
機能・相同性情報 complement factor D / Alternative complement activation /
complement factor D / Alternative complement activation /  complement activation, alternative pathway /
complement activation, alternative pathway /  補体 / serine-type peptidase activity / platelet alpha granule lumen / response to bacterium / Platelet degranulation / secretory granule lumen / ficolin-1-rich granule lumen ...
補体 / serine-type peptidase activity / platelet alpha granule lumen / response to bacterium / Platelet degranulation / secretory granule lumen / ficolin-1-rich granule lumen ... complement factor D / Alternative complement activation /
complement factor D / Alternative complement activation /  complement activation, alternative pathway /
complement activation, alternative pathway /  補体 / serine-type peptidase activity / platelet alpha granule lumen / response to bacterium / Platelet degranulation / secretory granule lumen / ficolin-1-rich granule lumen / serine-type endopeptidase activity / Neutrophil degranulation /
補体 / serine-type peptidase activity / platelet alpha granule lumen / response to bacterium / Platelet degranulation / secretory granule lumen / ficolin-1-rich granule lumen / serine-type endopeptidase activity / Neutrophil degranulation /  タンパク質分解 / extracellular exosome / extracellular region
タンパク質分解 / extracellular exosome / extracellular region
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.47 Å
分子置換 / 解像度: 1.47 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Chem.Biol. / 年: 2016
ジャーナル: Nat.Chem.Biol. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5fbi.cif.gz
5fbi.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5fbi.ent.gz
pdb5fbi.ent.gz PDB形式
PDB形式 5fbi.json.gz
5fbi.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/fb/5fbi
https://data.pdbj.org/pub/pdb/validation_reports/fb/5fbi ftp://data.pdbj.org/pub/pdb/validation_reports/fb/5fbi
ftp://data.pdbj.org/pub/pdb/validation_reports/fb/5fbi リンク
リンク 集合体
集合体
 要素
要素 Factor D / Adipsin / C3 convertase activator / Properdin factor D
Factor D / Adipsin / C3 convertase activator / Properdin factor D
 Homo sapiens (ヒト) / 遺伝子: CFD, DF, PFD / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CFD, DF, PFD / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P00746,
Escherichia coli (大腸菌) / 参照: UniProt: P00746,  complement factor D
complement factor D グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X10SA / 波長: 0.9837 Å
/ ビームライン: X10SA / 波長: 0.9837 Å : 0.9837 Å / 相対比: 1
: 0.9837 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj