登録情報 データベース : PDB / ID : 5bx3タイトル Crystal structure of Thermoanaerobacterium xylanolyticum GH116 beta-glucosidase with deoxynojirimycin beta-glucosidase キーワード / / / / 機能・相同性 生物種 Thermoanaerobacterium xylanolyticum LX-11 (バクテリア)手法 / / / 解像度 : 1.96 Å データ登録者 Charoenwattanasatien, R. / Pengthaisong, S. / Sansenya, S. / Mutoh, R. / Tankrathok, A. / Tanaka, H. / Kurisu, G. / Ketudat Cairns, J.R. 資金援助 組織 認可番号 国 The Thailand Research Fund BRG5680012 Suranaree University of Technology, National Research University Project of the Commission on Higher Education of Thailand Institute for Protein Research, Osaka University
ジャーナル : Acs Chem.Biol. / 年 : 2016タイトル : Bacterial beta-Glucosidase Reveals the Structural and Functional Basis of Genetic Defects in Human Glucocerebrosidase 2 (GBA2)著者: Charoenwattanasatien, R. / Pengthaisong, S. / Breen, I. / Mutoh, R. / Sansenya, S. / Hua, Y. / Tankrathok, A. / Wu, L. / Songsiriritthigul, C. / Tanaka, H. / Williams, S.J. / Davies, G.J. / ... 著者 : Charoenwattanasatien, R. / Pengthaisong, S. / Breen, I. / Mutoh, R. / Sansenya, S. / Hua, Y. / Tankrathok, A. / Wu, L. / Songsiriritthigul, C. / Tanaka, H. / Williams, S.J. / Davies, G.J. / Kurisu, G. / Ketudat Cairns, J.R. 履歴 登録 2015年6月8日 登録サイト / 処理サイト 改定 1.0 2016年5月18日 Provider / タイプ 改定 1.1 2016年7月27日 Group 改定 1.2 2020年2月19日 Group / Derived calculations / カテゴリ / pdbx_struct_oper_listItem / _pdbx_struct_oper_list.symmetry_operation改定 1.3 2023年11月8日 Group / Database references / Refinement descriptionカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード HYDROLASE (加水分解酵素) / Thermoanaerobacterium xylolyticum / GH116 /
HYDROLASE (加水分解酵素) / Thermoanaerobacterium xylolyticum / GH116 /  beta-glucosidase /
beta-glucosidase /  deoxynojirimycin (1-デオキシノジリマイシン)
deoxynojirimycin (1-デオキシノジリマイシン) 機能・相同性情報
機能・相同性情報 グルコシルセラミダーゼ / glucosylceramide catabolic process /
グルコシルセラミダーゼ / glucosylceramide catabolic process /  glucosylceramidase activity / carbohydrate metabolic process /
glucosylceramidase activity / carbohydrate metabolic process /  生体膜 /
生体膜 /  metal ion binding
metal ion binding Thermoanaerobacterium xylanolyticum LX-11 (バクテリア)
Thermoanaerobacterium xylanolyticum LX-11 (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.96 Å
分子置換 / 解像度: 1.96 Å  データ登録者
データ登録者 タイ,
タイ,  日本, 3件
日本, 3件  引用
引用 ジャーナル: Acs Chem.Biol. / 年: 2016
ジャーナル: Acs Chem.Biol. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5bx3.cif.gz
5bx3.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5bx3.ent.gz
pdb5bx3.ent.gz PDB形式
PDB形式 5bx3.json.gz
5bx3.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/bx/5bx3
https://data.pdbj.org/pub/pdb/validation_reports/bx/5bx3 ftp://data.pdbj.org/pub/pdb/validation_reports/bx/5bx3
ftp://data.pdbj.org/pub/pdb/validation_reports/bx/5bx3 リンク
リンク 集合体
集合体
 要素
要素
 Thermoanaerobacterium xylanolyticum LX-11 (バクテリア)
Thermoanaerobacterium xylanolyticum LX-11 (バクテリア)
 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: F6BL85,
Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: F6BL85,  beta-glucosidase
beta-glucosidase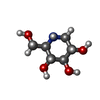








 1-デオキシノジリマイシン
1-デオキシノジリマイシン グリセリン
グリセリン エチレングリコール
エチレングリコール 水
水 X線回折
X線回折 試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SPring-8
SPring-8  / ビームライン: BL44XU / 波長: 0.9 Å
/ ビームライン: BL44XU / 波長: 0.9 Å : 0.9 Å / 相対比: 1
: 0.9 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj








