登録情報 データベース : PDB / ID : 4exoタイトル Revised, rerefined crystal structure of PDB entry 2QHK, methyl accepting chemotaxis protein Methyl-accepting chemotaxis protein キーワード / / / / / 機能・相同性 生物種 Vibrio parahaemolyticus (腸炎ビブリオ)手法 / / / 解像度 : 1.9 Å データ登録者 Sweeney, E.G. / Henderson, J.N. / Goers, J. / Wreden, C. / Hicks, K.G. / Foster, J.K. / Parthasarathy, R. / Remington, S.J. / Guillemin, K. ジャーナル : To be Published タイトル : The crystal structure of the methyl-accepting chemotaxis protein from Vibrio parahaemolyticus RIMD 2210633著者 : Zhang, R. / Li, H. / Clancy, S. / Joachimiak, A. 履歴 登録 2012年4月30日 登録サイト / 処理サイト 改定 1.0 2012年5月30日 Provider / タイプ 改定 1.1 2012年8月29日 Group 改定 1.2 2017年11月15日 Group / カテゴリ 改定 1.3 2023年9月13日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_special_symmetry / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id 改定 2.0 2023年11月15日 Group / Data collection / カテゴリ / chem_comp_atom / chem_comp_bondItem _atom_site.auth_atom_id / _atom_site.label_atom_id ... _atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp_atom.atom_id / _chem_comp_bond.atom_id_1 / _chem_comp_bond.atom_id_2
すべて表示 表示を減らす Remark 0 THIS ENTRY 4EXO REFLECTS AN ALTERNATIVE MODELING OF THE STRUCTURAL DATA IN R2QHKSF ORIGINAL DATA ... THIS ENTRY 4EXO REFLECTS AN ALTERNATIVE MODELING OF THE STRUCTURAL DATA IN R2QHKSF ORIGINAL DATA DETERMINED BY AUTHOR:ZHANG, R., LI, H., CLANCY, S., JOACHIMIAK, A., MIDWEST CENTER FOR STRUCTURAL GENOMICS (MCSG)
 データを開く
データを開く 基本情報
基本情報 要素
要素 Methyl-accepting chemotaxis proteins
Methyl-accepting chemotaxis proteins  キーワード
キーワード SIGNALING PROTEIN / chemotaxis receptor /
SIGNALING PROTEIN / chemotaxis receptor /  PAS domain / four helix bundle / methyl accepting chemotaxis receptor /
PAS domain / four helix bundle / methyl accepting chemotaxis receptor /  periplasmic domain (ペリプラズム)
periplasmic domain (ペリプラズム) 機能・相同性情報
機能・相同性情報
 Vibrio parahaemolyticus (腸炎ビブリオ)
Vibrio parahaemolyticus (腸炎ビブリオ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4exo.cif.gz
4exo.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4exo.ent.gz
pdb4exo.ent.gz PDB形式
PDB形式 4exo.json.gz
4exo.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ex/4exo
https://data.pdbj.org/pub/pdb/validation_reports/ex/4exo ftp://data.pdbj.org/pub/pdb/validation_reports/ex/4exo
ftp://data.pdbj.org/pub/pdb/validation_reports/ex/4exo リンク
リンク 集合体
集合体

 要素
要素 Methyl-accepting chemotaxis proteins
Methyl-accepting chemotaxis proteins
 Vibrio parahaemolyticus (腸炎ビブリオ)
Vibrio parahaemolyticus (腸炎ビブリオ)
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: Q87T87
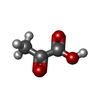
Escherichia coli (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: Q87T87 ピルビン酸
ピルビン酸 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 19-ID / 波長: 0.9794 Å
/ ビームライン: 19-ID / 波長: 0.9794 Å : 0.9794 Å / 相対比: 1
: 0.9794 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj