+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4bv2 | ||||||
|---|---|---|---|---|---|---|---|
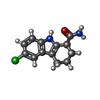
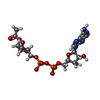
| タイトル | CRYSTAL STRUCTURE OF SIR2 IN COMPLEX WITH THE INHIBITOR EX-527, 2'-O-ACETYL-ADP-RIBOSE AND DEACETYLATED P53-PEPTIDE | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / NAD-DEPENDENT DEACETYLASE / HYDROLASE (加水分解酵素) / NAD-DEPENDENT DEACETYLASE /  SIRTUIN (サーチュイン遺伝子) / SIRTUIN (サーチュイン遺伝子) /  INHIBITOR COMPLEX (酵素阻害剤) / EX-527 / ACETYLATED ADP-RIBOSE / HYDROLASE-HYDROLASE INHIBITOR COMPLEX (酵素阻害剤) / EX-527 / ACETYLATED ADP-RIBOSE / HYDROLASE-HYDROLASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein acetyllysine N-acetyltransferase / NAD-dependent histone deacetylase activity / Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria ...protein acetyllysine N-acetyltransferase / NAD-dependent histone deacetylase activity / Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity / negative regulation of helicase activity / regulation of cell cycle G2/M phase transition / intrinsic apoptotic signaling pathway in response to hypoxia / regulation of fibroblast apoptotic process / oxidative stress-induced premature senescence / oligodendrocyte apoptotic process / negative regulation of miRNA processing / positive regulation of thymocyte apoptotic process / glucose catabolic process to lactate via pyruvate /  regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death / regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death /  転写 (生物学) / bone marrow development / circadian behavior / T cell proliferation involved in immune response / regulation of mitochondrial membrane permeability involved in apoptotic process / histone deacetylase regulator activity / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of neuroblast proliferation / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / Regulation of TP53 Activity through Association with Co-factors / positive regulation of execution phase of apoptosis / mitochondrial DNA repair / T cell lineage commitment / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / positive regulation of cardiac muscle cell apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / entrainment of circadian clock by photoperiod / cardiac septum morphogenesis / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding / 転写 (生物学) / bone marrow development / circadian behavior / T cell proliferation involved in immune response / regulation of mitochondrial membrane permeability involved in apoptotic process / histone deacetylase regulator activity / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of neuroblast proliferation / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / Regulation of TP53 Activity through Association with Co-factors / positive regulation of execution phase of apoptosis / mitochondrial DNA repair / T cell lineage commitment / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / positive regulation of cardiac muscle cell apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / TP53 Regulates Transcription of Caspase Activators and Caspases / entrainment of circadian clock by photoperiod / cardiac septum morphogenesis / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding /  マイトファジー / negative regulation of telomere maintenance via telomerase / SUMOylation of transcription factors / intrinsic apoptotic signaling pathway by p53 class mediator / neuroblast proliferation / general transcription initiation factor binding / cellular response to actinomycin D / Transcriptional Regulation by VENTX / DNA damage response, signal transduction by p53 class mediator / response to X-ray / マイトファジー / negative regulation of telomere maintenance via telomerase / SUMOylation of transcription factors / intrinsic apoptotic signaling pathway by p53 class mediator / neuroblast proliferation / general transcription initiation factor binding / cellular response to actinomycin D / Transcriptional Regulation by VENTX / DNA damage response, signal transduction by p53 class mediator / response to X-ray /  ヘイフリック限界 / chromosome organization / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / ヘイフリック限界 / chromosome organization / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress /  NAD+ binding / NAD+ binding /  gastrulation / cellular response to UV-C / response to inorganic substance / hematopoietic stem cell differentiation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of reactive oxygen species metabolic process / MDM2/MDM4 family protein binding / embryonic organ development / glial cell proliferation / gastrulation / cellular response to UV-C / response to inorganic substance / hematopoietic stem cell differentiation / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of reactive oxygen species metabolic process / MDM2/MDM4 family protein binding / embryonic organ development / glial cell proliferation /  Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest / Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest /  somitogenesis / type II interferon-mediated signaling pathway / DNA damage response, signal transduction by p53 class mediator resulting in cell cycle arrest / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cardiac muscle cell apoptotic process / response to salt stress somitogenesis / type II interferon-mediated signaling pathway / DNA damage response, signal transduction by p53 class mediator resulting in cell cycle arrest / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / negative regulation of fibroblast proliferation / cardiac muscle cell apoptotic process / response to salt stress類似検索 - 分子機能 | ||||||
| 生物種 |    THERMOTOGA MARITIMA (テルモトガ・マリティマ) THERMOTOGA MARITIMA (テルモトガ・マリティマ)  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.3 Å 分子置換 / 解像度: 3.3 Å | ||||||
 データ登録者 データ登録者 | Gertz, M. / Weyand, M. / Steegborn, C. | ||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013タイトル: Ex-527 Inhibits Sirtuins by Exploiting Their Unique Nad+-Dependent Deacetylation Mechanism 著者: Gertz, M. / Fischer, F. / Nguyen, G.T.T. / Lakshminarasimhan, M. / Schutkowski, M. / Weyand, M. / Steegborn, C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4bv2.cif.gz 4bv2.cif.gz | 211.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4bv2.ent.gz pdb4bv2.ent.gz | 173 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4bv2.json.gz 4bv2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bv/4bv2 https://data.pdbj.org/pub/pdb/validation_reports/bv/4bv2 ftp://data.pdbj.org/pub/pdb/validation_reports/bv/4bv2 ftp://data.pdbj.org/pub/pdb/validation_reports/bv/4bv2 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| |||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| |||||||||||||||||||||||||||||||||||||||||||
| 5 | 
| |||||||||||||||||||||||||||||||||||||||||||
| 6 | 
| |||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| |||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 27569.793 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)    THERMOTOGA MARITIMA (テルモトガ・マリティマ) THERMOTOGA MARITIMA (テルモトガ・マリティマ)発現宿主:   ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3)参照: UniProt: Q9WYW0,  加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用#2: タンパク質・ペプチド |  P53遺伝子 / DEACETYLATED P53-PEPTIDE / ANTIGEN NY-CO-13 / PHOSPHOPROTEIN P53 / TUMOR SUPPRESSOR P53 / P53 P53遺伝子 / DEACETYLATED P53-PEPTIDE / ANTIGEN NY-CO-13 / PHOSPHOPROTEIN P53 / TUMOR SUPPRESSOR P53 / P53分子量: 1597.900 Da / 分子数: 2 / Fragment: RESIDUES 380-384 / 由来タイプ: 合成 / 由来: (合成)   HOMO SAPIENS (ヒト) / 参照: UniProt: P04637 HOMO SAPIENS (ヒト) / 参照: UniProt: P04637#3: 化合物 | #4: 化合物 | #5: 化合物 | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.26 Å3/Da / 溶媒含有率: 45.61 % / 解説: NONE |
|---|---|
結晶化 | pH: 5.9 / 詳細: 20% PEG 6000, 0.1 M BIS-TRIS PH5.9 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  BESSY BESSY  / ビームライン: 14.1 / 波長: 0.918 / ビームライン: 14.1 / 波長: 0.918 |
| 検出器 | タイプ: MARMOSAIC 225 mm CCD / 検出器: CCD / 日付: 2012年11月14日 / 詳細: COLLIMATOR |
| 放射 | モノクロメーター: SI(111) MONOCHROMATOR / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.918 Å / 相対比: 1 : 0.918 Å / 相対比: 1 |
| 反射 | 解像度: 3.3→60.47 Å / Num. obs: 31261 / % possible obs: 99.8 % / Observed criterion σ(I): -3 / 冗長度: 4.2 % / Rmerge(I) obs: 0.11 / Net I/σ(I): 11.54 |
| 反射 シェル | 解像度: 3.3→3.4 Å / 冗長度: 4.4 % / Rmerge(I) obs: 1.05 / Mean I/σ(I) obs: 1.5 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1YC5 解像度: 3.3→60.47 Å / Cor.coef. Fo:Fc: 0.915 / Cor.coef. Fo:Fc free: 0.845 / SU B: 108.33 / SU ML: 0.846 / 交差検証法: THROUGHOUT / ESU R Free: 0.787 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT. U VALUES WITH TLS ADDED
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 114.439 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.3→60.47 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj