+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3zsz | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Small molecule inhibitors of the LEDGF site of HIV type 1 integrase identified by fragment screening and structure based drug design | ||||||
 要素 要素 | INTEGRASE インテグラーゼ インテグラーゼ | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  AIDS (後天性免疫不全症候群) AIDS (後天性免疫不全症候群) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 HIV-1 retropepsin / HIV-1 retropepsin /  : / : /  retroviral ribonuclease H / retroviral ribonuclease H /  exoribonuclease H / exoribonuclease H /  : / : /  exoribonuclease H activity / host multivesicular body / DNA integration / exoribonuclease H activity / host multivesicular body / DNA integration /  逆転写酵素 / viral genome integration into host DNA ... 逆転写酵素 / viral genome integration into host DNA ... HIV-1 retropepsin / HIV-1 retropepsin /  : / : /  retroviral ribonuclease H / retroviral ribonuclease H /  exoribonuclease H / exoribonuclease H /  : / : /  exoribonuclease H activity / host multivesicular body / DNA integration / exoribonuclease H activity / host multivesicular body / DNA integration /  逆転写酵素 / viral genome integration into host DNA / viral penetration into host nucleus / establishment of integrated proviral latency / 逆転写酵素 / viral genome integration into host DNA / viral penetration into host nucleus / establishment of integrated proviral latency /  RNA-directed DNA polymerase activity / RNA-directed DNA polymerase activity /  転移酵素; リンを含む基を移すもの; 核酸を移すもの / RNA-DNA hybrid ribonuclease activity / viral nucleocapsid / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / RNA-DNA hybrid ribonuclease activity / viral nucleocapsid /  endonuclease activity / DNA recombination / endonuclease activity / DNA recombination /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  DNAポリメラーゼ / aspartic-type endopeptidase activity / DNAポリメラーゼ / aspartic-type endopeptidase activity /  DNA-directed DNA polymerase activity / symbiont entry into host cell / symbiont-mediated suppression of host gene expression / DNA-directed DNA polymerase activity / symbiont entry into host cell / symbiont-mediated suppression of host gene expression /  lipid binding / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / lipid binding / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity /  タンパク質分解 / タンパク質分解 /  DNA binding / DNA binding /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  生体膜 生体膜類似検索 - 分子機能 | ||||||
| 生物種 |    HUMAN IMMUNODEFICIENCY VIRUS (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS (ヒト免疫不全ウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Peat, T.S. / Newman, J. / Rhodes, D.I. / Vandergraaff, N. / Le, G. / Jones, E.D. / Smith, J.A. / Coates, J.A.V. / Thienthong, N. / Dolezal, O. ...Peat, T.S. / Newman, J. / Rhodes, D.I. / Vandergraaff, N. / Le, G. / Jones, E.D. / Smith, J.A. / Coates, J.A.V. / Thienthong, N. / Dolezal, O. / Ryan, J.H. / Savage, G.P. / Francis, C.L. / Deadman, J.J. | ||||||
 引用 引用 |  ジャーナル: Plos One / 年: 2012 ジャーナル: Plos One / 年: 2012タイトル: Small Molecule Inhibitors of the Ledgf Site of Human Immunodeficiency Virus Integrase Identified by Fragment Screening and Structure Based Design. 著者: Peat, T.S. / Rhodes, D.I. / Vandegraaff, N. / Le, G. / Smith, J.A. / Clark, L.J. / Jones, E.D. / Coates, J.A.V. / Thienthong, N. / Newman, J. / Dolezal, O. / Mulder, R. / Ryan, J.H. / Savage, ...著者: Peat, T.S. / Rhodes, D.I. / Vandegraaff, N. / Le, G. / Smith, J.A. / Clark, L.J. / Jones, E.D. / Coates, J.A.V. / Thienthong, N. / Newman, J. / Dolezal, O. / Mulder, R. / Ryan, J.H. / Savage, G.P. / Francis, C.L. / Deadman, J.J. #1:  ジャーナル: Antivir.Chem.Chemother. / 年: 2011 ジャーナル: Antivir.Chem.Chemother. / 年: 2011タイトル: Structural Basis for a New Mechanism of Inhibition of HIV-1 Integrase Identified by Fragment Screening and Structure-Based Design. 著者: Rhodes, D.I. / Peat, T.S. / Vandegraaff, N. / Jeevarajah, D. / Le, G. / Jones, E.D. / Smith, J.A. / Coates, J.A. / Winfield, L.J. / Thienthong, N. / Newman, J. / Lucent, D. / Ryan, J.H. / ...著者: Rhodes, D.I. / Peat, T.S. / Vandegraaff, N. / Jeevarajah, D. / Le, G. / Jones, E.D. / Smith, J.A. / Coates, J.A. / Winfield, L.J. / Thienthong, N. / Newman, J. / Lucent, D. / Ryan, J.H. / Savage, G.P. / Francis, C.L. / Deadman, J.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3zsz.cif.gz 3zsz.cif.gz | 79.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3zsz.ent.gz pdb3zsz.ent.gz | 59.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3zsz.json.gz 3zsz.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/zs/3zsz https://data.pdbj.org/pub/pdb/validation_reports/zs/3zsz ftp://data.pdbj.org/pub/pdb/validation_reports/zs/3zsz ftp://data.pdbj.org/pub/pdb/validation_reports/zs/3zsz | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  3zcmC  3zsoC  3zsqC  3zsrC  3zsvC  3zswC  3zsxC  3zsyC  3zt0C  3zt1C  3zt2C  3zt3C  3zt4C  3nf7S C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 |  インテグラーゼ インテグラーゼ分子量: 18395.842 Da / 分子数: 2 / 断片: CORE CATALYTIC DOMAIN, RESIDUES 56-212 / 変異: YES / 由来タイプ: 組換発現 / 詳細: INHIBITOR BOUND TO LEDGF BINDING SITE 由来: (組換発現)    HUMAN IMMUNODEFICIENCY VIRUS (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS (ヒト免疫不全ウイルス)株: TYPE 1 / 発現宿主:   ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21 ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21参照: UniProt: Q76353, UniProt: P12497*PLUS,  転移酵素; リンを含む基を移すもの; 核酸を移すもの 転移酵素; リンを含む基を移すもの; 核酸を移すもの |
|---|
-非ポリマー , 5種, 110分子 

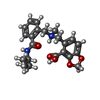






| #2: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩#3: 化合物 | ChemComp-ACT /  酢酸塩 酢酸塩#4: 化合物 | #5: 化合物 | ChemComp-CL / |  塩化物 塩化物#6: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 構成要素の詳細 | ENGINEERED RESIDUE IN CHAIN A, CYS 56 TO SER ENGINEERED RESIDUE IN CHAIN A, PHE 139 TO ASP ...ENGINEERED |
|---|---|
| 非ポリマーの詳細 | OM2 (R)-[2-[[(2S)-BUTAN-2-YL]CARBAMOYL]PHENYL] METHYL-[(4-CARBOXY-1,3-BENZODIOXOL-5-YL)METHYL]- ...OM2 (R)-[2-[[(2S)-BUTAN-2-YL]CARBAMOYL]PHENYL] METHYL-[(4-CARBOXY-1,3-BENZODIOXO |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.45 Å3/Da / 溶媒含有率: 50 % / 解説: NONE |
|---|---|
結晶化 | pH: 5.5 詳細: THE PROTEIN WAS CONCENTRATED TO 5.5 MG/ML IN 40 MM TRIS PH 8.0, 250 MM NACL, 30 MM MGCL2, 5 MM DTT AND SET UP IN A 1:1 RATIO WITH 1.6 TO 2.0 M AMMONIUM SULFATE, 100 MM SODIUM ACETATE BUFFER PH 5.0 TO 5.5. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Australian Synchrotron Australian Synchrotron  / ビームライン: MX1 / 波長: 0.96 / ビームライン: MX1 / 波長: 0.96 |
| 検出器 | タイプ: ADSC QUANTUM 210r / 検出器: CCD / 日付: 2009年7月29日 / 詳細: MIRRORS |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.96 Å / 相対比: 1 : 0.96 Å / 相対比: 1 |
| 反射 | 解像度: 2→45.3 Å / Num. obs: 25674 / % possible obs: 100 % / Observed criterion σ(I): 1 / 冗長度: 5.6 % / Biso Wilson estimate: 27.12 Å2 / Rmerge(I) obs: 0.08 / Net I/σ(I): 16.4 |
| 反射 シェル | 解像度: 2→2.11 Å / 冗長度: 5.6 % / Rmerge(I) obs: 0.55 / Mean I/σ(I) obs: 3 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 3NF7 解像度: 2→31.48 Å / Cor.coef. Fo:Fc: 0.95 / Cor.coef. Fo:Fc free: 0.936 / SU B: 3.773 / SU ML: 0.107 / 交差検証法: THROUGHOUT / ESU R: 0.165 / ESU R Free: 0.148 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 26.833 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→31.48 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj



