+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2vt4 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | TURKEY BETA1 ADRENERGIC RECEPTOR WITH STABILISING MUTATIONS AND BOUND CYANOPINDOLOL | ||||||
 要素 要素 | BETA1 ADRENERGIC RECEPTOR | ||||||
 キーワード キーワード |  RECEPTOR (受容体) / RECEPTOR (受容体) /  GPCR (Gタンパク質共役受容体) / GPCR (Gタンパク質共役受容体) /  MEMBRANE (生体膜) / MEMBRANE (生体膜) /  PALMITATE (パルミチン酸) / PALMITATE (パルミチン酸) /  TRANSDUCER (トランスデューサー) / ANTAGONIST BOUND FORM / INTEGRAL MEMBRANE PROTEIN / TRANSDUCER (トランスデューサー) / ANTAGONIST BOUND FORM / INTEGRAL MEMBRANE PROTEIN /  G-PROTEIN COUPLED RECEPTOR (Gタンパク質共役受容体) / G-PROTEIN COUPLED RECEPTOR (Gタンパク質共役受容体) /  G PROTEIN COUPLED RECEPTOR (Gタンパク質共役受容体) / THERMOSTABILISING POINT MUTATIONS / G PROTEIN COUPLED RECEPTOR (Gタンパク質共役受容体) / THERMOSTABILISING POINT MUTATIONS /  PHOSPHOPROTEIN / SEVEN-HELIX RECEPTOR / PHOSPHOPROTEIN / SEVEN-HELIX RECEPTOR /  LIPOPROTEIN (リポタンパク質) / LIPOPROTEIN (リポタンパク質) /  7TM RECEPTOR (Gタンパク質共役受容体) / 7TM RECEPTOR (Gタンパク質共役受容体) /  GLYCOPROTEIN (糖タンパク質) / GLYCOPROTEIN (糖タンパク質) /  TRANSMEMBRANE (膜貫通型タンパク質) TRANSMEMBRANE (膜貫通型タンパク質) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報beta1-adrenergic receptor activity / positive regulation of heart contraction / regulation of circadian sleep/wake cycle, sleep / adenylate cyclase-activating adrenergic receptor signaling pathway / positive regulation of GTPase activity /  エンドソーム / エンドソーム /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   MELEAGRIS GALLOPAVO (シチメンチョウ) MELEAGRIS GALLOPAVO (シチメンチョウ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | Warne, A. / Serrano-Vega, M.J. / Baker, J.G. / Moukhametzianov, R. / Edwards, P.C. / Henderson, R. / Leslie, A.G.W. / Tate, C.G. / Schertler, G.F.X. | ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2008 ジャーナル: Nature / 年: 2008タイトル: Structure of a Beta1-Adrenergic G-Protein-Coupled Receptor. 著者: Warne, A. / Serrano-Vega, M.J. / Baker, J.G. / Moukhametzianov, R. / Edwards, P.C. / Henderson, R. / Leslie, A.G.W. / Tate, C.G. / Schertler, G.F.X. #1: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2008 タイトル: Conformational Thermostabilisation of the Beta1-Adrenergic Receptor in a Detergent Resistant Form 著者: Serrano-Vega, M.J. / Magnani, F. / Shibata, Y. / Tate, C.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2vt4.cif.gz 2vt4.cif.gz | 233.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2vt4.ent.gz pdb2vt4.ent.gz | 188 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2vt4.json.gz 2vt4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/vt/2vt4 https://data.pdbj.org/pub/pdb/validation_reports/vt/2vt4 ftp://data.pdbj.org/pub/pdb/validation_reports/vt/2vt4 ftp://data.pdbj.org/pub/pdb/validation_reports/vt/2vt4 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  2rh1S S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 / 糖 , 2種, 16分子 ABCD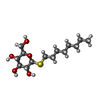

| #1: タンパク質 | 分子量: 35752.598 Da / 分子数: 4 / 断片: RESIDUES 33-243,272-276,279-367 / 変異: YES / 由来タイプ: 組換発現 由来: (組換発現)   MELEAGRIS GALLOPAVO (シチメンチョウ) MELEAGRIS GALLOPAVO (シチメンチョウ)Cell: ERYTHROCYTE / プラスミド: PBACPAK8 / 細胞株 (発現宿主): High Five / 発現宿主:   TRICHOPLUSIA NI (イラクサキンウワバ) / 参照: UniProt: P07700 TRICHOPLUSIA NI (イラクサキンウワバ) / 参照: UniProt: P07700#4: 糖 | ChemComp-SOG /  N-Octyl beta-D-thioglucopyranoside N-Octyl beta-D-thioglucopyranoside |
|---|
-非ポリマー , 4種, 41分子 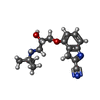






| #2: 化合物 | ChemComp-P32 / #3: 化合物 | ChemComp-NA / #5: 化合物 |  デカン デカン#6: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 構成要素の詳細 | ENGINEERED RESIDUE IN CHAIN A, ARG 68 TO SER ENGINEERED RESIDUE IN CHAIN A, MET 90 TO VAL ...ENGINEERED |
|---|---|
| 配列の詳細 | RESIDUES 3-32 AT THE N-TERMINUS AND RESIDUES 244-271 AND 277-278 OF THE THIRD INTRCELLULAR LOOP ...RESIDUES 3-32 AT THE N-TERMINUS AND RESIDUES 244-271 AND 277-278 OF THE THIRD INTRCELLUL |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.94 Å3/Da / 溶媒含有率: 58 % 解説: THE LYSOZYME COMPONENT OF 2RH1 WAS REMOVED FOR THE MOLECULAR REPLACEMENT |
|---|---|
結晶化 | 手法: 蒸気拡散法 / pH: 7.1 詳細: VAPOUR DIFFUSION. EQUAL VOLUMES OF PROTEIN (6MG/ML) IN 10MM TRIS-HCL PH7.7, 50MM NACL, 0.1MM EDTA, 0.35% OCTYLTHIOGLUCOSIDE, 0.5MM CYANOPINDOLOL AND RESERVOIR 0.1M ADA, PH 6.9-7.3, 29-32% PEG600. |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID23-2 / 波長: 0.8726 / ビームライン: ID23-2 / 波長: 0.8726 |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2007年12月14日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.8726 Å / 相対比: 1 : 0.8726 Å / 相対比: 1 |
| 反射 | 解像度: 2.7→46.4 Å / Num. obs: 41499 / % possible obs: 96.2 % / Observed criterion σ(I): 0 / 冗長度: 1.8 % / Biso Wilson estimate: 39.32 Å2 / Rmerge(I) obs: 0.14 / Net I/σ(I): 5.8 |
| 反射 シェル | 解像度: 2.7→2.85 Å / 冗長度: 1.8 % / Rmerge(I) obs: 0.67 / Mean I/σ(I) obs: 1.5 / % possible all: 95.7 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 2RH1 解像度: 2.7→45.1 Å / SU ML: 0.44 / 位相誤差: 25.37 / 立体化学のターゲット値: ML 詳細: STRICT NCS, SIGMA 0.025, APPLIED TO CHAINS A AND D AND TO CHAINS B AND C, EXCLUDING DETERGENTS
| ||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 52.82 Å2 / ksol: 0.343 e/Å3 | ||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 46.32 Å2
| ||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.7→45.1 Å
| ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj











