| 登録構造単位 | A: PROTEIN (RIBONUCLEASE T1)
B: PROTEIN (RIBONUCLEASE T1)
C: PROTEIN (RIBONUCLEASE T1)
D: PROTEIN (RIBONUCLEASE T1)
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 46,221 | 13 |
|---|
| ポリマ- | 44,207 | 4 |
|---|
| 非ポリマー | 2,014 | 9 |
|---|
| 水 | 3,801 | 211 |
|---|
|
|---|
| 1 | A: PROTEIN (RIBONUCLEASE T1)
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 11,415 | 2 |
|---|
| ポリマ- | 11,052 | 1 |
|---|
| 非ポリマー | 363 | 1 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
|
|---|
| 2 | B: PROTEIN (RIBONUCLEASE T1)
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 11,533 | 4 |
|---|
| ポリマ- | 11,052 | 1 |
|---|
| 非ポリマー | 481 | 3 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
|
|---|
| 3 | C: PROTEIN (RIBONUCLEASE T1)
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 11,818 | 4 |
|---|
| ポリマ- | 11,052 | 1 |
|---|
| 非ポリマー | 767 | 3 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
|
|---|
| 4 | D: PROTEIN (RIBONUCLEASE T1)
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 11,455 | 3 |
|---|
| ポリマ- | 11,052 | 1 |
|---|
| 非ポリマー | 403 | 2 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
|
|---|
| 単位格子 | | Length a, b, c (Å) | 59.370, 60.560, 101.170 |
|---|
| Angle α, β, γ (deg.) | 90.00, 90.00, 90.00 |
|---|
| Int Tables number | 19 |
|---|
| Space group name H-M | P212121 |
|---|
|
|---|
| 非結晶学的対称性 (NCS) | NCS oper: | ID | Code | Matrix | ベクター |
|---|
| 1 | given(-0.122589, -0.814916, -0.566466), (0.861869, -0.370421, 0.346368), (0.492091, -0.445758, 0.74776)54.31885, 63.69774, -10.60282| 2 | given(0.563091, 0.815358, 0.134611), (-0.430631, 0.428531, -0.794303), (-0.705326, 0.389297, 0.592421)55.91476, -57.56683, 57.5301| 3 | given(-0.013481, 0.860923, 0.508558), (-0.891036, 0.220448, -0.396809), (-0.453733, -0.458492, 0.764141)| 82.45089, 0.83091, 35.26741 | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HYDROLASE (加水分解酵素) /
HYDROLASE (加水分解酵素) /  ENDORIBONUCLEASE /
ENDORIBONUCLEASE /  RIBONUCLEASE (リボヌクレアーゼ) /
RIBONUCLEASE (リボヌクレアーゼ) /  ENDONUCLEASE (エンドヌクレアーゼ)
ENDONUCLEASE (エンドヌクレアーゼ) 機能・相同性情報
機能・相同性情報 ribonuclease T1 activity /
ribonuclease T1 activity /  ribonuclease T1 / cell septum /
ribonuclease T1 / cell septum /  endonuclease activity /
endonuclease activity /  lyase activity /
lyase activity /  RNA binding
RNA binding
 Aspergillus oryzae (米麹菌)
Aspergillus oryzae (米麹菌) X線回折 /
X線回折 /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Protein Sci. / 年: 1999
ジャーナル: Protein Sci. / 年: 1999 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2hoh.cif.gz
2hoh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2hoh.ent.gz
pdb2hoh.ent.gz PDB形式
PDB形式 2hoh.json.gz
2hoh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ho/2hoh
https://data.pdbj.org/pub/pdb/validation_reports/ho/2hoh ftp://data.pdbj.org/pub/pdb/validation_reports/ho/2hoh
ftp://data.pdbj.org/pub/pdb/validation_reports/ho/2hoh リンク
リンク 集合体
集合体
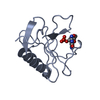
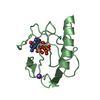


 要素
要素
 Aspergillus oryzae (米麹菌) / 発現宿主:
Aspergillus oryzae (米麹菌) / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): WK6 / 参照: UniProt: P00651, EC: 3.1.27.3
Escherichia coli (大腸菌) / 株 (発現宿主): WK6 / 参照: UniProt: P00651, EC: 3.1.27.3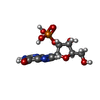








 グアニル酸
グアニル酸 リン酸塩
リン酸塩 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418
回転陽極 / タイプ: RIGAKU RU200 / 波長: 1.5418  : 1.5418 Å / 相対比: 1
: 1.5418 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



