+ データを開く
データを開く
- 基本情報
基本情報
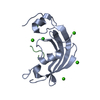
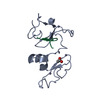
| 登録情報 | データベース: PDB / ID: 2a8v | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | RHO TRANSCRIPTION TERMINATION FACTOR/RNA COMPLEX | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PROTEIN/RNA / RHO / OB FOLD / SINGLE-STRANDED NUCLEIC ACID BINDING DOMAIN / PROTEIN-RNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATP-dependent activity, acting on RNA /  helicase activity / DNA-templated transcription termination / helicase activity / DNA-templated transcription termination /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 /  hydrolase activity / hydrolase activity /  ATP hydrolysis activity / ATP hydrolysis activity /  RNA binding / RNA binding /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.4 Å 分子置換 / 解像度: 2.4 Å | ||||||
 データ登録者 データ登録者 | Bogden, C.E. / Fass, D. / Bergman, N. / Nichols, M.D. / Berger, J.M. | ||||||
 引用 引用 |  ジャーナル: Mol.Cell / 年: 1999 ジャーナル: Mol.Cell / 年: 1999タイトル: The structural basis for terminator recognition by the Rho transcription termination factor. 著者: Bogden, C.E. / Fass, D. / Bergman, N. / Nichols, M.D. / Berger, J.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2a8v.cif.gz 2a8v.cif.gz | 85.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2a8v.ent.gz pdb2a8v.ent.gz | 65.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2a8v.json.gz 2a8v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/a8/2a8v https://data.pdbj.org/pub/pdb/validation_reports/a8/2a8v ftp://data.pdbj.org/pub/pdb/validation_reports/a8/2a8v ftp://data.pdbj.org/pub/pdb/validation_reports/a8/2a8v | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| 単位格子 |
| ||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
| #1: RNA鎖 | 分子量: 870.588 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli (大腸菌) / 株: DH5A / プラスミド: PET24B / 生物種 (発現宿主): Escherichia coli / 発現宿主: Escherichia coli (大腸菌) / 株: DH5A / プラスミド: PET24B / 生物種 (発現宿主): Escherichia coli / 発現宿主:   Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 | ||
|---|---|---|---|
| #2: RNA鎖 | 分子量: 1786.133 Da / 分子数: 1 / 由来タイプ: 合成 | ||
| #3: タンパク質 | 分子量: 13218.041 Da / 分子数: 3 / Fragment: N-TERMINAL RNA BINDING DOMAIN / 由来タイプ: 組換発現 / 参照: UniProt: P03002, UniProt: P0AG30*PLUS #4: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.53 % | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | pH: 4.5 / 詳細: pH 4.5 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 30 ℃ / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 110 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS NSLS  / ビームライン: X4A / 波長: 0.9779 / ビームライン: X4A / 波長: 0.9779 |
| 検出器 | タイプ: RIGAKU RAXIS IV / 検出器: IMAGE PLATE / 日付: 1998年4月15日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9779 Å / 相対比: 1 : 0.9779 Å / 相対比: 1 |
| 反射 | 解像度: 2.4→14 Å / Num. obs: 14393 / % possible obs: 99.7 % / Observed criterion σ(I): 2 / 冗長度: 3.7 % / Biso Wilson estimate: 41 Å2 / Rsym value: 0.074 / Net I/σ(I): 16 |
| 反射 シェル | 解像度: 2.4→2.49 Å / 冗長度: 3 % / Mean I/σ(I) obs: 3.6 / Rsym value: 0.283 / % possible all: 99.6 |
| 反射 | *PLUS Rmerge(I) obs: 0.074 |
| 反射 シェル | *PLUS % possible obs: 99.6 % / Rmerge(I) obs: 0.283 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1A8V 解像度: 2.4→14 Å / 交差検証法: THROUGHOUT / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.4→14 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: RESTRAINTS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj



