+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ne7 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HUMAN GLUCOSAMINE-6-PHOSPHATE DEAMINASE ISOMERASE AT 1.75 A RESOLUTION COMPLEXED WITH N-ACETYL-GLUCOSAMINE-6-PHOSPHATE AND 2-DEOXY-2-AMINO-GLUCITOL-6-PHOSPHATE | ||||||||||||
 要素 要素 | Glucosamine-6-phosphate isomerase | ||||||||||||
 キーワード キーワード |  HYDROLASE (加水分解酵素) / V-TYPE LIKE ALLOSTERIC ENZYME / CONFORMATIONAL DISORDER / CONFORMATIONAL DIFFERENCES HYDROLASE (加水分解酵素) / V-TYPE LIKE ALLOSTERIC ENZYME / CONFORMATIONAL DISORDER / CONFORMATIONAL DIFFERENCES | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glucosamine catabolic process /  glucosamine-6-phosphate deaminase / glucosamine-6-phosphate deaminase /  glucosamine-6-phosphate deaminase activity / N-acetylglucosamine catabolic process / N-acetylneuraminate catabolic process / UDP-N-acetylglucosamine biosynthetic process / glucosamine-6-phosphate deaminase activity / N-acetylglucosamine catabolic process / N-acetylneuraminate catabolic process / UDP-N-acetylglucosamine biosynthetic process /  解糖系 / single fertilization / generation of precursor metabolites and energy / carbohydrate metabolic process ...glucosamine catabolic process / 解糖系 / single fertilization / generation of precursor metabolites and energy / carbohydrate metabolic process ...glucosamine catabolic process /  glucosamine-6-phosphate deaminase / glucosamine-6-phosphate deaminase /  glucosamine-6-phosphate deaminase activity / N-acetylglucosamine catabolic process / N-acetylneuraminate catabolic process / UDP-N-acetylglucosamine biosynthetic process / glucosamine-6-phosphate deaminase activity / N-acetylglucosamine catabolic process / N-acetylneuraminate catabolic process / UDP-N-acetylglucosamine biosynthetic process /  解糖系 / single fertilization / generation of precursor metabolites and energy / carbohydrate metabolic process / extracellular exosome / identical protein binding / 解糖系 / single fertilization / generation of precursor metabolites and energy / carbohydrate metabolic process / extracellular exosome / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.75 Å 分子置換 / 解像度: 1.75 Å | ||||||||||||
 データ登録者 データ登録者 | Arreola, R. / Valderrama, B. / Morante, M.L. / Horjales, E. | ||||||||||||
 引用 引用 |  ジャーナル: Febs Lett. / 年: 2003 ジャーナル: Febs Lett. / 年: 2003タイトル: Two mammalian glucosamine-6-phosphate deaminases: a structural and genetic study. 著者: Arreola, R. / Valderrama, B. / Morante, M.L. / Horjales, E. #1:  ジャーナル: BIOCHIM.BIOPHYS.ACTA / 年: 1998 ジャーナル: BIOCHIM.BIOPHYS.ACTA / 年: 1998タイトル: Glucosamine-6-phosphate deaminase from beef kidney is an allosteric system of the V-type. 著者: Lara-Lemus, R. / Calcagno, M.L. #2:  ジャーナル: Structure / 年: 1995 ジャーナル: Structure / 年: 1995タイトル: Structure and catalytic mechanism of glucosamine 6-phosphate deaminase from Escherichia coli at 2.1 A resolution. 著者: Oliva, G. / Fontes, M.R. / Garratt, R.C. / Altamirano, M.M. / Calcagno, M.L. / Horjales, E. | ||||||||||||
| 履歴 |
| ||||||||||||
| Remark 600 | HETEROGEN THE LIGANDS SO4 AND AGP ARE IN ALTERNATE POSITIONS. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ne7.cif.gz 1ne7.cif.gz | 396.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ne7.ent.gz pdb1ne7.ent.gz | 319.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ne7.json.gz 1ne7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ne/1ne7 https://data.pdbj.org/pub/pdb/validation_reports/ne/1ne7 ftp://data.pdbj.org/pub/pdb/validation_reports/ne/1ne7 ftp://data.pdbj.org/pub/pdb/validation_reports/ne/1ne7 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1deaS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | The biological assembly is the hexamer in the asymmetric unit reported in this entry. Chains: A, B, C, D, E & F. |
- 要素
要素
-タンパク質 , 1種, 6分子 ABCDEF
| #1: タンパク質 | 分子量: 32712.506 Da / 分子数: 6 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: GNPI OR HLN OR KIAA0060 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GNPI OR HLN OR KIAA0060 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P46926, Escherichia coli (大腸菌) / 参照: UniProt: P46926,  glucosamine-6-phosphate deaminase glucosamine-6-phosphate deaminase |
|---|
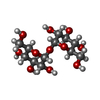
-糖 , 2種, 12分子 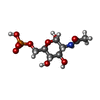
| #2: 多糖 | alpha-D-glucopyranose-(1-1)-alpha-D-glucopyranose /  トレハロース トレハロース#3: 糖 | ChemComp-16G / |
|---|
-非ポリマー , 3種, 2207分子 
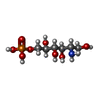



| #4: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩#5: 化合物 | ChemComp-AGP / #6: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.81 Å3/Da / 溶媒含有率: 56.17 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 291 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: 1.68 M ammonium sulphate, 10 mM N-acetyl-glucosamine-6-phosphate and 0.1 mM 2-deoxy-2-amino D-glucitol 6-phosphate, pH 7.0, VAPOR DIFFUSION, SITTING DROP, temperature 291K | ||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 18 ℃ / pH: 7 / 手法: 蒸気拡散法, シッティングドロップ法 | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 93 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL9-1 / 波長: 0.78 Å / ビームライン: BL9-1 / 波長: 0.78 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1999年3月18日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.78 Å / 相対比: 1 : 0.78 Å / 相対比: 1 |
| 反射 | 解像度: 1.75→19.97 Å / Num. all: 221759 / Num. obs: 209143 / % possible obs: 94.3 % / Observed criterion σ(F): 1 / Observed criterion σ(I): 0 / 冗長度: 3.8 % / Biso Wilson estimate: 19.5 Å2 / Rsym value: 0.052 / Net I/σ(I): 9.9 |
| 反射 シェル | 解像度: 1.75→1.86 Å / 冗長度: 3.6 % / Mean I/σ(I) obs: 3.4 / Num. unique all: 32182 / Rsym value: 0.224 / % possible all: 95.8 |
| 反射 | *PLUS Rmerge(I) obs: 0.052 |
| 反射 シェル | *PLUS % possible obs: 96.8 % / Rmerge(I) obs: 0.224 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1DEA 解像度: 1.75→19.97 Å / Rfactor Rfree error: 0.002 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 67.2697 Å2 / ksol: 0.376291 e/Å3 | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 25.9 Å2
| ||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.75→19.97 Å
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.75→1.76 Å / Rfactor Rfree error: 0.004 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS % reflection Rfree: 10 % | ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
| ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | *PLUS 最低解像度: 1.86 Å / Rfactor Rfree: 0.245 / Rfactor Rwork: 0.223 |
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj