+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ha3 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | ELONGATION FACTOR TU IN COMPLEX WITH aurodox | ||||||
 要素 要素 | ELONGATION FACTOR TU EF-Tu EF-Tu | ||||||
 キーワード キーワード |  TRANSLATION (翻訳 (生物学)) / TRANSLATION (翻訳 (生物学)) /  GTPASE (GTPアーゼ) / AURODOX / N-METHYL-KIRROMYCIN / GTPASE (GTPアーゼ) / AURODOX / N-METHYL-KIRROMYCIN /  ANTIBIOTIC (抗生物質) / ANTIBIOTIC (抗生物質) /  RIBOSOME (リボソーム) RIBOSOME (リボソーム) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 translation elongation factor activity / translation elongation factor activity /  GTPase activity / GTP binding / GTPase activity / GTP binding /  RNA binding / RNA binding /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |    THERMUS THERMOPHILUS (サーマス・サーモフィルス) THERMUS THERMOPHILUS (サーマス・サーモフィルス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Vogeley, L. / Palm, G.J. / Mesters, J.R. / Hilgenfeld, R. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2001 ジャーナル: J.Biol.Chem. / 年: 2001タイトル: Conformational Change of Elongation Factor TU Induced by Antibiotic Binding: Crystal Structure of the Complex between EF-TU:Gdp and Aurodox 著者: Vogeley, L. / Palm, G.J. / Mesters, J.R. / Hilgenfeld, R. #1:  ジャーナル: The Ribosome: Structure, Function, Antibiotics, and Cellular Interactions ジャーナル: The Ribosome: Structure, Function, Antibiotics, and Cellular Interactions年: 2000 タイトル: Insights Into the Gtpase Mechanism of EF-TU from Structural Studies 著者: Hilgenfeld, R. / Mesters, J. / Hogg, T. #3: ジャーナル: Nature / 年: 1993 タイトル: Crystal Structure of Active Elongation Factor TU Reveals Major Domain Rearrangements 著者: Berchtold, H. / Reshetnikova, L. / Reiser, C.O.A. / Schirmer, N.K. / Sprinzl, M. / Hilgenfeld, R. #4:  ジャーナル: J.Cryst.Growth / 年: 1992 ジャーナル: J.Cryst.Growth / 年: 1992タイトル: Crystals of Intact Elongation Factor TU from Thermus Thermophilus Diffracting to 1.45 Angstrom Resolution 著者: Reshetnikova, L. / Schirmer, N.K. / Reiser, C.O.A. / Berchtold, H. / Storm, R. / Hilgenfeld, R. / Sprinzl, M. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "D" IN EACH CHAIN ON SHEET RECORDS BELOW ... SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "D" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 6-STRANDED BARREL THIS IS REPRESENTED BY A 7-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ha3.cif.gz 1ha3.cif.gz | 182.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ha3.ent.gz pdb1ha3.ent.gz | 142.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ha3.json.gz 1ha3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ha/1ha3 https://data.pdbj.org/pub/pdb/validation_reports/ha/1ha3 ftp://data.pdbj.org/pub/pdb/validation_reports/ha/1ha3 ftp://data.pdbj.org/pub/pdb/validation_reports/ha/1ha3 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1exmS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (0.981974, -0.188989, -0.003066), ベクター  : : |
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 |  EF-Tu / EF-TU EF-Tu / EF-TU分子量: 44709.887 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: TERNARY COMPLEX WITH GUANOSINE DIPHOSPHATE AND AURODOX 由来: (組換発現)    THERMUS THERMOPHILUS (サーマス・サーモフィルス) THERMUS THERMOPHILUS (サーマス・サーモフィルス)株: HB8 / 遺伝子: TUFB / プラスミド: PEFTU10 / 発現宿主:   ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3) ESCHERICHIA COLI (大腸菌) / 株 (発現宿主): BL21(DE3)参照: UniProt: P07157, UniProt: Q5SHN6*PLUS,  DGTPアーゼ DGTPアーゼ |
|---|
-非ポリマー , 5種, 606分子 

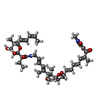






| #2: 化合物 |  グアノシン二リン酸 グアノシン二リン酸#3: 化合物 | #4: 化合物 | #5: 化合物 |  2-メルカプトエタノール 2-メルカプトエタノール#6: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.8 Å3/Da / 溶媒含有率: 55 % 解説: DOMAIN 1 AND 2/3 HAD TO BE USED AS SEPARATE SEARCH MODELS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8 詳細: HANGING DROP METHOD (PROTEIN SOLUTION:WELL 1:1). PROTEIN CONCENTRATION 10 MG/ML. WELL: 50 MM TRIS, 200 MM NAOAC, 23-25% PEG4000, PH 8.0 MOLAR RATIO EF-TU:GDP:AURODOX = 1:1:5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 19 ℃ / pH: 7 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 105 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: X13 / 波長: 0.913 / ビームライン: X13 / 波長: 0.913 |
| 検出器 | タイプ: X-RAY RESEARCH, HAMBURG, GERMANY / 検出器: CCD / 日付: 2000年9月15日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.913 Å / 相対比: 1 : 0.913 Å / 相対比: 1 |
| 反射 | 解像度: 2→60 Å / Num. obs: 67199 / % possible obs: 99.8 % / Observed criterion σ(I): -3 / 冗長度: 4.3 % / Biso Wilson estimate: 24.7 Å2 / Rsym value: 0.067 / Net I/σ(I): 18.2 |
| 反射 シェル | 解像度: 2→2.03 Å / 冗長度: 2.8 % / Mean I/σ(I) obs: 3.9 / Rsym value: 0.263 / % possible all: 98.7 |
| 反射 | *PLUS Num. measured all: 287768 / Rmerge(I) obs: 0.067 |
| 反射 シェル | *PLUS % possible obs: 98.7 % / Num. unique obs: 3322 / Num. measured obs: 9419 / Rmerge(I) obs: 0.263 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 1EXM 解像度: 2→20 Å / Rfactor Rfree error: 0.004 / Data cutoff high absF: 1000000 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 51.18 Å2 / ksol: 0.36 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 30.4 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2→2.07 Å / Rfactor Rfree error: 0.016 / Total num. of bins used: 10
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / バージョン: 1 / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj












