+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1h2b | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal Structure of the Alcohol Dehydrogenase from the Hyperthermophilic Archaeon Aeropyrum pernix at 1.65A Resolution | ||||||
 要素 要素 | ALCOHOL DEHYDROGENASE アルコールデヒドロゲナーゼ アルコールデヒドロゲナーゼ | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  ARCHAEA (古細菌) / ARCHAEA (古細菌) /  HYPERTHERMOPHILE (超好熱菌) / ALCOHOL DEHYDROGENASE OXIDOREDUCTASE / HYPERTHERMOPHILE (超好熱菌) / ALCOHOL DEHYDROGENASE OXIDOREDUCTASE /  ZINC (亜鉛) ZINC (亜鉛) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alcohol dehydrogenase (NAD+) activity /  アルコールデヒドロゲナーゼ / アルコールデヒドロゲナーゼ /  nucleotide binding / zinc ion binding nucleotide binding / zinc ion binding類似検索 - 分子機能 | ||||||
| 生物種 |    AEROPYRUM PERNIX (アエロピュルム・ペルニクス) AEROPYRUM PERNIX (アエロピュルム・ペルニクス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 1.62 Å 多波長異常分散 / 解像度: 1.62 Å | ||||||
 データ登録者 データ登録者 | Guy, J.E. / Isupov, M.N. / Littlechild, J.A. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2003 ジャーナル: J.Mol.Biol. / 年: 2003タイトル: The structure of an alcohol dehydrogenase from the hyperthermophilic archaeon Aeropyrum pernix. 著者: Guy, J.E. / Isupov, M.N. / Littlechild, J.A. #1: ジャーナル: Acta Crystallogr.,Sect.D / 年: 2003 タイトル: Crystallization and Preliminary X-Ray Diffraction Studies of a Novel Alcohol Dehydrogenase from the Hyperthermophilic Archaeon Aeropyrum Pernix. 著者: Guy, J.E. / Isupov, M.N. / Littlechild, J.A. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN ... SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN THE SHEET RECORDS BELOW, TWO SHEETS ARE DEFINED. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1h2b.cif.gz 1h2b.cif.gz | 163.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1h2b.ent.gz pdb1h2b.ent.gz | 127.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1h2b.json.gz 1h2b.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/h2/1h2b https://data.pdbj.org/pub/pdb/validation_reports/h2/1h2b ftp://data.pdbj.org/pub/pdb/validation_reports/h2/1h2b ftp://data.pdbj.org/pub/pdb/validation_reports/h2/1h2b | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1hszS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (0.11131, 0.99372, 0.0112), ベクター  : : |
- 要素
要素
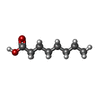
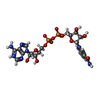
| #1: タンパク質 |  アルコールデヒドロゲナーゼ アルコールデヒドロゲナーゼ分子量: 39620.746 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)    AEROPYRUM PERNIX (アエロピュルム・ペルニクス) AEROPYRUM PERNIX (アエロピュルム・ペルニクス)プラスミド: PTRC 99 / 発現宿主:   Escherichia coli DH5[alpha] (大腸菌) Escherichia coli DH5[alpha] (大腸菌)参照: UniProt: Q9Y9P9,  アルコールデヒドロゲナーゼ アルコールデヒドロゲナーゼ#2: 化合物 |  カプリル酸 カプリル酸#3: 化合物 |  ニコチンアミドアデニンジヌクレオチド ニコチンアミドアデニンジヌクレオチド#4: 化合物 | ChemComp-ZN / #5: 水 | ChemComp-HOH / |  水 水構成要素の詳細 | BIOLOGICAL | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.2 Å3/Da / 溶媒含有率: 44 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | pH: 6.75 / 詳細: pH 6.75 | ||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, シッティングドロップ法 | ||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: BW7A / 波長: 1.0704 / ビームライン: BW7A / 波長: 1.0704 |
| 検出器 | タイプ: MARRESEARCH / 検出器: CCD / 日付: 2002年5月15日 |
| 放射 | プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.0704 Å / 相対比: 1 : 1.0704 Å / 相対比: 1 |
| 反射 | 解像度: 1.62→15 Å / Num. obs: 1008062 / % possible obs: 97.9 % / 冗長度: 11.7 % / Biso Wilson estimate: 21.8 Å2 / Rmerge(I) obs: 0.054 / Net I/σ(I): 28.9 |
| 反射 シェル | 解像度: 1.62→1.65 Å / Rmerge(I) obs: 0.932 / Mean I/σ(I) obs: 1.52 / % possible all: 95.6 |
| 反射 | *PLUS 最高解像度: 1.62 Å / 最低解像度: 15 Å / Num. obs: 88078 / 冗長度: 11.7 % / Rmerge(I) obs: 0.054 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  多波長異常分散 多波長異常分散開始モデル: PDB ENTRY 1HSZ 解像度: 1.62→15 Å / SU B: 1.546 / SU ML: 0.054 / 交差検証法: THROUGHOUT / ESU R: 0.086 / ESU R Free: 0.088
| ||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 25.697 Å2
| ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.62→15 Å
| ||||||||||||||||||||
| 精密化 | *PLUS Rfactor Rfree : 0.2033 / Rfactor Rwork : 0.2033 / Rfactor Rwork : 0.168 : 0.168 | ||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj