| 登録情報 | データベース: PDB / ID: 1euq
|
|---|
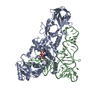
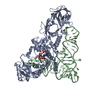
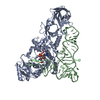
| タイトル | CRYSTAL STRUCTURE OF GLUTAMINYL-TRNA SYNTHETASE COMPLEXED WITH A TRNA-GLN MUTANT AND AN ACTIVE-SITE INHIBITOR |
|---|
 要素 要素 | - GLUTAMINYL TRNA
- GLUTAMINYL-TRNA SYNTHETASE
 QARS QARS
|
|---|
 キーワード キーワード | ligase/RNA /  TRNA SYNTHETASE (アミノアシルtRNA合成酵素) / TRNA SYNTHETASE (アミノアシルtRNA合成酵素) /  GLUTAMINE (グルタミン) / TRNAGLN / GLUTAMINE (グルタミン) / TRNAGLN /  E. COLI (大腸菌) / RNA-PROTEIN COMPLEX / ligase-RNA COMPLEX E. COLI (大腸菌) / RNA-PROTEIN COMPLEX / ligase-RNA COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 グルタミンtRNAリガーゼ / グルタミンtRNAリガーゼ /  glutamine-tRNA ligase activity / glutaminyl-tRNA aminoacylation / glutamyl-tRNA aminoacylation / glutamine-tRNA ligase activity / glutaminyl-tRNA aminoacylation / glutamyl-tRNA aminoacylation /  ATP binding / ATP binding /  細胞質基質類似検索 - 分子機能 細胞質基質類似検索 - 分子機能 Glutamine-tRNA ligase, bacterial / Glutamine-tRNA synthetase / Glutamine-tRNA ligase, bacterial / Glutamine-tRNA synthetase /  Glutamine-tRNA ligase, alpha-bundle domain superfamily / Glutamyl-tRNA Synthetase; domain 2 / Glutamyl-trna Synthetase; Domain 2 / Glutamyl-tRNA Synthetase; domain 3 / Glutamyl-tRNA Synthetase; Domain 3 / Glutamine-tRNA ligase, alpha-bundle domain superfamily / Glutamyl-tRNA Synthetase; domain 2 / Glutamyl-trna Synthetase; Domain 2 / Glutamyl-tRNA Synthetase; domain 3 / Glutamyl-tRNA Synthetase; Domain 3 /  Ribosomal Protein L25; Chain P / Ribosomal Protein L25; Chain P /  : / tRNA synthetases class I (E and Q), anti-codon binding domain ... : / tRNA synthetases class I (E and Q), anti-codon binding domain ... Glutamine-tRNA ligase, bacterial / Glutamine-tRNA synthetase / Glutamine-tRNA ligase, bacterial / Glutamine-tRNA synthetase /  Glutamine-tRNA ligase, alpha-bundle domain superfamily / Glutamyl-tRNA Synthetase; domain 2 / Glutamyl-trna Synthetase; Domain 2 / Glutamyl-tRNA Synthetase; domain 3 / Glutamyl-tRNA Synthetase; Domain 3 / Glutamine-tRNA ligase, alpha-bundle domain superfamily / Glutamyl-tRNA Synthetase; domain 2 / Glutamyl-trna Synthetase; Domain 2 / Glutamyl-tRNA Synthetase; domain 3 / Glutamyl-tRNA Synthetase; Domain 3 /  Ribosomal Protein L25; Chain P / Ribosomal Protein L25; Chain P /  : / tRNA synthetases class I (E and Q), anti-codon binding domain / Glutamyl/glutaminyl-tRNA synthetase, class Ib, anti-codon binding domain / tRNA synthetases class I (E and Q), anti-codon binding domain / : / tRNA synthetases class I (E and Q), anti-codon binding domain / Glutamyl/glutaminyl-tRNA synthetase, class Ib, anti-codon binding domain / tRNA synthetases class I (E and Q), anti-codon binding domain /  Ribosomal Protein L25; Chain P / Glutamyl/glutaminyl-tRNA synthetase / Glutamyl/glutaminyl-tRNA synthetase, class Ib, catalytic domain / tRNA synthetases class I (E and Q), catalytic domain / Ribosomal Protein L25; Chain P / Glutamyl/glutaminyl-tRNA synthetase / Glutamyl/glutaminyl-tRNA synthetase, class Ib, catalytic domain / tRNA synthetases class I (E and Q), catalytic domain /  Aminoacyl-tRNA synthetase, class I, conserved site / Aminoacyl-transfer RNA synthetases class-I signature. / HUPs / Rossmann-like alpha/beta/alpha sandwich fold / Aminoacyl-tRNA synthetase, class I, conserved site / Aminoacyl-transfer RNA synthetases class-I signature. / HUPs / Rossmann-like alpha/beta/alpha sandwich fold /  Ribosomal protein L25/Gln-tRNA synthetase, N-terminal / Ribosomal protein L25/Gln-tRNA synthetase, N-terminal /  Ribosomal protein L25/Gln-tRNA synthetase, anti-codon-binding domain superfamily / Ribosomal protein L25/Gln-tRNA synthetase, anti-codon-binding domain superfamily /  Alpha-Beta Complex / Alpha-Beta Complex /  Βバレル / Βバレル /  ロスマンフォールド / Orthogonal Bundle / 3-Layer(aba) Sandwich / Mainly Beta / Mainly Alpha / Alpha Beta類似検索 - ドメイン・相同性 ロスマンフォールド / Orthogonal Bundle / 3-Layer(aba) Sandwich / Mainly Beta / Mainly Alpha / Alpha Beta類似検索 - ドメイン・相同性5'-O-[N-(L-GLUTAMINYL)-SULFAMOYL]ADENOSINE /  リボ核酸 / RNA (> 10) / Glutamine--tRNA ligase類似検索 - 構成要素 リボ核酸 / RNA (> 10) / Glutamine--tRNA ligase類似検索 - 構成要素 |
|---|
| 生物種 |    Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 3.1 Å シンクロトロン / 解像度: 3.1 Å |
|---|
 データ登録者 データ登録者 | Sherlin, L.D. / Bullock, T.L. / Newberry, K.J. / Lipman, R.S.A. / Hou, Y.-M. / Beijer, B. / Sproat, B.S. / Perona, J.J. |
|---|
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2000 ジャーナル: J.Mol.Biol. / 年: 2000
タイトル: Influence of transfer RNA tertiary structure on aminoacylation efficiency by glutaminyl and cysteinyl-tRNA synthetases.
著者: Sherlin, L.D. / Bullock, T.L. / Newberry, K.J. / Lipman, R.S. / Hou, Y.M. / Beijer, B. / Sproat, B.S. / Perona, J.J. |
|---|
| 履歴 | | 登録 | 2000年4月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2000年6月4日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2011年12月28日 | Group: Advisory |
|---|
| 改定 1.4 | 2024年2月7日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRNA SYNTHETASE (アミノアシルtRNA合成酵素) /
TRNA SYNTHETASE (アミノアシルtRNA合成酵素) /  GLUTAMINE (グルタミン) / TRNAGLN /
GLUTAMINE (グルタミン) / TRNAGLN /  E. COLI (大腸菌) / RNA-PROTEIN COMPLEX / ligase-RNA COMPLEX
E. COLI (大腸菌) / RNA-PROTEIN COMPLEX / ligase-RNA COMPLEX 機能・相同性情報
機能・相同性情報 グルタミンtRNAリガーゼ /
グルタミンtRNAリガーゼ /  glutamine-tRNA ligase activity / glutaminyl-tRNA aminoacylation / glutamyl-tRNA aminoacylation /
glutamine-tRNA ligase activity / glutaminyl-tRNA aminoacylation / glutamyl-tRNA aminoacylation /  ATP binding /
ATP binding /  細胞質基質
細胞質基質
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  シンクロトロン / 解像度: 3.1 Å
シンクロトロン / 解像度: 3.1 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2000
ジャーナル: J.Mol.Biol. / 年: 2000 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1euq.cif.gz
1euq.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1euq.ent.gz
pdb1euq.ent.gz PDB形式
PDB形式 1euq.json.gz
1euq.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/eu/1euq
https://data.pdbj.org/pub/pdb/validation_reports/eu/1euq ftp://data.pdbj.org/pub/pdb/validation_reports/eu/1euq
ftp://data.pdbj.org/pub/pdb/validation_reports/eu/1euq リンク
リンク 集合体
集合体
 要素
要素 QARS / E.C.6.1.1.18 / GLNRS / GLUTAMINE-TRNA LIGASE
QARS / E.C.6.1.1.18 / GLNRS / GLUTAMINE-TRNA LIGASE
 Escherichia coli (大腸菌) / 発現宿主:
Escherichia coli (大腸菌) / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P00962,
Escherichia coli (大腸菌) / 参照: UniProt: P00962,  グルタミンtRNAリガーゼ
グルタミンtRNAリガーゼ X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.2 / 波長: 0.98
/ ビームライン: 5.0.2 / 波長: 0.98  : 0.98 Å / 相対比: 1
: 0.98 Å / 相対比: 1  解析
解析 X-PLOR / バージョン: 3.851 / 分類: refinement
X-PLOR / バージョン: 3.851 / 分類: refinement ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj