| 登録情報 | データベース: PDB / ID: 1dul
|
|---|
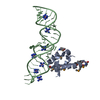
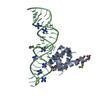
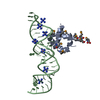
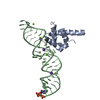
| タイトル | STRUCTURE OF THE RIBONUCLEOPROTEIN CORE OF THE E. COLI SIGNAL RECOGNITION PARTICLE |
|---|
 要素 要素 | - 4.5 S RNA DOMAIN IV
- Signal recognition particle protein
 シグナル認識粒子 シグナル認識粒子
|
|---|
 キーワード キーワード |  SIGNALING PROTEIN/RNA / protein-RNA complex / SIGNALING PROTEIN/RNA / protein-RNA complex /  double helix / double helix /  tetraloop / tetraloop /  internal loop / internal loop /  signal recognition particle (シグナル認識粒子) / SRP / signal recognition particle (シグナル認識粒子) / SRP /  ribonucleoprotein (核タンパク質) / ribonucleoprotein (核タンパク質) /  SIGNALING PROTEIN-RNA COMPLEX SIGNALING PROTEIN-RNA COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 シグナル認識粒子 / シグナル認識粒子 /  signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane /  ribonucleoprotein complex / ribonucleoprotein complex /  GTPase activity / GTP binding / GTPase activity / GTP binding /  ATP hydrolysis activity / ATP hydrolysis activity /  細胞質基質類似検索 - 分子機能 細胞質基質類似検索 - 分子機能 Signal recognition particle, SRP54 subunit, M-domain / Signal recognition particle, SRP54 subunit, M-domain /  Signal recognition particle protein / Signal recognition particle protein /  Signal recognition particle, SRP54 subunit / Signal recognition particle, SRP54 subunit /  Signal recognition particle, SRP54 subunit, M-domain / Signal recognition particle, SRP54 subunit, M-domain /  Signal recognition particle, SRP54 subunit, M-domain superfamily / Signal peptide binding domain / SRP54-type proteins GTP-binding domain signature. / Signal recognition particle SRP54, helical bundle / Signal recognition particle SRP54, N-terminal domain superfamily / SRP54-type protein, helical bundle domain ... Signal recognition particle, SRP54 subunit, M-domain superfamily / Signal peptide binding domain / SRP54-type proteins GTP-binding domain signature. / Signal recognition particle SRP54, helical bundle / Signal recognition particle SRP54, N-terminal domain superfamily / SRP54-type protein, helical bundle domain ... Signal recognition particle, SRP54 subunit, M-domain / Signal recognition particle, SRP54 subunit, M-domain /  Signal recognition particle protein / Signal recognition particle protein /  Signal recognition particle, SRP54 subunit / Signal recognition particle, SRP54 subunit /  Signal recognition particle, SRP54 subunit, M-domain / Signal recognition particle, SRP54 subunit, M-domain /  Signal recognition particle, SRP54 subunit, M-domain superfamily / Signal peptide binding domain / SRP54-type proteins GTP-binding domain signature. / Signal recognition particle SRP54, helical bundle / Signal recognition particle SRP54, N-terminal domain superfamily / SRP54-type protein, helical bundle domain / SRP54-type protein, helical bundle domain / Signal recognition particle, SRP54 subunit, M-domain superfamily / Signal peptide binding domain / SRP54-type proteins GTP-binding domain signature. / Signal recognition particle SRP54, helical bundle / Signal recognition particle SRP54, N-terminal domain superfamily / SRP54-type protein, helical bundle domain / SRP54-type protein, helical bundle domain /  Signal recognition particle, SRP54 subunit, GTPase domain / SRP54-type protein, GTPase domain / SRP54-type protein, GTPase domain / 434 Repressor (Amino-terminal Domain) / ATPases associated with a variety of cellular activities / AAA+ ATPase domain / P-loop containing nucleoside triphosphate hydrolase / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 Signal recognition particle, SRP54 subunit, GTPase domain / SRP54-type protein, GTPase domain / SRP54-type protein, GTPase domain / 434 Repressor (Amino-terminal Domain) / ATPases associated with a variety of cellular activities / AAA+ ATPase domain / P-loop containing nucleoside triphosphate hydrolase / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 : / : /  リボ核酸 / RNA (> 10) / リボ核酸 / RNA (> 10) /  Signal recognition particle protein類似検索 - 構成要素 Signal recognition particle protein類似検索 - 構成要素 |
|---|
| 生物種 |    Escherichia coli (大腸菌) Escherichia coli (大腸菌)
synthetic construct (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.8 Å シンクロトロン / 解像度: 1.8 Å |
|---|
 データ登録者 データ登録者 | Batey, R.T. / Rambo, R.P. / Lucast, L. / Rha, B. / Doudna, J.A. |
|---|
 引用 引用 |  ジャーナル: Science / 年: 2000 ジャーナル: Science / 年: 2000
タイトル: Crystal structure of the ribonucleoprotein core of the signal recognition particle.
著者: Batey, R.T. / Rambo, R.P. / Lucast, L. / Rha, B. / Doudna, J.A. |
|---|
| 履歴 | | 登録 | 2000年1月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2000年2月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 2.0 | 2019年11月6日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Database references / Derived calculations / Experimental preparation / Non-polymer description / Polymer sequence / Source and taxonomy / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / entity_name_com / entity_poly / entity_poly_seq / entity_src_gen / exptl_crystal / ndb_struct_na_base_pair / ndb_struct_na_base_pair_step / pdbx_entity_src_syn / pdbx_nonpoly_scheme / pdbx_poly_seq_scheme / pdbx_struct_conn_angle / pdbx_struct_mod_residue / pdbx_unobs_or_zero_occ_atoms / pdbx_unobs_or_zero_occ_residues / pdbx_validate_polymer_linkage / struct_conf / struct_conn / struct_ref / struct_ref_seq / struct_ref_seq_dif / struct_site_gen
Item: _chem_comp.formula / _chem_comp.formula_weight ..._chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_fragment / _entity_name_com.name / _entity_poly.pdbx_seq_one_letter_code / _entity_poly.pdbx_seq_one_letter_code_can / _entity_src_gen.pdbx_beg_seq_num / _entity_src_gen.pdbx_end_seq_num / _entity_src_gen.pdbx_gene_src_gene / _entity_src_gen.pdbx_seq_type / _exptl_crystal.density_Matthews / _ndb_struct_na_base_pair.hbond_type_12 / _ndb_struct_na_base_pair.hbond_type_28 / _ndb_struct_na_base_pair.propeller / _ndb_struct_na_base_pair_step.roll / _ndb_struct_na_base_pair_step.twist / _pdbx_nonpoly_scheme.asym_id / _pdbx_nonpoly_scheme.auth_seq_num / _pdbx_nonpoly_scheme.ndb_seq_num / _pdbx_nonpoly_scheme.pdb_seq_num / _pdbx_nonpoly_scheme.pdb_strand_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_unobs_or_zero_occ_atoms.label_seq_id / _struct_conf.beg_label_seq_id / _struct_conf.end_label_seq_id / _struct_ref.db_code / _struct_ref.db_name / _struct_ref.entity_id / _struct_ref.pdbx_align_begin / _struct_ref.pdbx_db_accession / _struct_ref.pdbx_seq_one_letter_code / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_ref_seq.pdbx_auth_seq_align_beg / _struct_ref_seq.pdbx_auth_seq_align_end / _struct_ref_seq.pdbx_db_accession / _struct_ref_seq.pdbx_strand_id / _struct_ref_seq.seq_align_beg / _struct_ref_seq.seq_align_end / _struct_site_gen.auth_seq_id / _struct_site_gen.label_seq_id |
|---|
| 改定 2.1 | 2020年10月14日 | Group: Derived calculations / Structure summary
カテゴリ: audit_author / pdbx_struct_conn_angle ...audit_author / pdbx_struct_conn_angle / struct_conn / struct_site
Item: _audit_author.name / _pdbx_struct_conn_angle.ptnr1_auth_asym_id ..._audit_author.name / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード SIGNALING PROTEIN/RNA / protein-RNA complex /
SIGNALING PROTEIN/RNA / protein-RNA complex /  double helix /
double helix /  tetraloop /
tetraloop /  internal loop /
internal loop /  signal recognition particle (シグナル認識粒子) / SRP /
signal recognition particle (シグナル認識粒子) / SRP /  ribonucleoprotein (核タンパク質) /
ribonucleoprotein (核タンパク質) /  SIGNALING PROTEIN-RNA COMPLEX
SIGNALING PROTEIN-RNA COMPLEX 機能・相同性情報
機能・相同性情報 シグナル認識粒子 /
シグナル認識粒子 /  signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane /
signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane /  ribonucleoprotein complex /
ribonucleoprotein complex /  GTPase activity / GTP binding /
GTPase activity / GTP binding /  ATP hydrolysis activity /
ATP hydrolysis activity /  細胞質基質
細胞質基質
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) X線回折 /
X線回折 /  シンクロトロン / 解像度: 1.8 Å
シンクロトロン / 解像度: 1.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Science / 年: 2000
ジャーナル: Science / 年: 2000 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1dul.cif.gz
1dul.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1dul.ent.gz
pdb1dul.ent.gz PDB形式
PDB形式 1dul.json.gz
1dul.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/du/1dul
https://data.pdbj.org/pub/pdb/validation_reports/du/1dul ftp://data.pdbj.org/pub/pdb/validation_reports/du/1dul
ftp://data.pdbj.org/pub/pdb/validation_reports/du/1dul リンク
リンク 集合体
集合体
 要素
要素 シグナル認識粒子 / Fifty-four homolog / Ffh / p48
シグナル認識粒子 / Fifty-four homolog / Ffh / p48
 Escherichia coli (大腸菌) / 遺伝子: ffh / 発現宿主:
Escherichia coli (大腸菌) / 遺伝子: ffh / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: P0AGD7
Escherichia coli (大腸菌) / 参照: UniProt: P0AGD7 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X4A / 波長: 0.9793,0.9787,0.9667
/ ビームライン: X4A / 波長: 0.9793,0.9787,0.9667 解析
解析 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj


































