+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1df7 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | DIHYDROFOLATE REDUCTASE OF MYCOBACTERIUM TUBERCULOSIS COMPLEXED WITH NADPH AND METHOTREXATE | ||||||
 要素 要素 | DIHYDROFOLATE REDUCTASE ジヒドロ葉酸レダクターゼ ジヒドロ葉酸レダクターゼ | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  DIHYDROFOLATE REDUCTASE (ジヒドロ葉酸レダクターゼ) / STRUCTURE-BASED INHIBITOR DESIGN / FOLATEANALOGS / DIHYDROFOLATE REDUCTASE (ジヒドロ葉酸レダクターゼ) / STRUCTURE-BASED INHIBITOR DESIGN / FOLATEANALOGS /  ROSSMANN FOLD (ロスマンフォールド) / ROSSMANN FOLD (ロスマンフォールド) /  NICOTINAMIDE ADENINE DINUCLEOTIDE (ニコチンアミドアデニンジヌクレオチド) / NICOTINAMIDE ADENINE DINUCLEOTIDE (ニコチンアミドアデニンジヌクレオチド) /  METHOTREXATE (メトトレキサート) / METHOTREXATE (メトトレキサート) /  TUBERCULOSIS (結核) TUBERCULOSIS (結核) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dihydrofolate metabolic process /  NADP+ binding / folic acid metabolic process / glycine biosynthetic process / NADP+ binding / folic acid metabolic process / glycine biosynthetic process /  ジヒドロ葉酸レダクターゼ / ジヒドロ葉酸レダクターゼ /  dihydrofolate reductase activity / tetrahydrofolate biosynthetic process / one-carbon metabolic process / dihydrofolate reductase activity / tetrahydrofolate biosynthetic process / one-carbon metabolic process /  NADP binding / NADP binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.7 Å シンクロトロン / 解像度: 1.7 Å | ||||||
 データ登録者 データ登録者 | Li, R. / Sirawaraporn, R. / Chitnumsub, P. / Sirawaraporn, W. / Wooden, J. / Athappilly, F. / Turley, S. / Hol, W.G. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2000 ジャーナル: J.Mol.Biol. / 年: 2000タイトル: Three-dimensional structure of M. tuberculosis dihydrofolate reductase reveals opportunities for the design of novel tuberculosis drugs. 著者: Li, R. / Sirawaraporn, R. / Chitnumsub, P. / Sirawaraporn, W. / Wooden, J. / Athappilly, F. / Turley, S. / Hol, W.G. #1:  ジャーナル: Biochemistry / 年: 1997 ジャーナル: Biochemistry / 年: 1997タイトル: Comparison of Two Independent Crystal Structures of Human Dihydrofolate Reductase Ternary Complexes Reduced with Nicotinamide Adenine Dinucleotide Phosphate and the Very Tight-Binding Inhibitor PT523 著者: Cody, V. / Galitsky, N. / Luft, J.R. / Pangborn, W. / Rosowsky, A. / Blakley, R.L. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1df7.cif.gz 1df7.cif.gz | 52.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1df7.ent.gz pdb1df7.ent.gz | 36.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1df7.json.gz 1df7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/df/1df7 https://data.pdbj.org/pub/pdb/validation_reports/df/1df7 ftp://data.pdbj.org/pub/pdb/validation_reports/df/1df7 ftp://data.pdbj.org/pub/pdb/validation_reports/df/1df7 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  ジヒドロ葉酸レダクターゼ / DHFR ジヒドロ葉酸レダクターゼ / DHFR分子量: 17660.992 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌)参照: UniProt: P0A546, UniProt: P9WNX1*PLUS,  ジヒドロ葉酸レダクターゼ ジヒドロ葉酸レダクターゼ |
|---|
-非ポリマー , 5種, 202分子 

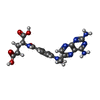






| #2: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩 | ||
|---|---|---|---|
| #3: 化合物 | ChemComp-NDP /  ニコチンアミドアデニンジヌクレオチドリン酸 ニコチンアミドアデニンジヌクレオチドリン酸 | ||
| #4: 化合物 | ChemComp-MTX /  メトトレキサート メトトレキサート | ||
| #5: 化合物 | ChemComp-GOL /  グリセリン グリセリン#6: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.05 Å3/Da / 溶媒含有率: 59.64 % | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法 / pH: 4.5 詳細: AMMONIUM SULFATE, SODIUM ACETATE, GLYCEROL, NADPH, METHOTREXATE, POTASSIUM PHOSPHATE, DTT, POTASSIUM CHLORIDE, pH 4.5, VAPOR DIFFUSION, temperature 277K | ||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 1.0188 / ビームライン: 19-ID / 波長: 1.0188 |
| 検出器 | タイプ: SBC-2 / 検出器: CCD / 日付: 1998年9月17日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.0188 Å / 相対比: 1 : 1.0188 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→50 Å / Num. all: 23394 / Num. obs: 109952 / % possible obs: 99.7 % / 冗長度: 4.7 % / Rmerge(I) obs: 0.042 / Net I/σ(I): 27 |
| 反射 シェル | 解像度: 1.7→1.78 Å / 冗長度: 4.7 % / Rmerge(I) obs: 0.263 / % possible all: 99.7 |
| 反射 | *PLUS Num. obs: 23394 / Num. measured all: 109952 |
| 反射 シェル | *PLUS % possible obs: 99.7 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.7→50 Å / σ(F): 1 / 立体化学のターゲット値: ENGH & HUBER
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / 分類: refinement X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS % reflection Rfree: 10 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





