+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1cld | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | DNA-binding protein | ||||||
 要素 要素 | CD2-LAC9 | ||||||
 キーワード キーワード |  TRANSCRIPTION REGULATION / ZINC-BINDING DOMAIN TRANSCRIPTION REGULATION / ZINC-BINDING DOMAIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報galactose metabolic process / DNA-binding transcription factor activity, RNA polymerase II-specific / DNA-templated transcription / positive regulation of transcription by RNA polymerase II /  DNA binding / zinc ion binding / DNA binding / zinc ion binding /  細胞核 細胞核類似検索 - 分子機能 | ||||||
| 生物種 |   Kluyveromyces lactis (酵母) Kluyveromyces lactis (酵母) | ||||||
| 手法 |  溶液NMR 溶液NMR | ||||||
 データ登録者 データ登録者 | Gardner, K.H. / Coleman, J.E. | ||||||
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 1995 ジャーナル: Nat.Struct.Biol. / 年: 1995タイトル: Solution structure of the Kluyveromyces lactis LAC9 Cd2 Cys6 DNA-binding domain. 著者: Gardner, K.H. / Anderson, S.F. / Coleman, J.E. #1:  ジャーナル: J.Biomol.NMR / 年: 1994 ジャーナル: J.Biomol.NMR / 年: 1994タイトル: 113Cd-1H Heterotocsy: A Method for Determining Metal-Protein Connectivities 著者: Gardner, K.H. / Coleman, J.E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1cld.cif.gz 1cld.cif.gz | 316.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1cld.ent.gz pdb1cld.ent.gz | 269.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1cld.json.gz 1cld.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cl/1cld https://data.pdbj.org/pub/pdb/validation_reports/cl/1cld ftp://data.pdbj.org/pub/pdb/validation_reports/cl/1cld ftp://data.pdbj.org/pub/pdb/validation_reports/cl/1cld | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 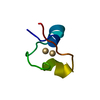
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| Atom site foot note | 1: CIS PROLINE - PRO 110 MODEL 1 / 2: CIS PROLINE - PRO 110 MODEL 2 / 3: CIS PROLINE - PRO 110 MODEL 3 / 4: CIS PROLINE - PRO 110 MODEL 4 / 5: CIS PROLINE - PRO 110 MODEL 5 / 6: CIS PROLINE - PRO 110 MODEL 6 / 7: CIS PROLINE - PRO 110 MODEL 7 / 8: CIS PROLINE - PRO 110 MODEL 8 / 9: CIS PROLINE - PRO 110 MODEL 9 / 10: CIS PROLINE - PRO 110 MODEL 10 / 11: CIS PROLINE - PRO 110 MODEL 11 / 12: CIS PROLINE - PRO 110 MODEL 12 / 13: CIS PROLINE - PRO 110 MODEL 13 / 14: CIS PROLINE - PRO 110 MODEL 14 / 15: CIS PROLINE - PRO 110 MODEL 15 / 16: CIS PROLINE - PRO 110 MODEL 16 / 17: CIS PROLINE - PRO 110 MODEL 17 / 18: CIS PROLINE - PRO 110 MODEL 18 / 19: CIS PROLINE - PRO 110 MODEL 19 / 20: CIS PROLINE - PRO 110 MODEL 20 / 21: CIS PROLINE - PRO 110 MODEL 21 / 22: CIS PROLINE - PRO 110 MODEL 22 / 23: CIS PROLINE - PRO 110 MODEL 23 / 24: CIS PROLINE - PRO 110 MODEL 24 / 25: CIS PROLINE - PRO 110 MODEL 25 / 26: CIS PROLINE - PRO 110 MODEL 26 / 27: CIS PROLINE - PRO 110 MODEL 27 / 28: CIS PROLINE - PRO 110 MODEL 28 / 29: CIS PROLINE - PRO 110 MODEL 29 | |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 7105.403 Da / 分子数: 1 / 変異: INITIATOR MET / 由来タイプ: 組換発現 / 由来: (組換発現)   Kluyveromyces lactis (酵母) / 発現宿主: Kluyveromyces lactis (酵母) / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P08657 Escherichia coli (大腸菌) / 参照: UniProt: P08657 |
|---|---|
| #2: 化合物 |
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR |
|---|
- 試料調製
試料調製
結晶化 | *PLUS 手法: その他 / 詳細: NMR |
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR software | 名称:  X-PLOR / バージョン: 3.1 / 開発者: BRUNGER / 分類: 精密化 X-PLOR / バージョン: 3.1 / 開発者: BRUNGER / 分類: 精密化 | ||||||||||||
| NMRアンサンブル | 登録したコンフォーマーの数: 29 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj

