+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9895 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of human T cell receptor-CD3 complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of lymphocyte apoptotic process / gamma-delta T cell receptor complex / Fc-gamma receptor III complex / T cell anergy / positive regulation of cell-cell adhesion mediated by integrin / T cell activation involved in immune response / positive regulation of T cell anergy / gamma-delta T cell activation / Fc-gamma receptor signaling pathway / CD4-positive, alpha-beta T cell proliferation ...regulation of lymphocyte apoptotic process / gamma-delta T cell receptor complex / Fc-gamma receptor III complex / T cell anergy / positive regulation of cell-cell adhesion mediated by integrin / T cell activation involved in immune response / positive regulation of T cell anergy / gamma-delta T cell activation / Fc-gamma receptor signaling pathway / CD4-positive, alpha-beta T cell proliferation / negative thymic T cell selection / positive regulation of CD4-positive, alpha-beta T cell proliferation / alpha-beta T cell receptor complex / positive regulation of protein localization to cell surface / positive thymic T cell selection / signal complex assembly / Nef and signal transduction / positive regulation of cell-matrix adhesion / T cell receptor complex / smoothened signaling pathway / establishment or maintenance of cell polarity / Translocation of ZAP-70 to Immunological synapse / Phosphorylation of CD3 and TCR zeta chains / positive regulation of interleukin-4 production / dendrite development / protein complex oligomerization / alpha-beta T cell activation / Generation of second messenger molecules / FCGR activation / immunological synapse / Co-inhibition by PD-1 / Role of phospholipids in phagocytosis / T cell receptor binding / positive regulation of T cell proliferation / T cell costimulation / positive regulation of interleukin-2 production / endomembrane system / cerebellum development / FCGR3A-mediated IL10 synthesis / positive regulation of calcium-mediated signaling / T cell activation / cell surface receptor protein tyrosine kinase signaling pathway / protein tyrosine kinase binding / negative regulation of smoothened signaling pathway / FCGR3A-mediated phagocytosis / response to bacterium / apoptotic signaling pathway / clathrin-coated endocytic vesicle membrane / calcium-mediated signaling / peptide antigen binding / Regulation of actin dynamics for phagocytic cup formation / SH3 domain binding / positive regulation of type II interferon production / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / transmembrane signaling receptor activity / protein transport / cell-cell junction / Downstream TCR signaling / Cargo recognition for clathrin-mediated endocytosis / signaling receptor complex adaptor activity / T cell receptor signaling pathway / Clathrin-mediated endocytosis / cell body / protein-containing complex assembly / regulation of apoptotic process / dendritic spine / adaptive immune response / cell surface receptor signaling pathway / G protein-coupled receptor signaling pathway / protein heterodimerization activity / external side of plasma membrane / negative regulation of gene expression / positive regulation of gene expression / protein kinase binding / cell surface / endoplasmic reticulum / Golgi apparatus / protein homodimerization activity / identical protein binding / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Dong D / Zheng L | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2019 ジャーナル: Nature / 年: 2019タイトル: Structural basis of assembly of the human T cell receptor-CD3 complex. 著者: De Dong / Lvqin Zheng / Jianquan Lin / Bailing Zhang / Yuwei Zhu / Ningning Li / Shuangyu Xie / Yuhang Wang / Ning Gao / Zhiwei Huang /  要旨: The αβ T cell receptor (TCR), in association with the CD3γε-CD3δε-CD3ζζ signalling hexamer, is the primary determinant of T cell development and activation, and of immune responses to foreign ...The αβ T cell receptor (TCR), in association with the CD3γε-CD3δε-CD3ζζ signalling hexamer, is the primary determinant of T cell development and activation, and of immune responses to foreign antigens. The mechanism of assembly of the TCR-CD3 complex remains unknown. Here we report a cryo-electron microscopy structure of human TCRαβ in complex with the CD3 hexamer at 3.7 Å resolution. The structure contains the complete extracellular domains and all the transmembrane helices of TCR-CD3. The octameric TCR-CD3 complex is assembled with 1:1:1:1 stoichiometry of TCRαβ:CD3γε:CD3δε:CD3ζζ. Assembly of the extracellular domains of TCR-CD3 is mediated by the constant domains and connecting peptides of TCRαβ that pack against CD3γε-CD3δε, forming a trimer-like structure proximal to the plasma membrane. The transmembrane segment of the CD3 complex adopts a barrel-like structure formed by interaction of the two transmembrane helices of CD3ζζ with those of CD3γε and CD3δε. Insertion of the transmembrane helices of TCRαβ into the barrel-like structure via both hydrophobic and ionic interactions results in transmembrane assembly of the TCR-CD3 complex. Together, our data reveal the structural basis for TCR-CD3 complex assembly, providing clues to TCR triggering and a foundation for rational design of immunotherapies that target the complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9895.map.gz emd_9895.map.gz | 78.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9895-v30.xml emd-9895-v30.xml emd-9895.xml emd-9895.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9895.png emd_9895.png | 39.6 KB | ||
| Filedesc metadata |  emd-9895.cif.gz emd-9895.cif.gz | 7.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9895 http://ftp.pdbj.org/pub/emdb/structures/EMD-9895 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9895 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9895 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9895.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9895.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.057 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
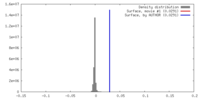
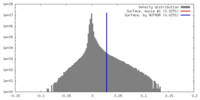
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : complex
| 全体 | 名称: complex |
|---|---|
| 要素 |
|
-超分子 #1: complex
| 超分子 | 名称: complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: T-cell surface glycoprotein CD3 zeta chain
| 分子 | 名称: T-cell surface glycoprotein CD3 zeta chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.723439 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKWKALFTAA ILQAQLPITE AQSFGLLDPK LCYLLDGILF IYGVILTALF LRVKFSRSAD APAYQQGQNQ LYNELNLGRR EEYDVLDKR RGRDPEMGGK PQRRKNPQEG LYNELQKDKM AEAYSEIGMK GERRRGKGHD GLYQGLSTAT KDTYDALHMQ A LPPR UniProtKB: T-cell surface glycoprotein CD3 zeta chain |
-分子 #2: T-cell surface glycoprotein CD3 delta chain
| 分子 | 名称: T-cell surface glycoprotein CD3 delta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 18.949537 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEHSTFLSGL VLATLLSQVS PFKIPIEELE DRVFVNCNTS ITWVEGTVGT LLSDITRLDL GKRILDPRGI YRCNGTDIYK DKESTVQVH YRMCQSCVEL DPATVAGIIV TDVIATLLLA LGVFCFAGHE TGRLSGAADT QALLRNDQVY QPLRDRDDAQ Y SHLGGNWA RNK UniProtKB: T-cell surface glycoprotein CD3 delta chain |
-分子 #3: T-cell surface glycoprotein CD3 epsilon chain
| 分子 | 名称: T-cell surface glycoprotein CD3 epsilon chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.174227 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MQSGTHWRVL GLCLLSVGVW GQDGNEEMGG ITQTPYKVSI SGTTVILTCP QYPGSEILWQ HNDKNIGGDE DDKNIGSDED HLSLKEFSE LEQSGYYVCY PRGSKPEDAN FYLYLRARVC ENCMEMDVMS VATIVIVDIC ITGGLLLLVY YWSKNRKAKA K PVTRGAGA ...文字列: MQSGTHWRVL GLCLLSVGVW GQDGNEEMGG ITQTPYKVSI SGTTVILTCP QYPGSEILWQ HNDKNIGGDE DDKNIGSDED HLSLKEFSE LEQSGYYVCY PRGSKPEDAN FYLYLRARVC ENCMEMDVMS VATIVIVDIC ITGGLLLLVY YWSKNRKAKA K PVTRGAGA GGRQRGQNKE RPPPVPNPDY EPIRKGQRDL YSGLNQRRI UniProtKB: T-cell surface glycoprotein CD3 epsilon chain |
-分子 #4: T-cell surface glycoprotein CD3 gamma chain
| 分子 | 名称: T-cell surface glycoprotein CD3 gamma chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.493457 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEQGKGLAVL ILAIILLQGT LAQSIKGNHL VKVYDYQEDG SVLLTCDAEA KNITWFKDGK MIGFLTEDKK KWNLGSNAKD PRGMYQCKG SQNKSKPLQV YYRMCQNCIE LNAATISGFL FAEIVSIFVL AVGVYFIAGQ DGVRQSRASD KQTLLPNDQL Y QPLKDRED DQYSHLQGNQ LRRN UniProtKB: T-cell surface glycoprotein CD3 gamma chain |
-分子 #5: T cell receptor alpha variable 12-3,Possible J 11 gene segment,T ...
| 分子 | 名称: T cell receptor alpha variable 12-3,Possible J 11 gene segment,T cell receptor alpha constant タイプ: protein_or_peptide / ID: 5 詳細: fusion protein of residues 22-114 from A0A0B4J271, residues 116-132 from A0N4Z6 and residues 134-273 from P01848. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 28.308662 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QQKEVEQDPG PLSVPEGAIV SLNCTYSNSA FQYFMWYRQY SRKGPELLMY TYSSGNKEDG RFTAQVDKSS KYISLFIRDS QPSDSATYL CAMSKGYSTL TFGKGTMLLV SPDIQNPDPA VYQLRDSKSS DKSVCLFTDF DSQTNVSQSK DSDVYITDKT V LDMRSMDF ...文字列: QQKEVEQDPG PLSVPEGAIV SLNCTYSNSA FQYFMWYRQY SRKGPELLMY TYSSGNKEDG RFTAQVDKSS KYISLFIRDS QPSDSATYL CAMSKGYSTL TFGKGTMLLV SPDIQNPDPA VYQLRDSKSS DKSVCLFTDF DSQTNVSQSK DSDVYITDKT V LDMRSMDF KSNSAVAWSN KSDFACANAF NNSIIPEDTF FPSPESSCDV KLVEKSFETD TNLNFQNLSV IGFRILLLKV AG FNLLMTL RLWSS UniProtKB: T cell receptor alpha variable 12-3, Possible J 11 gene segment, T cell receptor alpha chain constant |
-分子 #6: T cell receptor beta variable 6-5,M1-specific T cell receptor bet...
| 分子 | 名称: T cell receptor beta variable 6-5,M1-specific T cell receptor beta chain,T cell receptor beta constant 2 タイプ: protein_or_peptide / ID: 6 詳細: fusion protein of residues 22-112 from A0A0K0K1A5, residues 121-134 from P0DSE2 and residues 135-312 from A0A0G2JMB4. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.498463 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GVTQTPKFQV LKTGQSMTLQ CAQDMNHEYM SWYRQDPGMG LRLIHYSVGA GITDQGEVPN GYNVSRSTTE DFPLRLLSAA PSQTSVYFC ASRRRQGASG EQYFGPGTRL TVTEDLKNVF PPEVAVFEPS EAEISHTQKA TLVCLATGFY PDHVELSWWV N GKEVHSGV ...文字列: GVTQTPKFQV LKTGQSMTLQ CAQDMNHEYM SWYRQDPGMG LRLIHYSVGA GITDQGEVPN GYNVSRSTTE DFPLRLLSAA PSQTSVYFC ASRRRQGASG EQYFGPGTRL TVTEDLKNVF PPEVAVFEPS EAEISHTQKA TLVCLATGFY PDHVELSWWV N GKEVHSGV STDPQPLKEQ PALNDSRYCL SSRLRVSATF WQNPRNHFRC QVQFYGLSEN DEWTQDRAKP VTQIVSAEAW GR ADCGFTS ESYQQGVLSA TILYEILLGK ATLYAVLVSA LVLMAMVKRK DSRG UniProtKB: T cell receptor beta variable 6-5, M1-specific T cell receptor beta chain, T cell receptor beta constant 2 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 64.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: DARK FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)