+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9663 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
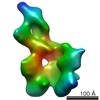
| タイトル | Saccharomyces cerevisiae SAGA complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SLIK (SAGA-like) complex / SAGA complex /  NuA4 histone acetyltransferase complex / NuA4 histone acetyltransferase complex /  クロマチンリモデリング / クロマチンリモデリング /  DNA修復 / DNA-templated transcription / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA修復 / DNA-templated transcription / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.9 Å クライオ電子顕微鏡法 / 解像度: 6.9 Å | |||||||||
 データ登録者 データ登録者 | Zheng XD / Liu GC / Guan HP / Li HT | |||||||||
 引用 引用 |  ジャーナル: Cell Discov / 年: 2019 ジャーナル: Cell Discov / 年: 2019タイトル: Architecture of SAGA complex. 著者: Gaochao Liu / Xiangdong Zheng / Haipeng Guan / Yong Cao / Hongyuan Qu / Junqing Kang / Xiangle Ren / Jianlin Lei / Meng-Qiu Dong / Xueming Li / Haitao Li /  | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9663.map.gz emd_9663.map.gz | 10 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9663-v30.xml emd-9663-v30.xml emd-9663.xml emd-9663.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9663_fsc.xml emd_9663_fsc.xml | 12.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9663.png emd_9663.png | 38.5 KB | ||
| マスクデータ |  emd_9663_msk_1.map emd_9663_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| その他 |  emd_9663_additional.map.gz emd_9663_additional.map.gz emd_9663_half_map_1.map.gz emd_9663_half_map_1.map.gz emd_9663_half_map_2.map.gz emd_9663_half_map_2.map.gz | 10 MB 141 MB 141 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9663 http://ftp.pdbj.org/pub/emdb/structures/EMD-9663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9663 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9663.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9663.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.401 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_9663_msk_1.map emd_9663_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
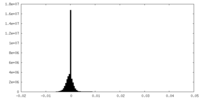
| 密度ヒストグラム |
-追加マップ: Z-plane reversed map, for docking and comparison.
| ファイル | emd_9663_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Z-plane reversed map, for docking and comparison. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
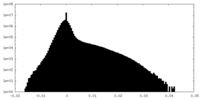
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_9663_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
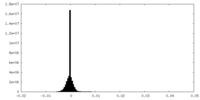
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_9663_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Saccharomyces cerevisiae SAGA (Spt-Ada-Gcn5-Acetyltransferase) complex
| 全体 | 名称: Saccharomyces cerevisiae SAGA (Spt-Ada-Gcn5-Acetyltransferase) complex |
|---|---|
| 要素 |
|
-超分子 #1: Saccharomyces cerevisiae SAGA (Spt-Ada-Gcn5-Acetyltransferase) complex
| 超分子 | 名称: Saccharomyces cerevisiae SAGA (Spt-Ada-Gcn5-Acetyltransferase) complex タイプ: complex / ID: 1 / 親要素: 0 / 詳細: The multi-functional co-activator SAGA complex |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: BYL4741 / Organelle: Nucleus / 細胞中の位置: Nucleus Saccharomyces cerevisiae (パン酵母) / 株: BYL4741 / Organelle: Nucleus / 細胞中の位置: Nucleus |
| 分子量 | 理論値: 1.8 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
詳細: 20mM HEPES pH 8.5 150mM Nacl | ||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 5.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 101.325 kPa 詳細: The grid was coated with a home-made thin continuous carbon film. | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.2 µm / 倍率(補正後): 81000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 0.1 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 Bright-field microscopy / Cs: 0.1 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 81000 |
| 特殊光学系 | 球面収差補正装置: Cs-corrector are used |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 3-30 / 実像数: 8526 / 平均露光時間: 0.25 sec. / 平均電子線量: 5.6 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X