[日本語] English
 万見
万見- EMDB-7009: Structure of an acid sensing ion channel in a resting state at high pH -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7009 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of an acid sensing ion channel in a resting state at high pH | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / Stimuli-sensing channels /  ligand-gated sodium channel activity / cellular response to pH / protein homotrimerization / sodium ion transmembrane transport / identical protein binding / ligand-gated sodium channel activity / cellular response to pH / protein homotrimerization / sodium ion transmembrane transport / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Gallus (ヤケイ属) / Gallus (ヤケイ属) /   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Yoder N / Yoshioka C / Gouaux E | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2018 ジャーナル: Nature / 年: 2018タイトル: Gating mechanisms of acid-sensing ion channels. 著者: Nate Yoder / Craig Yoshioka / Eric Gouaux /  要旨: Acid-sensing ion channels (ASICs) are trimeric, proton-gated and sodium-selective members of the epithelial sodium channel/degenerin (ENaC/DEG) superfamily of ion channels and are expressed ...Acid-sensing ion channels (ASICs) are trimeric, proton-gated and sodium-selective members of the epithelial sodium channel/degenerin (ENaC/DEG) superfamily of ion channels and are expressed throughout vertebrate central and peripheral nervous systems. Gating of ASICs occurs on a millisecond time scale and the mechanism involves three conformational states: high pH resting, low pH open and low pH desensitized. Existing X-ray structures of ASIC1a describe the conformations of the open and desensitized states, but the structure of the high pH resting state and detailed mechanisms of the activation and desensitization of the channel have remained elusive. Here we present structures of the high pH resting state of homotrimeric chicken (Gallus gallus) ASIC1a, determined by X-ray crystallography and single particle cryo-electron microscopy, and present a comprehensive molecular mechanism for proton-dependent gating in ASICs. In the resting state, the position of the thumb domain is further from the three-fold molecular axis, thereby expanding the 'acidic pocket' in comparison to the open and desensitized states. Activation therefore involves 'closure' of the thumb into the acidic pocket, expansion of the lower palm domain and an iris-like opening of the channel gate. Furthermore, we demonstrate how the β11-β12 linkers that demarcate the upper and lower palm domains serve as a molecular 'clutch', and undergo a simple rearrangement to permit rapid desensitization. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7009.map.gz emd_7009.map.gz | 23.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7009-v30.xml emd-7009-v30.xml emd-7009.xml emd-7009.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7009.png emd_7009.png | 148.5 KB | ||
| その他 |  emd_7009_additional.map.gz emd_7009_additional.map.gz emd_7009_half_map_1.map.gz emd_7009_half_map_1.map.gz emd_7009_half_map_2.map.gz emd_7009_half_map_2.map.gz | 28.3 MB 13.8 MB 13.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7009 http://ftp.pdbj.org/pub/emdb/structures/EMD-7009 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7009 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7009 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7009.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7009.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_7009_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
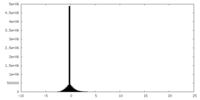
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_7009_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
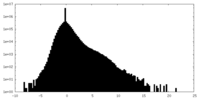
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_7009_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
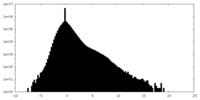
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Acid Sensing Ion Channel 1a
| 全体 | 名称: Acid Sensing Ion Channel 1a |
|---|---|
| 要素 |
|
-超分子 #1: Acid Sensing Ion Channel 1a
| 超分子 | 名称: Acid Sensing Ion Channel 1a / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Gallus (ヤケイ属) Gallus (ヤケイ属) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 180.03144 KDa |
-分子 #1: Acid-sensing ion channel 1
| 分子 | 名称: Acid-sensing ion channel 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Gallus gallus (ニワトリ) Gallus gallus (ニワトリ) |
| 分子量 | 理論値: 60.080324 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MMDLKVDEEE VDSGQPVSIQ AFASSSTLHG ISHIFSYERL SLKRVVWALC FMGSLALLAL VCTNRIQYYF LYPHVTKLDE VAATRLTFP AVTFCNLNEF RFSRVTKNDL YHAGELLALL NNRYEIPDTQ TADEKQLEIL QDKANFRNFK PKPFNMLEFY D RAGHDIRE ...文字列: MMDLKVDEEE VDSGQPVSIQ AFASSSTLHG ISHIFSYERL SLKRVVWALC FMGSLALLAL VCTNRIQYYF LYPHVTKLDE VAATRLTFP AVTFCNLNEF RFSRVTKNDL YHAGELLALL NNRYEIPDTQ TADEKQLEIL QDKANFRNFK PKPFNMLEFY D RAGHDIRE MLLSCFFRGE QCSPEDFKVV FTRYGKCYTF NAGQDGKPRL ITMKGGTGNG LEIMLDIQQD EYLPVWGETD ET SFEAGIK VQIHSQDEPP LIDQLGFGVA PGFQTFVSCQ EQRLIYLPPP WGDCKATTGD SEFYDTYSIT ACRIDCETRY LVE NCNCRM VHMPGDAPYC TPEQYKECAD PALDFLVEKD NEYCVCEMPC NVTRYGKELS MVKIPSKASA KYLAKKYNKS EQYI GENIL VLDIFFEALN YETIEQKKAY EVAGLLGDIG GQMGLFIGAS ILTVLELFDY AYEVIKHRLC RRGKCRKNHK RNNTD KGVA LSMDDVKRHN PCESLRGHPA GMTYAANILP HHPARGTFED FTC |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.2 mg/mL | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 詳細: 15 mA | ||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 撮影したグリッド数: 1 / 平均露光時間: 10.0 sec. / 平均電子線量: 45.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: Gctf |
|---|---|
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 2.1b1) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: cisTEM |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C3 (3回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM / 使用した粒子像数: 26117 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM / 使用した粒子像数: 26117 |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6ave: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X