+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Focus refined map of the swiveled head domain of the 30S subunit of ratcheted Listeria innocua ribosome in complex with HflXr and pe/E-tRNA (structure II-D) | |||||||||||||||
 マップデータ マップデータ | Focus refined cryo-EM map of Listeria innocua 30S head from Structure II-D | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  cryo-EM (低温電子顕微鏡法) / cryo-EM (低温電子顕微鏡法) /  recycling (リサイクル) / HflXr / time-resolved / recycling (リサイクル) / HflXr / time-resolved /  RIBOSOME (リボソーム) RIBOSOME (リボソーム) | |||||||||||||||
| 生物種 |   Listeria innocua (バクテリア) Listeria innocua (バクテリア) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||||||||
 データ登録者 データ登録者 | Seely SM / Basu RS / Gagnon MG | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2024 ジャーナル: Nucleic Acids Res / 年: 2024タイトル: Mechanistic insights into the alternative ribosome recycling by HflXr. 著者: Savannah M Seely / Ritwika S Basu / Matthieu G Gagnon /  要旨: During stress conditions such as heat shock and antibiotic exposure, ribosomes stall on messenger RNAs, leading to inhibition of protein synthesis. To remobilize ribosomes, bacteria use rescue ...During stress conditions such as heat shock and antibiotic exposure, ribosomes stall on messenger RNAs, leading to inhibition of protein synthesis. To remobilize ribosomes, bacteria use rescue factors such as HflXr, a homolog of the conserved housekeeping GTPase HflX that catalyzes the dissociation of translationally inactive ribosomes into individual subunits. Here we use time-resolved cryo-electron microscopy to elucidate the mechanism of ribosome recycling by Listeria monocytogenes HflXr. Within the 70S ribosome, HflXr displaces helix H69 of the 50S subunit and induces long-range movements of the platform domain of the 30S subunit, disrupting inter-subunit bridges B2b, B2c, B4, B7a and B7b. Our findings unveil a unique ribosome recycling strategy by HflXr which is distinct from that mediated by RRF and EF-G. The resemblance between HflXr and housekeeping HflX suggests that the alternative ribosome recycling mechanism reported here is universal in the prokaryotic kingdom. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42573.map.gz emd_42573.map.gz | 253.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42573-v30.xml emd-42573-v30.xml emd-42573.xml emd-42573.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_42573.png emd_42573.png | 167.1 KB | ||
| マスクデータ |  emd_42573_msk_1.map emd_42573_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42573.cif.gz emd-42573.cif.gz | 4.7 KB | ||
| その他 |  emd_42573_additional_1.map.gz emd_42573_additional_1.map.gz emd_42573_half_map_1.map.gz emd_42573_half_map_1.map.gz emd_42573_half_map_2.map.gz emd_42573_half_map_2.map.gz | 483.4 MB 475.6 MB 475.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42573 http://ftp.pdbj.org/pub/emdb/structures/EMD-42573 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42573 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42573 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42573.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42573.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focus refined cryo-EM map of Listeria innocua 30S head from Structure II-D | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42573_msk_1.map emd_42573_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
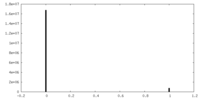
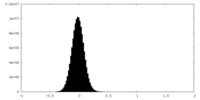
| 密度ヒストグラム |
-追加マップ: Focus refined map of Listeria innocua 30S head...
| ファイル | emd_42573_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focus refined map of Listeria innocua 30S head from Structure II-D (Sharpened map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
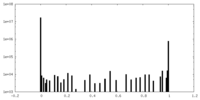
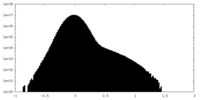
| 密度ヒストグラム |
-ハーフマップ: Focus refined map of Listeria innocua 30S head...
| ファイル | emd_42573_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focus refined map of Listeria innocua 30S head from Structure II-D (Half map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
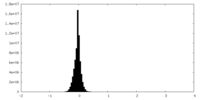
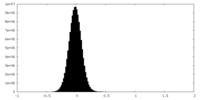
| 密度ヒストグラム |
-ハーフマップ: Focus refined map of Listeria innocua 30S head...
| ファイル | emd_42573_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focus refined map of Listeria innocua 30S head from Structure II-D (Half map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
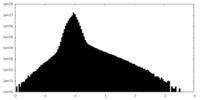
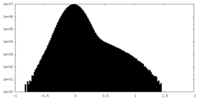
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Listeria innocua 70S ribosome in complex with HflXr and pe/E-tRNA
| 全体 | 名称: Listeria innocua 70S ribosome in complex with HflXr and pe/E-tRNA |
|---|---|
| 要素 |
|
-超分子 #1: Listeria innocua 70S ribosome in complex with HflXr and pe/E-tRNA
| 超分子 | 名称: Listeria innocua 70S ribosome in complex with HflXr and pe/E-tRNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#52 |
|---|---|
| 由来(天然) | 生物種:   Listeria innocua (バクテリア) Listeria innocua (バクテリア) |
| 分子量 | 理論値: 2.5 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 295 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 撮影したグリッド数: 1 / 実像数: 10151 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) |
| 最終 3次元分類 | ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 4.1.2) / 使用した粒子像数: 80381 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー


































 Z
Z Y
Y X
X