+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Hermes transposase bound to two right-ends of its DNA transposon. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  transposase / transpososome / BED domain / transposase / transpososome / BED domain /  protein-DNA complex (デオキシリボ核酸) / RECOMBINATION-DNA complex protein-DNA complex (デオキシリボ核酸) / RECOMBINATION-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nucleic acid metabolic process /  protein dimerization activity / protein dimerization activity /  DNA binding / DNA binding /  metal ion binding / metal ion binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Musca domestica (イエバエ) Musca domestica (イエバエ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.1 Å クライオ電子顕微鏡法 / 解像度: 5.1 Å | |||||||||
 データ登録者 データ登録者 | Lannes L / Dyda F | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Zinc-finger BED domains drive the formation of the active Hermes transpososome by asymmetric DNA binding. 著者: Laurie Lannes / Christopher M Furman / Alison B Hickman / Fred Dyda /  要旨: The Hermes DNA transposon is a member of the eukaryotic hAT superfamily, and its transposase forms a ring-shaped tetramer of dimers. Our investigation, combining biochemical, crystallography and cryo- ...The Hermes DNA transposon is a member of the eukaryotic hAT superfamily, and its transposase forms a ring-shaped tetramer of dimers. Our investigation, combining biochemical, crystallography and cryo-electron microscopy, and in-cell assays, shows that the full-length Hermes octamer extensively interacts with its transposon left-end through multiple BED domains of three Hermes protomers contributed by three dimers explaining the role of the unusual higher-order assembly. By contrast, the right-end is bound to no BED domains at all. Thus, this work supports a model in which Hermes multimerizes to gather enough BED domains to find its left-end among the abundant genomic DNA, facilitating the subsequent interaction with the right-end. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40553.map.gz emd_40553.map.gz | 86.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40553-v30.xml emd-40553-v30.xml emd-40553.xml emd-40553.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40553_fsc.xml emd_40553_fsc.xml | 10.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40553.png emd_40553.png | 42.3 KB | ||
| その他 |  emd_40553_additional_1.map.gz emd_40553_additional_1.map.gz emd_40553_half_map_1.map.gz emd_40553_half_map_1.map.gz emd_40553_half_map_2.map.gz emd_40553_half_map_2.map.gz | 83.1 MB 72.9 MB 72.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40553 http://ftp.pdbj.org/pub/emdb/structures/EMD-40553 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40553 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40553 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40553.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40553.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: half-maps processed with DeepEMhancer.
| ファイル | emd_40553_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-maps processed with DeepEMhancer. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
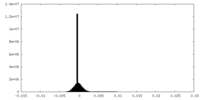
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_40553_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
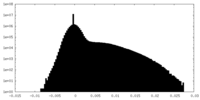
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40553_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Two right-end Hermes transpososome
| 全体 | 名称: Two right-end Hermes transpososome |
|---|---|
| 要素 |
|
-超分子 #1: Two right-end Hermes transpososome
| 超分子 | 名称: Two right-end Hermes transpososome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Hermes transposase tetramer of dimers complex bound to two transposon right-end DNAs. The complex was obtained by mixing the purified protein and the DNA. |
|---|---|
| 由来(天然) | 生物種:   Musca domestica (イエバエ) Musca domestica (イエバエ) |
| 分子量 | 理論値: 627 KDa |
-分子 #1: Hermes transposase
| 分子 | 名称: Hermes transposase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Musca domestica (イエバエ) Musca domestica (イエバエ) |
| 分子量 | 理論値: 70.21057 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MEKMDNLEVK AKINQGLYKI TPRHKGTSFI WNVLADIQKE DDTLVEGWVF CRKCEKVLKY TTRQTSNLCR HKCCASLKQS RELKTVSAD CKKEAIEKCA QWVVRDCRPF SAVSGSGFID MIKFFIKVGA EYGEHVNVEE LLPSPITLSR KVTSDAKEKK A LISREIKS ...文字列: MEKMDNLEVK AKINQGLYKI TPRHKGTSFI WNVLADIQKE DDTLVEGWVF CRKCEKVLKY TTRQTSNLCR HKCCASLKQS RELKTVSAD CKKEAIEKCA QWVVRDCRPF SAVSGSGFID MIKFFIKVGA EYGEHVNVEE LLPSPITLSR KVTSDAKEKK A LISREIKS AVEKDGASAT IDLWTDNYIK RNFLGVTLHY HENNELRDLI LGLKSLDFER STAENIYKKL KAIFSQFNVE DL SSIKFVT DRGANVVKSL ANNIRINCSS HLLSNVLENS FEETPELNMP ILACKNIVKY FKKANLQHRL RSSLKSECPT RWN STYTML RSILDNWESV IQILSEAGET QRIVHINKSI IQTMVNILDG FERIFKELQT CSSPSLCFVV PSILKVKEIC SPDV GDVAD IAKLKVNIIK NVRIIWEENL SIWHYTAFFF YPPALHMQQE KVAQIKEFCL SKMEDLELIN RMSSFNELSA TQLNQ SDSN SHNSIDLTSH SKDISTTSFF FPQLTQNNSR EPPVCPSDEF EFYRKEIVIL SEDFKVMEWW NLNSKKYPKL SKLALS LLS IPASSAASER TFSLAGNIIT EKRNRIGQQT VDSLLFLNSF YKNFCKLDI UniProtKB: Hermes transposase |
-分子 #2: DNA (55-MER)
| 分子 | 名称: DNA (55-MER) / タイプ: dna / ID: 2 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Musca domestica (イエバエ) Musca domestica (イエバエ) |
| 分子量 | 理論値: 16.909779 KDa |
| 配列 | 文字列: (DC)(DT)(DT)(DA)(DT)(DC)(DT)(DA)(DT)(DG) (DT)(DG)(DG)(DC)(DT)(DT)(DA)(DC)(DG)(DT) (DT)(DT)(DG)(DC)(DC)(DT)(DG)(DT)(DG) (DG)(DC)(DT)(DT)(DG)(DT)(DT)(DG)(DA)(DA) (DG) (DT)(DT)(DC)(DT)(DC) ...文字列: (DC)(DT)(DT)(DA)(DT)(DC)(DT)(DA)(DT)(DG) (DT)(DG)(DG)(DC)(DT)(DT)(DA)(DC)(DG)(DT) (DT)(DT)(DG)(DC)(DC)(DT)(DG)(DT)(DG) (DG)(DC)(DT)(DT)(DG)(DT)(DT)(DG)(DA)(DA) (DG) (DT)(DT)(DC)(DT)(DC)(DT)(DG)(DG) (DT)(DT)(DC)(DA)(DC)(DG)(DC) |
-分子 #3: DNA (46-MER)
| 分子 | 名称: DNA (46-MER) / タイプ: dna / ID: 3 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Musca domestica (イエバエ) Musca domestica (イエバエ) |
| 分子量 | 理論値: 14.197199 KDa |
| 配列 | 文字列: (DA)(DG)(DA)(DG)(DA)(DA)(DC)(DT)(DT)(DC) (DA)(DA)(DC)(DA)(DA)(DG)(DC)(DC)(DA)(DC) (DA)(DG)(DG)(DC)(DA)(DA)(DA)(DC)(DG) (DT)(DA)(DA)(DG)(DC)(DC)(DA)(DC)(DA)(DT) (DA) (DG)(DA)(DT)(DA)(DA)(DG) |
-分子 #4: DNA (8-MER)
| 分子 | 名称: DNA (8-MER) / タイプ: dna / ID: 4 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Musca domestica (イエバエ) Musca domestica (イエバエ) |
| 分子量 | 理論値: 2.162448 KDa |
| 配列 | 文字列: (DG)(DC)(DG)(DT)(DG)(DA)(DA) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | The complex was formed in vitro by mixing the purified protein with the DNA and further purified by size exclusion chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 9500 / 平均露光時間: 1.66 sec. / 平均電子線量: 48.7 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||||
| 得られたモデル |  PDB-8sjd: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X