+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of OmpC3-MlaA-MlaC Complex in MSP2N2 Nanodiscs | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  bacteria (細菌) / outer membrane / bacteria (細菌) / outer membrane /  phospholipid (リン脂質) / phospholipid (リン脂質) /  lipid asymmetry (脂質二重層) / lipid asymmetry (脂質二重層) /  membrane protein (膜タンパク質) / protein complex structure / channel / LIPID TRANSPORT membrane protein (膜タンパク質) / protein complex structure / channel / LIPID TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報intermembrane phospholipid transfer / phospholipid transport /  porin activity / pore complex / monoatomic ion transmembrane transport / cell outer membrane / virus receptor activity / outer membrane-bounded periplasmic space / receptor-mediated virion attachment to host cell / DNA damage response ...intermembrane phospholipid transfer / phospholipid transport / porin activity / pore complex / monoatomic ion transmembrane transport / cell outer membrane / virus receptor activity / outer membrane-bounded periplasmic space / receptor-mediated virion attachment to host cell / DNA damage response ...intermembrane phospholipid transfer / phospholipid transport /  porin activity / pore complex / monoatomic ion transmembrane transport / cell outer membrane / virus receptor activity / outer membrane-bounded periplasmic space / receptor-mediated virion attachment to host cell / DNA damage response / identical protein binding / porin activity / pore complex / monoatomic ion transmembrane transport / cell outer membrane / virus receptor activity / outer membrane-bounded periplasmic space / receptor-mediated virion attachment to host cell / DNA damage response / identical protein binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) | |||||||||
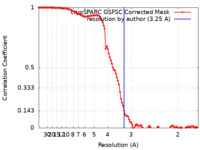
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.25 Å クライオ電子顕微鏡法 / 解像度: 3.25 Å | |||||||||
 データ登録者 データ登録者 | Yeow J / Luo M / Chng SS | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Molecular mechanism of phospholipid transport at the bacterial outer membrane interface. 著者: Jiang Yeow / Min Luo / Shu-Sin Chng /  要旨: The outer membrane (OM) of Gram-negative bacteria is an asymmetric lipid bilayer with outer leaflet lipopolysaccharides and inner leaflet phospholipids (PLs). This unique lipid asymmetry renders the ...The outer membrane (OM) of Gram-negative bacteria is an asymmetric lipid bilayer with outer leaflet lipopolysaccharides and inner leaflet phospholipids (PLs). This unique lipid asymmetry renders the OM impermeable to external insults, including antibiotics and bile salts. To maintain this barrier, the OmpC-Mla system removes mislocalized PLs from the OM outer leaflet, and transports them to the inner membrane (IM); in the first step, the OmpC-MlaA complex transfers PLs to the periplasmic chaperone MlaC, but mechanistic details are lacking. Here, we biochemically and structurally characterize the MlaA-MlaC transient complex. We map the interaction surfaces between MlaA and MlaC in Escherichia coli, and show that electrostatic interactions are important for MlaC recruitment to the OM. We further demonstrate that interactions with MlaC modulate conformational states in MlaA. Finally, we solve a 2.9-Å cryo-EM structure of a disulfide-trapped OmpC-MlaA-MlaC complex in nanodiscs, reinforcing the mechanism of MlaC recruitment, and highlighting membrane thinning as a plausible strategy for directing lipids for transport. Our work offers critical insights into retrograde PL transport by the OmpC-Mla system in maintaining OM lipid asymmetry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35253.map.gz emd_35253.map.gz | 62.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35253-v30.xml emd-35253-v30.xml emd-35253.xml emd-35253.xml | 27.4 KB 27.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35253_fsc.xml emd_35253_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35253.png emd_35253.png | 112.9 KB | ||
| マスクデータ |  emd_35253_msk_1.map emd_35253_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-35253.cif.gz emd-35253.cif.gz | 7.6 KB | ||
| その他 |  emd_35253_half_map_1.map.gz emd_35253_half_map_1.map.gz emd_35253_half_map_2.map.gz emd_35253_half_map_2.map.gz | 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35253 http://ftp.pdbj.org/pub/emdb/structures/EMD-35253 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35253 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35253 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35253.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35253.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.834 Å | ||||||||||||||||||||
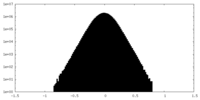
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_35253_msk_1.map emd_35253_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
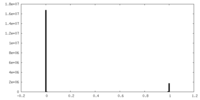
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_35253_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
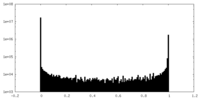
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35253_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
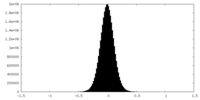
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : OmpC3-MlaA complex in MSP2N2 nanodiscs disulfide-bonded to MlaC
| 全体 | 名称: OmpC3-MlaA complex in MSP2N2 nanodiscs disulfide-bonded to MlaC |
|---|---|
| 要素 |
|
-超分子 #1: OmpC3-MlaA complex in MSP2N2 nanodiscs disulfide-bonded to MlaC
| 超分子 | 名称: OmpC3-MlaA complex in MSP2N2 nanodiscs disulfide-bonded to MlaC タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Outer membrane complex of 3OmpC with disulfide-trapped MlaA and MlaC reconstituted in Escherichia coli polar lipids and MSP2N2 nanodiscs |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K12 / 細胞中の位置: Outer membrane Escherichia coli (大腸菌) / 株: K12 / 細胞中の位置: Outer membrane |
| 分子量 | 理論値: 52 KDa |
-超分子 #2: Outer membrane porin C
| 超分子 | 名称: Outer membrane porin C / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 詳細: Trimeric OmpC porins in complex with MlaA at the outer membrane disulfide-trapped with MlaC |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K12 / 細胞中の位置: Outer membrane Escherichia coli (大腸菌) / 株: K12 / 細胞中の位置: Outer membrane |
-超分子 #3: Complex of MlaA disulfide trapped with MlaC
| 超分子 | 名称: Complex of MlaA disulfide trapped with MlaC / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 詳細: Intermembrane phospholipid transport system lipoprotein MlaA disulfide trapped with MlaC |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K12 / 細胞中の位置: Outer membrane Escherichia coli (大腸菌) / 株: K12 / 細胞中の位置: Outer membrane |
-分子 #1: Outer membrane porin C
| 分子 | 名称: Outer membrane porin C / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 株: K-12 Escherichia coli K-12 (大腸菌) / 株: K-12 |
| 分子量 | 理論値: 38.336242 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: AEVYNKDGNK LDLYGKVDGL HYFSDNKDVD GDQTYMRLGF KGETQVTDQL TGYGQWEYQI QGNSAENENN SWTRVAFAGL KFQDVGSFD YGRNYGVVYD VTSWTDVLPE FGGDTYGSDN FMQQRGNGFA TYRNTDFFGL VDGLNFAVQY QGKNGNPSGE G FTSGVTNN ...文字列: AEVYNKDGNK LDLYGKVDGL HYFSDNKDVD GDQTYMRLGF KGETQVTDQL TGYGQWEYQI QGNSAENENN SWTRVAFAGL KFQDVGSFD YGRNYGVVYD VTSWTDVLPE FGGDTYGSDN FMQQRGNGFA TYRNTDFFGL VDGLNFAVQY QGKNGNPSGE G FTSGVTNN GRDALRQNGD GVGGSITYDY EGFGIGGAIS SSKRTDAQNT AAYIGNGDRA ETYTGGLKYD ANNIYLAAQY TQ TYNATRV GSLGWANKAQ NFEAVAQYQF DFGLRPSLAY LQSKGKNLGR GYDDEDILKY VDVGATYYFN KNMSTYVDYK INL LDDNQF TRDAGINTDN IVALGLVYQF UniProtKB: Outer membrane porin C |
-分子 #2: Intermembrane phospholipid transport system lipoprotein MlaA
| 分子 | 名称: Intermembrane phospholipid transport system lipoprotein MlaA タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 株: K-12 Escherichia coli K-12 (大腸菌) / 株: K-12 |
| 分子量 | 理論値: 26.298336 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: CASSGTDQQG RSDPLEGFNR TMYNFNFNVL DPYIVRPVAV AWRDYVPQPA RNGLSNFTGN LEEPAVMVNY FLQGDPYQGM VHFTRFFLN TILGMGGFID VAGMANPKLQ RTEPHRFGST LGHYGVGYGP YVQLPFYGSF TLRDDGGDMA DGFYPVLSWL T WPMSVGKW ...文字列: CASSGTDQQG RSDPLEGFNR TMYNFNFNVL DPYIVRPVAV AWRDYVPQPA RNGLSNFTGN LEEPAVMVNY FLQGDPYQGM VHFTRFFLN TILGMGGFID VAGMANPKLQ RTEPHRFGST LGHYGVGYGP YVQLPFYGSF TLRDDGGDMA DGFYPVLSWL T WPMSVGKW TLEGIETRAQ LLDSDGLLRC SSDPYIMVRE AYFQRHDFIA NGGELKPQEN PNAQAIQDDL KDIDSE UniProtKB: Intermembrane phospholipid transport system lipoprotein MlaA |
-分子 #3: Intermembrane phospholipid transport system binding protein MlaC
| 分子 | 名称: Intermembrane phospholipid transport system binding protein MlaC タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 株: K-12 Escherichia coli K-12 (大腸菌) / 株: K-12 |
| 分子量 | 理論値: 22.903922 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: AADQTNPYKL MDEAAQKTFD RLKNEQPQIR ANPDYLRTIV DQELLPYVQV KYAGALVLGQ YYKSATPAQR EAYFAAFREY LKQAYGQAL AMYHGQTYQI APEQPLGDKT IVPIRVTIID PNGRPPVRLD FQWRKNSQTG NWQAYDMIAE GCSMITTKQN E WGTLLRTK ...文字列: AADQTNPYKL MDEAAQKTFD RLKNEQPQIR ANPDYLRTIV DQELLPYVQV KYAGALVLGQ YYKSATPAQR EAYFAAFREY LKQAYGQAL AMYHGQTYQI APEQPLGDKT IVPIRVTIID PNGRPPVRLD FQWRKNSQTG NWQAYDMIAE GCSMITTKQN E WGTLLRTK GIDGLTAQLK SISQQKITLE EKKLEHHHHH H UniProtKB: Intermembrane phospholipid transport system binding protein MlaC |
-分子 #4: (2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[(...
| 分子 | 名称: (2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[(2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-carboxy-2-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-5-[[(3~{R})-3- ...名称: (2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-[(2~{R},4~{R},5~{R},6~{R})-6-[(1~{R})-1,2-bis(oxidanyl)ethyl]-2-carboxy-2-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-5-[[(3~{R})-3-dodecanoyloxytetradecanoyl]amino]-6-[[(2~{R},3~{S},4~{R},5~{R},6~{R})-3-oxidanyl-5-[[(3~{R})-3-oxidanyltetradecanoyl]amino]-4-[(3~{R})-3-oxidanyltetradecanoyl]oxy-6-phosphonooxy-oxan-2-yl]methoxy]-3-phosphonooxy-4-[(3~{R})-3-tetradecanoyloxytetradecanoyl]oxy-oxan-2-yl]methoxy]-5-oxidanyl-oxan-4-yl]oxy-4,5-bis(oxidanyl)oxane-2-carboxylic acid タイプ: ligand / ID: 4 / コピー数: 3 / 式: KDL |
|---|---|
| 分子量 | 理論値: 2.238718 KDa |
| Chemical component information |  ChemComp-KDL: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 12 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Tris-buffered saline (TBS) buffer (20 mM Tris HCl pH 8.0, 150 mM NaCl) | |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Tridiem 4K / エネルギーフィルター - スリット幅: 20 eV 詳細: Gatan GIF post-column energy filter operated in zero-loss mode |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 6018 / 平均露光時間: 5.99 sec. / 平均電子線量: 90.0 e/Å2 詳細: Images were collected in movie-mode at 50 frames per image |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 69.1 当てはまり具合の基準: Cross-correlation coefficient | ||||||||||||||||||
| 得られたモデル |  PDB-8i8x: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X