+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human ATP synthase F1 domain, state 3b | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of cell adhesion involved in substrate-bound cell migration / Formation of ATP by chemiosmotic coupling / Cristae formation / ATP biosynthetic process /  angiostatin binding / angiostatin binding /  Mitochondrial protein import / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / proton-transporting ATP synthase complex / cellular response to interleukin-7 ...negative regulation of cell adhesion involved in substrate-bound cell migration / Formation of ATP by chemiosmotic coupling / Cristae formation / ATP biosynthetic process / Mitochondrial protein import / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / proton-transporting ATP synthase complex / cellular response to interleukin-7 ...negative regulation of cell adhesion involved in substrate-bound cell migration / Formation of ATP by chemiosmotic coupling / Cristae formation / ATP biosynthetic process /  angiostatin binding / angiostatin binding /  Mitochondrial protein import / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / proton-transporting ATP synthase complex / cellular response to interleukin-7 / Mitochondrial protein import / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase, stator stalk / proton-transporting ATP synthase complex / cellular response to interleukin-7 /  酸化的リン酸化 / response to muscle activity / mitochondrial proton-transporting ATP synthase complex / mitochondrial proton-transporting ATP synthase complex, catalytic sector F(1) / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / negative regulation of endothelial cell proliferation / proton motive force-driven ATP synthesis / cellular response to nitric oxide / proton-transporting ATP synthase complex, catalytic core F(1) / positive regulation of blood vessel endothelial cell migration / MHC class I protein binding / 酸化的リン酸化 / response to muscle activity / mitochondrial proton-transporting ATP synthase complex / mitochondrial proton-transporting ATP synthase complex, catalytic sector F(1) / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / negative regulation of endothelial cell proliferation / proton motive force-driven ATP synthesis / cellular response to nitric oxide / proton-transporting ATP synthase complex, catalytic core F(1) / positive regulation of blood vessel endothelial cell migration / MHC class I protein binding /  ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton transmembrane transport / proton-transporting ATP synthase activity, rotational mechanism / cellular response to dexamethasone stimulus / generation of precursor metabolites and energy / ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton transmembrane transport / proton-transporting ATP synthase activity, rotational mechanism / cellular response to dexamethasone stimulus / generation of precursor metabolites and energy /  ADP binding / ADP binding /  regulation of intracellular pH / regulation of intracellular pH /  ミトコンドリア / Transcriptional activation of mitochondrial biogenesis / lipid metabolic process / osteoblast differentiation / ミトコンドリア / Transcriptional activation of mitochondrial biogenesis / lipid metabolic process / osteoblast differentiation /  血管新生 / response to ethanol / 血管新生 / response to ethanol /  ミトコンドリア内膜 / ミトコンドリア内膜 /  protease binding / protease binding /  ミトコンドリアマトリックス / ミトコンドリアマトリックス /  脂質ラフト / 脂質ラフト /  細胞膜 / 細胞膜 /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  RNA binding / extracellular exosome / RNA binding / extracellular exosome /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   human (ヒト) human (ヒト) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.02 Å クライオ電子顕微鏡法 / 解像度: 3.02 Å | |||||||||||||||
 データ登録者 データ登録者 | Lai Y / Zhang Y / Liu F / Gao Y / Gong H / Rao Z | |||||||||||||||
| 資金援助 |  中国, 4件 中国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Structure of the human ATP synthase. 著者: Yuezheng Lai / Yuying Zhang / Shan Zhou / Jinxu Xu / Zhanqiang Du / Ziyan Feng / Long Yu / Ziqing Zhao / Weiwei Wang / Yanting Tang / Xiuna Yang / Luke W Guddat / Fengjiang Liu / Yan Gao / ...著者: Yuezheng Lai / Yuying Zhang / Shan Zhou / Jinxu Xu / Zhanqiang Du / Ziyan Feng / Long Yu / Ziqing Zhao / Weiwei Wang / Yanting Tang / Xiuna Yang / Luke W Guddat / Fengjiang Liu / Yan Gao / Zihe Rao / Hongri Gong /   要旨: Biological energy currency ATP is produced by FF-ATP synthase. However, the molecular mechanism for human ATP synthase action remains unknown. Here, we present snapshot images for three main ...Biological energy currency ATP is produced by FF-ATP synthase. However, the molecular mechanism for human ATP synthase action remains unknown. Here, we present snapshot images for three main rotational states and one substate of human ATP synthase using cryoelectron microscopy. These structures reveal that the release of ADP occurs when the β subunit of FF-ATP synthase is in the open conformation, showing how ADP binding is coordinated during synthesis. The accommodation of the symmetry mismatch between F and F motors is resolved by the torsional flexing of the entire complex, especially the γ subunit, and the rotational substep of the c subunit. Water molecules are identified in the inlet and outlet half-channels, suggesting that the proton transfer in these two half-channels proceed via a Grotthus mechanism. Clinically relevant mutations are mapped to the structure, showing that they are mainly located at the subunit-subunit interfaces, thus causing instability of the complex. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34576.map.gz emd_34576.map.gz | 484.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34576-v30.xml emd-34576-v30.xml emd-34576.xml emd-34576.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34576.png emd_34576.png | 119.8 KB | ||
| その他 |  emd_34576_half_map_1.map.gz emd_34576_half_map_1.map.gz emd_34576_half_map_2.map.gz emd_34576_half_map_2.map.gz | 475 MB 475 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34576 http://ftp.pdbj.org/pub/emdb/structures/EMD-34576 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34576 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34576 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8h9pMC  8h9eC  8h9fC  8h9gC  8h9iC  8h9jC  8h9kC  8h9lC  8h9mC  8h9nC  8h9qC  8h9rC  8h9sC  8h9tC  8h9uC  8h9vC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34576.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34576.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.73 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34576_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
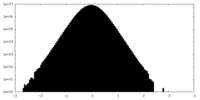
| 投影像・断面図 |
| ||||||||||||
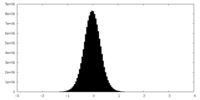
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34576_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
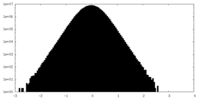
| 投影像・断面図 |
| ||||||||||||
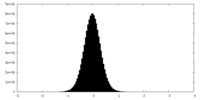
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human ATP synthase
| 全体 | 名称: Human ATP synthase ATP合成酵素 ATP合成酵素 |
|---|---|
| 要素 |
|
-超分子 #1: Human ATP synthase
| 超分子 | 名称: Human ATP synthase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 600 KDa |
-分子 #1: ATP synthase subunit alpha, mitochondrial
| 分子 | 名称: ATP synthase subunit alpha, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   human (ヒト) human (ヒト) |
| 分子量 | 理論値: 55.27616 KDa |
| 配列 | 文字列: QKTGTAEMSS ILEERILGAD TSVDLEETGR VLSIGDGIAR VHGLRNVQAE EMVEFSSGLK GMSLNLEPDN VGVVVFGNDK LIKEGDIVK RTGAIVDVPV GEELLGRVVD ALGNAIDGKG PIGSKTRRRV GLKAPGIIPR ISVREPMQTG IKAVDSLVPI G RGQRELII ...文字列: QKTGTAEMSS ILEERILGAD TSVDLEETGR VLSIGDGIAR VHGLRNVQAE EMVEFSSGLK GMSLNLEPDN VGVVVFGNDK LIKEGDIVK RTGAIVDVPV GEELLGRVVD ALGNAIDGKG PIGSKTRRRV GLKAPGIIPR ISVREPMQTG IKAVDSLVPI G RGQRELII GDRQTGKTSI AIDTIINQKR FNDGSDEKKK LYCIYVAIGQ KRSTVAQLVK RLTDADAMKY TIVVSATASD AA PLQYLAP YSGCSMGEYF RDNGKHALII YDDLSKQAVA YRQMSLLLRR PPGREAYPGD VFYLHSRLLE RAAKMNDAFG GGS LTALPV IETQAGDVSA YIPTNVISIT DGQIFLETEL FYKGIRPAIN VGLSVSRVGS AAQTRAMKQV AGTMKLELAQ YREV AAFAQ FGSDLDAATQ QLLSRGVRLT ELLKQGQYSP MAIEEQVAVI YAGVRGYLDK LEPSKITKFE NAFLSHVVSQ HQALL GTIR ADGKISEQSD AKLKEIVTNF LAGFEA |
-分子 #2: ATP synthase subunit beta, mitochondrial
| 分子 | 名称: ATP synthase subunit beta, mitochondrial / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   human (ヒト) human (ヒト) |
| 分子量 | 理論値: 51.821965 KDa |
| 配列 | 文字列: AQTSPSPKAG AATGRIVAVI GAVVDVQFDE GLPPILNALE VQGRETRLVL EVAQHLGEST VRTIAMDGTE GLVRGQKVLD SGAPIKIPV GPETLGRIMN VIGEPIDERG PIKTKQFAPI HAEAPEFMEM SVEQEILVTG IKVVDLLAPY AKGGKIGLFG G AGVGKTVL ...文字列: AQTSPSPKAG AATGRIVAVI GAVVDVQFDE GLPPILNALE VQGRETRLVL EVAQHLGEST VRTIAMDGTE GLVRGQKVLD SGAPIKIPV GPETLGRIMN VIGEPIDERG PIKTKQFAPI HAEAPEFMEM SVEQEILVTG IKVVDLLAPY AKGGKIGLFG G AGVGKTVL IMELINNVAK AHGGYSVFAG VGERTREGND LYHEMIESGV INLKDATSKV ALVYGQMNEP PGARARVALT GL TVAEYFR DQEGQDVLLF IDNIFRFTQA GSEVSALLGR IPSAVGYQPT LATDMGTMQE RITTTKKGSI TSVQAIYVPA DDL TDPAPA TTFAHLDATT VLSRAIAELG IYPAVDPLDS TSRIMDPNIV GSEHYDVARG VQKILQDYKS LQDIIAILGM DELS EEDKL TVSRARKIQR FLSQPFQVAE VFTGHMGKLV PLKETIKGFQ QILAGEYDHL PEQAFYMVGP IEEAVAKADK LAEEH SS |
-分子 #3: ATP synthase subunit gamma, mitochondrial
| 分子 | 名称: ATP synthase subunit gamma, mitochondrial / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   human (ヒト) human (ヒト) |
| 分子量 | 理論値: 30.207752 KDa |
| 配列 | 文字列: ATLKDITRRL KSIKNIQKIT KSMKMVAAAK YARAERELKP ARIYGLGSLA LYEKADIKGP EDKKKHLLIG VSSDRGLCGA IHSSIAKQM KSEVATLTAA GKEVMLVGIG DKIRGILYRT HSDQFLVAFK EVGRKPPTFG DASVIALELL NSGYEFDEGS I IFNKFRSV ...文字列: ATLKDITRRL KSIKNIQKIT KSMKMVAAAK YARAERELKP ARIYGLGSLA LYEKADIKGP EDKKKHLLIG VSSDRGLCGA IHSSIAKQM KSEVATLTAA GKEVMLVGIG DKIRGILYRT HSDQFLVAFK EVGRKPPTFG DASVIALELL NSGYEFDEGS I IFNKFRSV ISYKTEEKPI FSLNTVASAD SMSIYDDIDA DVLQNYQEYN LANIIYYSLK ESTTSEQSAR MTAMDNASKN AS EMIDKLT LTFNRTRQAV ITKELIEIIS GAAALD |
-分子 #4: ATP synthase subunit O, mitochondrial
| 分子 | 名称: ATP synthase subunit O, mitochondrial / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   human (ヒト) human (ヒト) |
| 分子量 | 理論値: 20.904488 KDa |
| 配列 | 文字列: FAKLVRPPVQ VYGIEGRYAT ALYSAASKQN KLEQVEKELL RVAQILKEPK VAASVLNPYV KRSIKVKSLN DITAKERFSP LTTNLINLL AENGRLSNTQ GVVSAFSTMM SVHRGEVPCT VTSASPLEEA TLSELKTVLK SFLSQGQVLK LEAKTDPSIL G GMIVRIGE ...文字列: FAKLVRPPVQ VYGIEGRYAT ALYSAASKQN KLEQVEKELL RVAQILKEPK VAASVLNPYV KRSIKVKSLN DITAKERFSP LTTNLINLL AENGRLSNTQ GVVSAFSTMM SVHRGEVPCT VTSASPLEEA TLSELKTVLK SFLSQGQVLK LEAKTDPSIL G GMIVRIGE KYVDMSVKTK IQKLGRAMRE IV |
-分子 #5: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: AlphaFold |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.02 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 23272 |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X