+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of Chikungunya Virus Nonstructural Protein 1 with inhibitor FHA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell filopodium /  ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase / ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity / polynucleotide 5'-phosphatase activity /  polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity ...host cell filopodium / polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity ...host cell filopodium /  ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase / ADP-ribose 1''-phosphate phosphatase / mRNA methyltransferase activity / mRNA 5'-triphosphate monophosphatase activity / mRNA 5'-phosphatase /  polynucleotide 5'-phosphatase activity / polynucleotide 5'-phosphatase activity /  polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / cysteine-type peptidase activity / polynucleotide adenylyltransferase / poly(A) RNA polymerase activity / regulation of cytoskeleton organization / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / 7-methylguanosine mRNA capping / symbiont-mediated suppression of host JAK-STAT cascade via inhibition of STAT1 activity / cysteine-type peptidase activity /  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / host cell cytoplasmic vesicle membrane / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / host cell cytoplasmic vesicle membrane /  転移酵素; リンを含む基を移すもの; 核酸を移すもの / nucleoside-triphosphate phosphatase / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / nucleoside-triphosphate phosphatase /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; システインプロテアーゼ /  RNA helicase activity / RNA helicase activity /  ヘリカーゼ / ヘリカーゼ /  RNA依存性RNAポリメラーゼ / viral RNA genome replication / RNA依存性RNAポリメラーゼ / viral RNA genome replication /  RNA-dependent RNA polymerase activity / DNA-templated transcription / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / GTP binding / host cell plasma membrane / RNA-dependent RNA polymerase activity / DNA-templated transcription / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / host cell nucleus / GTP binding / host cell plasma membrane /  ATP hydrolysis activity / ATP hydrolysis activity /  タンパク質分解 / タンパク質分解 /  RNA binding / RNA binding /  ATP binding / ATP binding /  生体膜 / 生体膜 /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Chikungunya virus strain S27-African prototype (チクングニア熱) Chikungunya virus strain S27-African prototype (チクングニア熱) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.62 Å クライオ電子顕微鏡法 / 解像度: 2.62 Å | |||||||||
 データ登録者 データ登録者 | Zhang K / Law MCY / Nguyen TM / Tan YB / Wirawan M / Law YS / Luo DH | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2022 ジャーナル: Cell Rep / 年: 2022タイトル: Molecular basis of specific viral RNA recognition and 5'-end capping by the Chikungunya virus nsP1. 著者: Kuo Zhang / Michelle Cheok Yien Law / Trinh Mai Nguyen / Yaw Bia Tan / Melissa Wirawan / Yee-Song Law / Lak Shin Jeong / Dahai Luo /    要旨: Many viruses encode RNA-modifying enzymes to edit the 5' end of viral RNA to mimic the cellular mRNA for effective protein translation, genome replication, and evasion of the host defense mechanisms. ...Many viruses encode RNA-modifying enzymes to edit the 5' end of viral RNA to mimic the cellular mRNA for effective protein translation, genome replication, and evasion of the host defense mechanisms. Alphavirus nsP1 synthesizes the 5' end Cap-0 structure of viral RNAs. However, the molecular basis of the capping process remains unclear. We determine high-resolution cryoelectron microscopy (cryo-EM) structures of Chikungunya virus nsP1 in complex with m7GTP/SAH, covalently attached m7GMP, and Cap-0 viral RNA. These structures reveal details of viral-RNA-capping reactions and uncover a sequence-specific virus RNA-recognition pattern that, in turn, regulates viral-RNA-capping efficiency to ensure optimal genome replication and subgenomic RNA transcription. This sequence-specific enzyme-RNA pairing is conserved across all alphaviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32914.map.gz emd_32914.map.gz | 398.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32914-v30.xml emd-32914-v30.xml emd-32914.xml emd-32914.xml | 10.8 KB 10.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32914.png emd_32914.png | 85.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32914 http://ftp.pdbj.org/pub/emdb/structures/EMD-32914 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32914 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32914 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32914.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32914.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Nonstructural Protein 1
| 全体 | 名称: Nonstructural Protein 1 |
|---|---|
| 要素 |
|
-超分子 #1: Nonstructural Protein 1
| 超分子 | 名称: Nonstructural Protein 1 / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Chikungunya virus strain S27-African prototype (チクングニア熱) Chikungunya virus strain S27-African prototype (チクングニア熱) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: mRNA-capping enzyme nsP1
| 分子 | 名称: mRNA-capping enzyme nsP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO EC番号:  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの |
|---|---|
| 由来(天然) | 生物種:   Chikungunya virus strain S27-African prototype (チクングニア熱) Chikungunya virus strain S27-African prototype (チクングニア熱) |
| 分子量 | 理論値: 62.056539 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDPVYVDIDA DSAFLKALQR AYPMFEVEPR QVTPNDHANA RAFSHLAIKL IEQEIDPDST ILDIGSAPAR RMMSDRKYHC VCPMRSAED PERLANYARK LASAAGKVLD RNISGKIGDL QAVMAVPDTE TPTFCLHTDV SCRQRADVAI YQDVYAVHAP T SLYHQAIK ...文字列: MDPVYVDIDA DSAFLKALQR AYPMFEVEPR QVTPNDHANA RAFSHLAIKL IEQEIDPDST ILDIGSAPAR RMMSDRKYHC VCPMRSAED PERLANYARK LASAAGKVLD RNISGKIGDL QAVMAVPDTE TPTFCLHTDV SCRQRADVAI YQDVYAVHAP T SLYHQAIK GVRLAYWVGF DTTPFMYNAM AGAYPSYSTN WADEQVLKAK NIGLCSTDLT EGRRGKLSIM RGKKLEPCDR VL FSVGSTL YPESRKLLKS WHLPSVFHLK GKLSFTCRCD TVVSCEGYVV KRITMSPGLY GKTTGYAVTH HADGFLMCKT TDT VDGERV SFSVCTYVPA TICDQMTGIL ATEVTPEDAQ KLLVGLNQRI VVNGRTQRNT NTMKNYMIPV VAQAFSKWAK ECRK DMEDE KLLGVRERTL TCCCLWAFKK QKTHTVYKRP DTQSIQKVQA EFDSFVVPSL WSSGLSIPLR TRIKWLLSKV PKTDL TPYS GDAQEARDAE KEAEEEREAE LTLEALPPLQ AAGGGGSWSH PQFEKMDYKD HDGDYKDHDI DYKDDDDK |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 12 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: (1R,2S,3S,4R,5R)-3-(6-aminopurin-9-yl)-4-fluoranyl-5-(2-hydroxyet...
| 分子 | 名称: (1R,2S,3S,4R,5R)-3-(6-aminopurin-9-yl)-4-fluoranyl-5-(2-hydroxyethyl)cyclopentane-1,2-diol タイプ: ligand / ID: 3 / コピー数: 12 / 式: 7XQ |
|---|---|
| 分子量 | 理論値: 297.286 Da |
| Chemical component information | 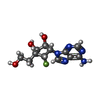 ChemComp-7XQ: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.62 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 76791 |
 ムービー
ムービー コントローラー
コントローラー














