+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 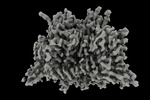 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
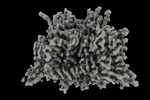
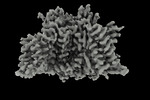
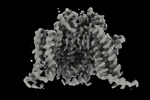
| タイトル | Apo human Nav1.8 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bundle of His cell action potential / AV node cell action potential /  clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization / clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization /  知覚 / 知覚 /  voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity / voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity /  voltage-gated sodium channel activity ...bundle of His cell action potential / AV node cell action potential / voltage-gated sodium channel activity ...bundle of His cell action potential / AV node cell action potential /  clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization / clathrin complex / membrane depolarization during action potential / regulation of atrial cardiac muscle cell membrane depolarization /  知覚 / 知覚 /  voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity / voltage-gated sodium channel complex / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / high voltage-gated calcium channel activity /  voltage-gated sodium channel activity / regulation of monoatomic ion transmembrane transport / Interaction between L1 and Ankyrins / voltage-gated sodium channel activity / regulation of monoatomic ion transmembrane transport / Interaction between L1 and Ankyrins /  voltage-gated calcium channel complex / Phase 0 - rapid depolarisation / odontogenesis of dentin-containing tooth / calcium ion import across plasma membrane / sodium ion transmembrane transport / regulation of cardiac muscle contraction / voltage-gated calcium channel complex / Phase 0 - rapid depolarisation / odontogenesis of dentin-containing tooth / calcium ion import across plasma membrane / sodium ion transmembrane transport / regulation of cardiac muscle contraction /  regulation of heart rate / regulation of heart rate /  presynaptic membrane / transmembrane transporter binding / presynaptic membrane / transmembrane transporter binding /  神経繊維 / glutamatergic synapse / extracellular exosome / 神経繊維 / glutamatergic synapse / extracellular exosome /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Yan N / Pan XJ / Huang XS / Huang GX | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structural basis for high-voltage activation and subtype-specific inhibition of human Na1.8. 著者: Xiaoshuang Huang / Xueqin Jin / Gaoxingyu Huang / Jian Huang / Tong Wu / Zhangqiang Li / Jiaofeng Chen / Fang Kong / Xiaojing Pan / Nieng Yan /  要旨: The dorsal root ganglia-localized voltage-gated sodium (Na) channel Na1.8 represents a promising target for developing next-generation analgesics. A prominent characteristic of Na1.8 is the ...The dorsal root ganglia-localized voltage-gated sodium (Na) channel Na1.8 represents a promising target for developing next-generation analgesics. A prominent characteristic of Na1.8 is the requirement of more depolarized membrane potential for activation. Here we present the cryogenic electron microscopy structures of human Na1.8 alone and bound to a selective pore blocker, A-803467, at overall resolutions of 2.7 to 3.2 Å. The first voltage-sensing domain (VSD) displays three different conformations. Structure-guided mutagenesis identified the extracellular interface between VSD and the pore domain (PD) to be a determinant for the high-voltage dependence of activation. A-803467 was clearly resolved in the central cavity of the PD, clenching S6. Our structure-guided functional characterizations show that two nonligand binding residues, Thr397 on S6 and Gly1406 on S6, allosterically modulate the channel's sensitivity to A-803467. Comparison of available structures of human Na channels suggests the extracellular loop region to be a potential site for developing subtype-specific pore-blocking biologics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32476.map.gz emd_32476.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32476-v30.xml emd-32476-v30.xml emd-32476.xml emd-32476.xml | 12.9 KB 12.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32476.png emd_32476.png | 87.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32476 http://ftp.pdbj.org/pub/emdb/structures/EMD-32476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32476 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32476 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32476.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32476.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.0825 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : sodium channel II
| 全体 | 名称: sodium channel II ナトリウムチャネル ナトリウムチャネル |
|---|---|
| 要素 |
|
-超分子 #1: sodium channel II
| 超分子 | 名称: sodium channel II / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Sodium channel protein type 10 subunit alpha
| 分子 | 名称: Sodium channel protein type 10 subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 220.9035 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEFPIGSLET NNFRRFTPES LVEIEKQIAA KQGTKKAREK HREQKDQEEK PRPQLDLKAC NQLPKFYGEL PAELIGEPLE DLDPFYSTH RTFMVLNKGR TISRFSATRA LWLFSPFNLI RRTAIKVSVH SWFSLFITVT ILVNCVCMTR TDLPEKIEYV F TVIYTFEA ...文字列: MEFPIGSLET NNFRRFTPES LVEIEKQIAA KQGTKKAREK HREQKDQEEK PRPQLDLKAC NQLPKFYGEL PAELIGEPLE DLDPFYSTH RTFMVLNKGR TISRFSATRA LWLFSPFNLI RRTAIKVSVH SWFSLFITVT ILVNCVCMTR TDLPEKIEYV F TVIYTFEA LIKILARGFC LNEFTYLRDP WNWLDFSVIT LAYVGTAIDL RGISGLRTFR VLRALKTVSV IPGLKVIVGA LI HSVKKLA DVTILTIFCL SVFALVGLQL FKGNLKNKCV KNDMAVNETT NYSSHRKPDI YINKRGTSDP LLCGNGSDSG HCP DGYICL KTSDNPDFNY TSFDSFAWAF LSLFRLMTQD SWERLYQQTL RTSGKIYMIF FVLVIFLGSF YLVNLILAVV TMAY EEQNQ ATTDEIEAKE KKFQEALEML RKEQEVLAAL GIDTTSLHSH NGSPLTSKNA SERRHRIKPR VSEGSTEDNK SPRSD PYNQ RRMSFLGLAS GKRRASHGSV FHFRSPGRDI SLPEGVTDDG VFPGDHESHR GSLLLGGGAG QQGPLPRSPL PQPSNP DSR HGEDEHQPPP TSELAPGAVD VSAFDAGQKK TFLSAEYLDE PFRAQRAMSV VSIITSVLEE LEESEQKCPP CLTSLSQ KY LIWDCCPMWV KLKTILFGLV TDPFAELTIT LCIVVNTIFM AMEHHGMSPT FEAMLQIGNI VFTIFFTAEM VFKIIAFD P YYYFQKKWNI FDCIIVTVSL LELGVAKKGS LSVLRSFRLL RVFKLAKSWP TLNTLIKIIG NSVGALGNLT IILAIIVFV FALVGKQLLG ENYRNNRKNI SAPHEDWPRW HMHDFFHSFL IVFRILCGEW IENMWACMEV GQKSICLILF LTVMVLGNLV VLNLFIALL LNSFFADNLT APEDDGEVNN LQVALARIQV FGHRTKQALC SFFSRSCPFP QPKAEPELVV KLPLSSSKAE N HIAANTAR GSSGGLQAPR GPRDEHSDFI ANPTVWVSVP IAEGESDLDD LEDDGGEDAQ SFQQEVIPKG QQEQLQQVER CG DHLTPRS PGTGTSSEDL APSLGETWKD ESVPQVPAEG VDDTSSSEGS TVDCLDPEEI LRKIPELADD LEEPDDCFTE GCI RHCPCC KLDTTKSPWD VGWQVRKTCY RIVEHSWFES FIIFMILLSS GSLAFEDYYL DQKPTVKALL EYTDRVFTFI FVFE MLLKW VAYGFKKYFT NAWCWLDFLI VNISLISLTA KILEYSEVAP IKALRTLRAL RPLRALSRFE GMRVVVDALV GAIPS IMNV LLVCLIFWLI FSIMGVNLFA GKFWRCINYT DGEFSLVPLS IVNNKSDCKI QNSTGSFFWV NVKVNFDNVA MGYLAL LQV ATFKGWMDIM YAAVDSREVN MQPKWEDNVY MYLYFVIFII FGGFFTLNLF VGVIIDNFNQ QKKKLGGQDI FMTEEQK KY YNAMKKLGSK KPQKPIPRPL NKFQGFVFDI VTRQAFDITI MVLICLNMIT MMVETDDQSE EKTKILGKIN QFFVAVFT G ECVMKMFALR QYYFTNGWNV FDFIVVVLSI ASLIFSAILK SLQSYFSPTL FRVIRLARIG RILRLIRAAK GIRTLLFAL MMSLPALFNI GLLLFLVMFI YSIFGMSSFP HVRWEAGIDD MFNFQTFANS MLCLFQITTS AGWDGLLSPI LNTGPPYCDP NLPNSNGTR GDCGSPAVGI IFFTTYIIIS FLIMVNMYIA VILENFNVAT EESTEPLSED DFDMFYETWE KFDPEATQFI T FSALSDFA DTLSGPLRIP KPNRNILIQM DLPLVPGDKI HCLDILFAFT KNVLGESGEL DSLKANMEEK FMATNLSKSS YE PIATTLR WKQEDISATV IQKAYRSYVL HRSMALSNTP CVPRAEEEAA SLPDEGFVAF TANENCVLPD KSETASATSF PPS YESVTR GLSDRVNMRT SSSIQNEDEA TSMELIAPGP |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 4 / コピー数: 4 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #5: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 5 / コピー数: 3 / 式: PCW |
|---|---|
| 分子量 | 理論値: 787.121 Da |
| Chemical component information |  ChemComp-PCW: |
-分子 #6: 1-O-OCTADECYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1-O-OCTADECYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 6 / コピー数: 8 / 式: LPE |
|---|---|
| 分子量 | 理論値: 510.708 Da |
| Chemical component information |  ChemComp-LPE: |
-分子 #7: O-[(R)-{[(2R)-2,3-bis(octadecanoyloxy)propyl]oxy}(hydroxy)phospho...
| 分子 | 名称: O-[(R)-{[(2R)-2,3-bis(octadecanoyloxy)propyl]oxy}(hydroxy)phosphoryl]-L-serine タイプ: ligand / ID: 7 / コピー数: 2 / 式: P5S |
|---|---|
| 分子量 | 理論値: 792.075 Da |
| Chemical component information |  ChemComp-P5S: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 410478 |
 ムービー
ムービー コントローラー
コントローラー