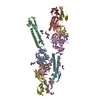+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3136 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A Single Alpha Helix Drives Extensive Remodeling of the Proteasome Lid and Completion of Regulatory Particle Assembly | |||||||||
 マップデータ マップデータ | Reconstruction of LP2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  proteasome (プロテアソーム) / proteasome (プロテアソーム) /  ubiquitin (ユビキチン) / ubiquitin (ユビキチン) /  proteolysis (タンパク質分解) / proteolysis (タンパク質分解) /  electron microscopy (電子顕微鏡) / electron microscopy (電子顕微鏡) /  mass spectrometry (質量分析法) mass spectrometry (質量分析法) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報protein deneddylation => GO:0000338 / molecular_function / SAGA complex localization to transcription regulatory region / peroxisome fission / proteasome storage granule assembly / transcription export complex 2 / protein deneddylation / maintenance of DNA trinucleotide repeats / filamentous growth /  COP9 signalosome ...protein deneddylation => GO:0000338 / molecular_function / SAGA complex localization to transcription regulatory region / peroxisome fission / proteasome storage granule assembly / transcription export complex 2 / protein deneddylation / maintenance of DNA trinucleotide repeats / filamentous growth / COP9 signalosome ...protein deneddylation => GO:0000338 / molecular_function / SAGA complex localization to transcription regulatory region / peroxisome fission / proteasome storage granule assembly / transcription export complex 2 / protein deneddylation / maintenance of DNA trinucleotide repeats / filamentous growth /  COP9 signalosome / proteasome regulatory particle / proteasome regulatory particle, lid subcomplex / COP9 signalosome / proteasome regulatory particle / proteasome regulatory particle, lid subcomplex /  mitochondrial fission / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / Ub-specific processing proteases / mitochondrial fission / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / Ub-specific processing proteases /  proteasome binding / regulation of protein catabolic process / protein deubiquitination / proteasome storage granule / proteasome binding / regulation of protein catabolic process / protein deubiquitination / proteasome storage granule /  proteasome assembly / enzyme regulator activity / mRNA export from nucleus / protein folding chaperone / Neutrophil degranulation / proteasome assembly / enzyme regulator activity / mRNA export from nucleus / protein folding chaperone / Neutrophil degranulation /  proteasome complex / double-strand break repair via homologous recombination / proteasome complex / double-strand break repair via homologous recombination /  metallopeptidase activity / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process / metallopeptidase activity / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process /  ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / molecular adaptor activity / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / molecular adaptor activity /  regulation of cell cycle / structural molecule activity / positive regulation of transcription by RNA polymerase II / regulation of cell cycle / structural molecule activity / positive regulation of transcription by RNA polymerase II /  ミトコンドリア / ミトコンドリア /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 16.0 Å ネガティブ染色法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Tomko Jr RJ / Taylor DW / Chen ZA / Wang HW / Rappsilber J / Hochstrasser M | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2015 ジャーナル: Cell / 年: 2015タイトル: A Single α Helix Drives Extensive Remodeling of the Proteasome Lid and Completion of Regulatory Particle Assembly. 著者: Robert J Tomko / David W Taylor / Zhuo A Chen / Hong-Wei Wang / Juri Rappsilber / Mark Hochstrasser /     要旨: Most short-lived eukaryotic proteins are degraded by the proteasome. A proteolytic core particle (CP) capped by regulatory particles (RPs) constitutes the 26S proteasome complex. RP biogenesis ...Most short-lived eukaryotic proteins are degraded by the proteasome. A proteolytic core particle (CP) capped by regulatory particles (RPs) constitutes the 26S proteasome complex. RP biogenesis culminates with the joining of two large subcomplexes, the lid and base. In yeast and mammals, the lid appears to assemble completely before attaching to the base, but how this hierarchical assembly is enforced has remained unclear. Using biochemical reconstitutions, quantitative cross-linking/mass spectrometry, and electron microscopy, we resolve the mechanistic basis for the linkage between lid biogenesis and lid-base joining. Assimilation of the final lid subunit, Rpn12, triggers a large-scale conformational remodeling of the nascent lid that drives RP assembly, in part by relieving steric clash with the base. Surprisingly, this remodeling is triggered by a single Rpn12 α helix. Such assembly-coupled conformational switching is reminiscent of viral particle maturation and may represent a commonly used mechanism to enforce hierarchical assembly in multisubunit complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3136.map.gz emd_3136.map.gz | 14.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3136-v30.xml emd-3136-v30.xml emd-3136.xml emd-3136.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3136.png emd_3136.png | 149.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3136 http://ftp.pdbj.org/pub/emdb/structures/EMD-3136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3136 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3136 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3136.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3136.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of LP2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Recombinant LP2 (Lid Particle 2)
| 全体 | 名称: Recombinant LP2 (Lid Particle 2) |
|---|---|
| 要素 |
|
-超分子 #1000: Recombinant LP2 (Lid Particle 2)
| 超分子 | 名称: Recombinant LP2 (Lid Particle 2) / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse. 集合状態: 1 Rpn3: 1 Rpn5: 1 Rpn6: 1 Rpn7: 1 Rpn8: 1 Rpn9: 1 Rpn11: 1 Sem1 Number unique components: 8 |
|---|---|
| 分子量 | 理論値: 343 KDa |
-分子 #1: 26S proteasome regulatory subunit RPN3
| 分子 | 名称: 26S proteasome regulatory subunit RPN3 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RPN3 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 64 KDa |
| 配列 | UniProtKB: 26S proteasome regulatory subunit RPN3 GO: ubiquitin-dependent protein catabolic process, regulation of protein catabolic process, molecular_function,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro: 26S proteasome regulatory subunit, C-terminal,  Proteasome component (PCI) domain Proteasome component (PCI) domain |
-分子 #2: 26S proteasome regulatory subunit RPN5
| 分子 | 名称: 26S proteasome regulatory subunit RPN5 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: RPN5 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 52 KDa |
| 配列 | UniProtKB: 26S proteasome regulatory subunit RPN5 GO: ubiquitin-dependent protein catabolic process, protein deneddylation => GO:0000338, molecular_function,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro:  Proteasome component (PCI) domain, ArsR-like helix-turn-helix domain Proteasome component (PCI) domain, ArsR-like helix-turn-helix domain |
-分子 #3: 26S proteasome regulatory subunit RPN6
| 分子 | 名称: 26S proteasome regulatory subunit RPN6 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: RPN6 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 50 KDa |
| 配列 | UniProtKB: 26S proteasome regulatory subunit RPN6 GO: ubiquitin-dependent protein catabolic process, structural molecule activity,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro:  Proteasome component (PCI) domain, Proteasome component (PCI) domain,  INTERPRO: IPR013143 INTERPRO: IPR013143 |
-分子 #4: 26S proteasome regulatory subunit RPN7
| 分子 | 名称: 26S proteasome regulatory subunit RPN7 / タイプ: protein_or_peptide / ID: 4 / Name.synonym: RPN7 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 49 KDa |
| 配列 | UniProtKB: 26S proteasome regulatory subunit RPN7 GO: ubiquitin-dependent protein catabolic process, structural molecule activity,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro: 26S proteasome regulatory subunit Rpn7/COP9 signalosome complex subunit 1,  Proteasome component (PCI) domain, Proteasome component (PCI) domain,  INTERPRO: IPR013026 INTERPRO: IPR013026 |
-分子 #5: 26S proteasome regulatory subunit RPN8
| 分子 | 名称: 26S proteasome regulatory subunit RPN8 / タイプ: protein_or_peptide / ID: 5 / Name.synonym: RPN8 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 38 KDa |
| 配列 | UniProtKB: 26S proteasome regulatory subunit RPN8 GO: ubiquitin-dependent protein catabolic process, molecular_function,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro: JAB1/MPN/MOV34 metalloenzyme domain, Rpn11/EIF3F, C-terminal |
-分子 #6: 26S proteasome regulatory subunit RPN9
| 分子 | 名称: 26S proteasome regulatory subunit RPN9 / タイプ: protein_or_peptide / ID: 6 / Name.synonym: RPN9 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 46 KDa |
| 配列 | UniProtKB: 26S proteasome regulatory subunit RPN9 GO: ubiquitin-dependent protein catabolic process, structural molecule activity,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro:  Proteasome component (PCI) domain, ArsR-like helix-turn-helix domain Proteasome component (PCI) domain, ArsR-like helix-turn-helix domain |
-分子 #7: 26S proteasome regulatory subunit RPN11
| 分子 | 名称: 26S proteasome regulatory subunit RPN11 / タイプ: protein_or_peptide / ID: 7 / Name.synonym: RPN11 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 34 KDa |
| 配列 | UniProtKB: Ubiquitin carboxyl-terminal hydrolase RPN11 GO: protein deubiquitination,  mitochondrial fission, cysteine-type deubiquitinase activity, mitochondrial fission, cysteine-type deubiquitinase activity,  proteasome complex, proteasome regulatory particle, lid subcomplex proteasome complex, proteasome regulatory particle, lid subcomplexInterPro: JAB1/MPN/MOV34 metalloenzyme domain, Rpn11/EIF3F, C-terminal |
-分子 #8: 26S proteasome regulatory subunit Sem1
| 分子 | 名称: 26S proteasome regulatory subunit Sem1 / タイプ: protein_or_peptide / ID: 8 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Yeast Saccharomyces cerevisiae (パン酵母) / 別称: Yeast |
| 分子量 | 理論値: 10 KDa |
| 配列 | UniProtKB:  26S proteasome complex subunit SEM1 26S proteasome complex subunit SEM1GO:  proteasome complex, proteasome regulatory particle, lid subcomplex, proteasome-mediated ubiquitin-dependent protein catabolic process, proteasome complex, proteasome regulatory particle, lid subcomplex, proteasome-mediated ubiquitin-dependent protein catabolic process,  proteasome assembly proteasome assemblyInterPro: DSS1/SEM1 |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.04 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM Tris, 150 mM NaCl, 5 mM MgCl2, and 10% glycerol |
| 染色 | タイプ: NEGATIVE 詳細: Sample was negatively stained with six consecutive droplets of 2% uranyl acetate. |
| グリッド | 詳細: 400 mesh Cu grid with thin carbon, glow discharged with a sputter coater. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 1.4 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 80000 Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 1.4 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 詳細 | Data acquired using Leginon. |
| 日付 | 2013年4月26日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 600 / 平均電子線量: 30 e/Å2 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, SPARX / 使用した粒子像数: 60000 |
 ムービー
ムービー コントローラー
コントローラー