+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-30398 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of S.typhimurium flagellar LP ring | |||||||||
 マップデータ マップデータ | CryoEM structure of S.typhimurium flagellar LP ring | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報bacterial-type flagellum basal body, distal rod, L ring / bacterial-type flagellum basal body, distal rod, P ring / cytoskeletal motor activity / bacterial-type flagellum-dependent cell motility / cell outer membrane / outer membrane-bounded periplasmic space / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |   Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) / Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) /   Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌) | |||||||||
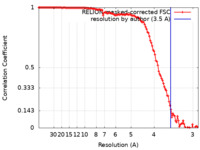
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Yamaguchi T / Makino F / Miyata T / Minamino T / Kato T / Namba K | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure of the molecular bushing of the bacterial flagellar motor. 著者: Tomoko Yamaguchi / Fumiaki Makino / Tomoko Miyata / Tohru Minamino / Takayuki Kato / Keiichi Namba /  要旨: The basal body of the bacterial flagellum is a rotary motor that consists of several rings (C, MS and LP) and a rod. The LP ring acts as a bushing supporting the distal rod for its rapid and stable ...The basal body of the bacterial flagellum is a rotary motor that consists of several rings (C, MS and LP) and a rod. The LP ring acts as a bushing supporting the distal rod for its rapid and stable rotation without much friction. Here, we use electron cryomicroscopy to describe the LP ring structure around the rod, at 3.5 Å resolution, from Salmonella Typhimurium. The structure shows 26-fold rotational symmetry and intricate intersubunit interactions of each subunit with up to six partners, which explains the structural stability. The inner surface is charged both positively and negatively. Positive charges on the P ring (the part of the LP ring that is embedded within the peptidoglycan layer) presumably play important roles in its initial assembly around the rod with a negatively charged surface. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_30398.map.gz emd_30398.map.gz | 7.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-30398-v30.xml emd-30398-v30.xml emd-30398.xml emd-30398.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_30398_fsc.xml emd_30398_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_30398.png emd_30398.png | 187.6 KB | ||
| マスクデータ |  emd_30398_msk_1.map emd_30398_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-30398 http://ftp.pdbj.org/pub/emdb/structures/EMD-30398 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30398 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-30398 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_30398.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_30398.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM structure of S.typhimurium flagellar LP ring | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.45 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_30398_msk_1.map emd_30398_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LP ring
| 全体 | 名称: LP ring |
|---|---|
| 要素 |
|
-超分子 #1: LP ring
| 超分子 | 名称: LP ring / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Bushing of flagellar motor |
|---|---|
| 由来(天然) | 生物種:   Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌)株: HK1002 |
-分子 #1: Flagellar P-ring protein
| 分子 | 名称: Flagellar P-ring protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 26 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌) |
| 分子量 | 理論値: 38.194176 KDa |
| 配列 | 文字列: MFKALAGIVL ALVATLAHAE RIRDLTSVQG VRENSLIGYG LVVGLDGTGD QTTQTPFTTQ TLNNMLSQLG ITVPTGTNMQ LKNVAAVMV TASYPPFARQ GQTIDVVVSS MGNAKSLRGG TLLMTPLKGV DSQVYALAQG NILVGGAGAS AGGSSVQVNQ L NGGRITNG ...文字列: MFKALAGIVL ALVATLAHAE RIRDLTSVQG VRENSLIGYG LVVGLDGTGD QTTQTPFTTQ TLNNMLSQLG ITVPTGTNMQ LKNVAAVMV TASYPPFARQ GQTIDVVVSS MGNAKSLRGG TLLMTPLKGV DSQVYALAQG NILVGGAGAS AGGSSVQVNQ L NGGRITNG AIIERELPTQ FGAGNTINLQ LNDEDFTMAQ QITDAINRAR GYGSATALDA RTVQVRVPSG NSSQVRFLAD IQ NMEVNVT PQDAKVVINS RTGSVVMNRE VTLDSCAVAQ GNLSVTVNRQ LNVNQPNTPF GGGQTVVTPQ TQIDLRQSGG SLQ SVRSSA NLNSVVRALN ALGATPMDLM SILQSMQSAG CLRAKLEII |
-分子 #2: Flagellar L-ring protein
| 分子 | 名称: Flagellar L-ring protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 26 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Salmonella typhimurium (サルモネラ菌) Salmonella typhimurium (サルモネラ菌) |
| 分子量 | 理論値: 24.726666 KDa |
| 配列 | 文字列: MQKYALHAYP VMALMVATLT GCAWIPAKPL VQGATTAQPI PGPVPVANGS IFQSAQPINY GYQPLFEDRR PRNIGDTLTI VLQENVSAS KSSSANASRD GKTSFGFDTV PRYLQGLFGN SRADMEASGG NSFNGKGGAN ASNTFSGTLT VTVDQVLANG N LHVVGEKQ ...文字列: MQKYALHAYP VMALMVATLT GCAWIPAKPL VQGATTAQPI PGPVPVANGS IFQSAQPINY GYQPLFEDRR PRNIGDTLTI VLQENVSAS KSSSANASRD GKTSFGFDTV PRYLQGLFGN SRADMEASGG NSFNGKGGAN ASNTFSGTLT VTVDQVLANG N LHVVGEKQ IAINQGTEFI RFSGVVNPRT ISGSNSVPST QVADARIEYV GNGYINEAQN MGWLQRFFLN LSPM |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 糖包埋 | 材質: buffer | ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: MOLYBDENUM / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 200 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 8.23 µm / 最小 デフォーカス(補正後): 0.275 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 1.4 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 40000 Bright-field microscopy / Cs: 1.4 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 40000 |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter |
| 試料ステージ | 試料ホルダーモデル: JEOL CRYOSPECPORTER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-50 / 実像数: 12759 / 平均露光時間: 10.0 sec. / 平均電子線量: 45.0 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 38 |
|---|---|
| 得られたモデル |  PDB-7clr: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X