+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2917 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM structure of ribosome-SRP-FtsY complex in "closed" state | |||||||||
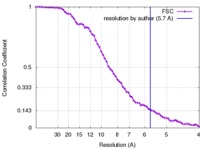
 マップデータ マップデータ | Reconstruction of ribosome-SRP-FtsY complex in the "closed" state. The map has been b-factor sharpened. The authors report resolution of 5.7 angstrom according to FSC=0.143, which was obtained from RELION post-process (for b-factor sharpening). Since the RELION post-process does not generate new half maps, the maps uploaded for FSC validation were the half maps before b-factor sharpening. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  protein targeting / protein targeting /  signal recognition particle (シグナル認識粒子) / signal recognition particle (シグナル認識粒子) /  signal sequence / signal sequence /  ribosome (リボソーム) ribosome (リボソーム) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 シグナル認識粒子 / シグナル認識粒子 /  signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / negative regulation of cytoplasmic translational initiation / signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / negative regulation of cytoplasmic translational initiation /  stringent response / stringent response /  transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity ... transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity ... シグナル認識粒子 / シグナル認識粒子 /  signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / negative regulation of cytoplasmic translational initiation / signal-recognition-particle GTPase / 7S RNA binding / SRP-dependent cotranslational protein targeting to membrane / protein targeting to membrane / negative regulation of cytoplasmic translational initiation /  stringent response / stringent response /  transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis / negative regulation of cytoplasmic translation / translational termination / DnaA-L2 complex / translation repressor activity / translational initiation / negative regulation of DNA-templated DNA replication initiation / transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis / negative regulation of cytoplasmic translation / translational termination / DnaA-L2 complex / translation repressor activity / translational initiation / negative regulation of DNA-templated DNA replication initiation /  ribosome assembly / mRNA regulatory element binding translation repressor activity / assembly of large subunit precursor of preribosome / response to reactive oxygen species / cytosolic ribosome assembly / ribosome assembly / mRNA regulatory element binding translation repressor activity / assembly of large subunit precursor of preribosome / response to reactive oxygen species / cytosolic ribosome assembly /  regulation of cell growth / DNA-templated transcription termination / response to radiation / mRNA 5'-UTR binding / regulation of cell growth / DNA-templated transcription termination / response to radiation / mRNA 5'-UTR binding /  ribosomal large subunit assembly / large ribosomal subunit rRNA binding / ribosomal large subunit assembly / large ribosomal subunit rRNA binding /  ribosome binding / large ribosomal subunit / cytoplasmic translation / ribosome binding / large ribosomal subunit / cytoplasmic translation /  5S rRNA binding / cytosolic large ribosomal subunit / 5S rRNA binding / cytosolic large ribosomal subunit /  transferase activity / transferase activity /  tRNA binding / negative regulation of translation / tRNA binding / negative regulation of translation /  rRNA binding / rRNA binding /  リボソーム / structural constituent of ribosome / リボソーム / structural constituent of ribosome /  翻訳 (生物学) / 翻訳 (生物学) /  ribonucleoprotein complex / response to antibiotic / ribonucleoprotein complex / response to antibiotic /  mRNA binding / mRNA binding /  GTPase activity / negative regulation of DNA-templated transcription / GTP binding / GTPase activity / negative regulation of DNA-templated transcription / GTP binding /  ATP hydrolysis activity / ATP hydrolysis activity /  DNA binding / DNA binding /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.7 Å クライオ電子顕微鏡法 / 解像度: 5.7 Å | |||||||||
 データ登録者 データ登録者 | von Loeffelholz O / Jiang Q / Ariosa A / Karuppasamy M / Huard K / Berger I / Shan S / Schaffitzel C | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2015 ジャーナル: Proc Natl Acad Sci U S A / 年: 2015タイトル: Ribosome-SRP-FtsY cotranslational targeting complex in the closed state. 著者: Ottilie von Loeffelholz / Qiyang Jiang / Aileen Ariosa / Manikandan Karuppasamy / Karine Huard / Imre Berger / Shu-ou Shan / Christiane Schaffitzel /    要旨: The signal recognition particle (SRP)-dependent pathway is essential for correct targeting of proteins to the membrane and subsequent insertion in the membrane or secretion. In Escherichia coli, the ...The signal recognition particle (SRP)-dependent pathway is essential for correct targeting of proteins to the membrane and subsequent insertion in the membrane or secretion. In Escherichia coli, the SRP and its receptor FtsY bind to ribosome-nascent chain complexes with signal sequences and undergo a series of distinct conformational changes, which ensures accurate timing and fidelity of protein targeting. Initial recruitment of the SRP receptor FtsY to the SRP-RNC complex results in GTP-independent binding of the SRP-FtsY GTPases at the SRP RNA tetraloop. In the presence of GTP, a closed state is adopted by the SRP-FtsY complex. The cryo-EM structure of the closed state reveals an ordered SRP RNA and SRP M domain with a signal sequence-bound. Van der Waals interactions between the finger loop and ribosomal protein L24 lead to a constricted signal sequence-binding pocket possibly preventing premature release of the signal sequence. Conserved M-domain residues contact ribosomal RNA helices 24 and 59. The SRP-FtsY GTPases are detached from the RNA tetraloop and flexible, thus liberating the ribosomal exit site for binding of the translocation machinery. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2917.map.gz emd_2917.map.gz | 20.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2917-v30.xml emd-2917-v30.xml emd-2917.xml emd-2917.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2917_fsc.xml emd_2917_fsc.xml | 7.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_2917.png emd_2917.png | 257.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2917 http://ftp.pdbj.org/pub/emdb/structures/EMD-2917 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2917 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2917 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2917.map.gz / 形式: CCP4 / 大きさ: 21.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2917.map.gz / 形式: CCP4 / 大きさ: 21.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of ribosome-SRP-FtsY complex in the "closed" state. The map has been b-factor sharpened. The authors report resolution of 5.7 angstrom according to FSC=0.143, which was obtained from RELION post-process (for b-factor sharpening). Since the RELION post-process does not generate new half maps, the maps uploaded for FSC validation were the half maps before b-factor sharpening. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli SRP-FtsY binds to E. coli ribosome with Lep50 nascent chain
| 全体 | 名称: E. coli SRP-FtsY binds to E. coli ribosome with Lep50 nascent chain |
|---|---|
| 要素 |
|
-超分子 #1000: E. coli SRP-FtsY binds to E. coli ribosome with Lep50 nascent chain
| 超分子 | 名称: E. coli SRP-FtsY binds to E. coli ribosome with Lep50 nascent chain タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 2.7 MDa |
-超分子 #1: E. coli 70S ribosome displaying Lep50 nascent chain
| 超分子 | 名称: E. coli 70S ribosome displaying Lep50 nascent chain / タイプ: complex / ID: 1 / Name.synonym: RNC 詳細: Ribosome-nascent chain complexes (RNCs) were prepared by in vitro transcription and translation from pUC19StrepLep50 and purified by sucrose gradient centrifugation and affinity chromatography. 組換発現: No / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 2.5 MDa |
-分子 #1: Signal recongnition particle and SRP receptor
| 分子 | 名称: Signal recongnition particle and SRP receptor / タイプ: protein_or_peptide / ID: 1 / Name.synonym: scSRP219 詳細: This is a single-chain construct comprising E.coli FtsY219 (truncated version lacking the N-terminal A-domain and the first helix of the N-domain) fused via a 31-amino acid glycine-serine- ...詳細: This is a single-chain construct comprising E.coli FtsY219 (truncated version lacking the N-terminal A-domain and the first helix of the N-domain) fused via a 31-amino acid glycine-serine-rich linker to full-length Ffh. コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 150 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) / 組換プラスミド: pET24 and pUC19 Escherichia coli BL21 (大腸菌) / 組換プラスミド: pET24 and pUC19 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50mM Hepes-KOH, 100mM KOAc, 8mM Mg(OAc)2, 500ug/ml chloramphenicol |
| グリッド | 詳細: 300 mesh quantifoil grid type R1.2/1.3 coated with a thin continuous carbon layer, glow-discharged for 30 s |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 77 K / 装置: FEI VITROBOT MARK IV / 手法: Blot for 2 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 77769 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 日付 | 2014年1月24日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 2840 / 平均電子線量: 24 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID:  2aw4 |
|---|---|
| ソフトウェア | 名称: Chimera, Coot |
| 詳細 | The structures were fitted by initial rigid body fitting in Chimera, followed by local refinement in Coot. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-5aka: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: Chimera, Coot, HHpred, CNS |
| 詳細 | The structures were fitted by initial rigid body fitting in Chimera, followed by local refinement in Coot. The finger loop region was modelled in HHpred. The whole structure was finally energy minimization in CNS Version 1.0. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
| 得られたモデル |  PDB-5aka: |
 ムービー
ムービー コントローラー
コントローラー