登録情報 データベース : EMDB / ID : EMD-29069タイトル Cryo-EM structure of the GR-Hsp90-FKBP51 complex Sharpened consensus map for the GR-Hsp90-FKBP51 complex 複合体 : Complex of the Glucocorticoid Receptor ligand binding domain, Hsp90 alpha dimer, and the co-chaperone FKBP51タンパク質・ペプチド : Heat shock protein HSP 90-alphaタンパク質・ペプチド : Glucocorticoid receptorタンパク質・ペプチド : Peptidyl-prolyl cis-trans isomerase FKBP5リガンド : ADENOSINE-5'-TRIPHOSPHATEリガンド : MAGNESIUM IONリガンド : DEXAMETHASONE / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.23 Å Noddings CM / Agard DA 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) GM118099
ジャーナル : Nat Struct Mol Biol / 年 : 2023タイトル : Cryo-EM reveals how Hsp90 and FKBP immunophilins co-regulate the glucocorticoid receptor.著者 : Chari M Noddings / Jill L Johnson / David A Agard / 要旨 : Hsp90 is an essential molecular chaperone responsible for the folding and activation of hundreds of 'client' proteins, including the glucocorticoid receptor (GR). Previously, we revealed that Hsp70 ... Hsp90 is an essential molecular chaperone responsible for the folding and activation of hundreds of 'client' proteins, including the glucocorticoid receptor (GR). Previously, we revealed that Hsp70 and Hsp90 remodel the conformation of GR to regulate ligand binding, aided by co-chaperones. In vivo, the co-chaperones FKBP51 and FKBP52 antagonistically regulate GR activity, but a molecular understanding is lacking. Here we present a 3.01 Å cryogenic electron microscopy structure of the human GR:Hsp90:FKBP52 complex, revealing how FKBP52 integrates into the GR chaperone cycle and directly binds to the active client, potentiating GR activity in vitro and in vivo. We also present a 3.23 Å cryogenic electron microscopy structure of the human GR:Hsp90:FKBP51 complex, revealing how FKBP51 competes with FKBP52 for GR:Hsp90 binding and demonstrating how FKBP51 can act as a potent antagonist to FKBP52. Altogether, we demonstrate how FKBP51 and FKBP52 integrate into the GR chaperone cycle to advance GR to the next stage of maturation. 履歴 登録 2022年12月10日 - ヘッダ(付随情報) 公開 2023年11月1日 - マップ公開 2023年11月1日 - 更新 2023年12月20日 - 現状 2023年12月20日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード chaperone (シャペロン) /
chaperone (シャペロン) /  steroid hormone receptor /
steroid hormone receptor /  ligand binding (リガンド) /
ligand binding (リガンド) /  ATP binding (アデノシン三リン酸) /
ATP binding (アデノシン三リン酸) /  protein folding (フォールディング)
protein folding (フォールディング) 機能・相同性情報
機能・相同性情報 steroid hormone binding / PTK6 Expression / neuroinflammatory response / glucocorticoid metabolic process / microglia differentiation /
steroid hormone binding / PTK6 Expression / neuroinflammatory response / glucocorticoid metabolic process / microglia differentiation /  maternal behavior / mammary gland duct morphogenesis ...Regulation of NPAS4 gene transcription / regulation of glucocorticoid biosynthetic process / nuclear glucocorticoid receptor activity /
maternal behavior / mammary gland duct morphogenesis ...Regulation of NPAS4 gene transcription / regulation of glucocorticoid biosynthetic process / nuclear glucocorticoid receptor activity /  steroid hormone binding / PTK6 Expression / neuroinflammatory response / glucocorticoid metabolic process / microglia differentiation /
steroid hormone binding / PTK6 Expression / neuroinflammatory response / glucocorticoid metabolic process / microglia differentiation /  maternal behavior / mammary gland duct morphogenesis / nucleus localization / astrocyte differentiation / cellular response to glucocorticoid stimulus /
maternal behavior / mammary gland duct morphogenesis / nucleus localization / astrocyte differentiation / cellular response to glucocorticoid stimulus /  motor behavior / sperm mitochondrial sheath /
motor behavior / sperm mitochondrial sheath /  dATP binding / Scavenging by Class F Receptors /
dATP binding / Scavenging by Class F Receptors /  sulfonylurea receptor binding / CTP binding / positive regulation of protein polymerization / vRNP Assembly / UTP binding / sperm plasma membrane / positive regulation of tau-protein kinase activity / protein insertion into mitochondrial outer membrane /
sulfonylurea receptor binding / CTP binding / positive regulation of protein polymerization / vRNP Assembly / UTP binding / sperm plasma membrane / positive regulation of tau-protein kinase activity / protein insertion into mitochondrial outer membrane /  regulation of gluconeogenesis /
regulation of gluconeogenesis /  chaperone-mediated autophagy /
chaperone-mediated autophagy /  telomerase holoenzyme complex assembly / adrenal gland development / Rho GDP-dissociation inhibitor binding / cellular response to steroid hormone stimulus /
telomerase holoenzyme complex assembly / adrenal gland development / Rho GDP-dissociation inhibitor binding / cellular response to steroid hormone stimulus /  FK506 binding / Uptake and function of diphtheria toxin / mitochondrial transport / Drug-mediated inhibition of ERBB2 signaling / Resistance of ERBB2 KD mutants to trastuzumab / Resistance of ERBB2 KD mutants to sapitinib / Resistance of ERBB2 KD mutants to tesevatinib / Resistance of ERBB2 KD mutants to neratinib / Resistance of ERBB2 KD mutants to osimertinib / Resistance of ERBB2 KD mutants to afatinib / Resistance of ERBB2 KD mutants to AEE788 / Resistance of ERBB2 KD mutants to lapatinib /
FK506 binding / Uptake and function of diphtheria toxin / mitochondrial transport / Drug-mediated inhibition of ERBB2 signaling / Resistance of ERBB2 KD mutants to trastuzumab / Resistance of ERBB2 KD mutants to sapitinib / Resistance of ERBB2 KD mutants to tesevatinib / Resistance of ERBB2 KD mutants to neratinib / Resistance of ERBB2 KD mutants to osimertinib / Resistance of ERBB2 KD mutants to afatinib / Resistance of ERBB2 KD mutants to AEE788 / Resistance of ERBB2 KD mutants to lapatinib /  Drug resistance in ERBB2 TMD/JMD mutants / PIWI-interacting RNA (piRNA) biogenesis / TPR domain binding /
Drug resistance in ERBB2 TMD/JMD mutants / PIWI-interacting RNA (piRNA) biogenesis / TPR domain binding /  non-chaperonin molecular chaperone ATPase / regulation of postsynaptic membrane neurotransmitter receptor levels / dendritic growth cone / Sema3A PAK dependent Axon repulsion / skeletal muscle contraction / regulation of protein ubiquitination / positive regulation of cell size / protein unfolding / HSF1-dependent transactivation / telomere maintenance via telomerase / response to unfolded protein / HSF1 activation / chaperone-mediated protein complex assembly / regulation of protein-containing complex assembly / Attenuation phase /
non-chaperonin molecular chaperone ATPase / regulation of postsynaptic membrane neurotransmitter receptor levels / dendritic growth cone / Sema3A PAK dependent Axon repulsion / skeletal muscle contraction / regulation of protein ubiquitination / positive regulation of cell size / protein unfolding / HSF1-dependent transactivation / telomere maintenance via telomerase / response to unfolded protein / HSF1 activation / chaperone-mediated protein complex assembly / regulation of protein-containing complex assembly / Attenuation phase /  estrogen response element binding / FOXO-mediated transcription of oxidative stress, metabolic and neuronal genes / RHOBTB2 GTPase cycle / positive regulation of lamellipodium assembly / eNOS activation /
estrogen response element binding / FOXO-mediated transcription of oxidative stress, metabolic and neuronal genes / RHOBTB2 GTPase cycle / positive regulation of lamellipodium assembly / eNOS activation /  DNA polymerase binding / axonal growth cone / chaperone-mediated protein folding / intracellular steroid hormone receptor signaling pathway / MECP2 regulates neuronal receptors and channels / core promoter sequence-specific DNA binding / Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / Loss of Nlp from mitotic centrosomes / Loss of proteins required for interphase microtubule organization from the centrosome / positive regulation of cardiac muscle contraction / Signaling by ERBB2 / cardiac muscle cell apoptotic process / Recruitment of mitotic centrosome proteins and complexes / positive regulation of telomerase activity / response to salt stress / positive regulation of defense response to virus by host / cellular response to transforming growth factor beta stimulus / endocytic vesicle lumen / Recruitment of NuMA to mitotic centrosomes /
DNA polymerase binding / axonal growth cone / chaperone-mediated protein folding / intracellular steroid hormone receptor signaling pathway / MECP2 regulates neuronal receptors and channels / core promoter sequence-specific DNA binding / Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / Loss of Nlp from mitotic centrosomes / Loss of proteins required for interphase microtubule organization from the centrosome / positive regulation of cardiac muscle contraction / Signaling by ERBB2 / cardiac muscle cell apoptotic process / Recruitment of mitotic centrosome proteins and complexes / positive regulation of telomerase activity / response to salt stress / positive regulation of defense response to virus by host / cellular response to transforming growth factor beta stimulus / endocytic vesicle lumen / Recruitment of NuMA to mitotic centrosomes /  heat shock protein binding / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Anchoring of the basal body to the plasma membrane /
heat shock protein binding / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Anchoring of the basal body to the plasma membrane /  protein tyrosine kinase binding / TBP-class protein binding /
protein tyrosine kinase binding / TBP-class protein binding /  steroid binding / response to cold / activation of innate immune response / positive regulation of interferon-beta production / nitric-oxide synthase regulator activity / cellular response to dexamethasone stimulus / lysosomal lumen / Constitutive Signaling by Overexpressed ERBB2 / ESR-mediated signaling / AURKA Activation by TPX2
steroid binding / response to cold / activation of innate immune response / positive regulation of interferon-beta production / nitric-oxide synthase regulator activity / cellular response to dexamethasone stimulus / lysosomal lumen / Constitutive Signaling by Overexpressed ERBB2 / ESR-mediated signaling / AURKA Activation by TPX2
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 単粒子再構成法 /
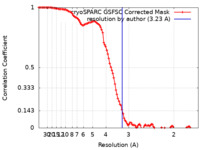
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.23 Å
クライオ電子顕微鏡法 / 解像度: 3.23 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Nat Struct Mol Biol / 年: 2023
ジャーナル: Nat Struct Mol Biol / 年: 2023
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_29069.map.gz
emd_29069.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-29069-v30.xml
emd-29069-v30.xml emd-29069.xml
emd-29069.xml EMDBヘッダ
EMDBヘッダ emd_29069_fsc.xml
emd_29069_fsc.xml FSCデータファイル
FSCデータファイル emd_29069.png
emd_29069.png emd_29069_msk_1.map
emd_29069_msk_1.map emd_29069_msk_2.map
emd_29069_msk_2.map emd_29069_msk_3.map
emd_29069_msk_3.map マスクマップ
マスクマップ emd-29069.cif.gz
emd-29069.cif.gz emd_29069_additional_1.map.gz
emd_29069_additional_1.map.gz emd_29069_additional_2.map.gz
emd_29069_additional_2.map.gz emd_29069_additional_3.map.gz
emd_29069_additional_3.map.gz emd_29069_additional_4.map.gz
emd_29069_additional_4.map.gz emd_29069_half_map_1.map.gz
emd_29069_half_map_1.map.gz emd_29069_half_map_2.map.gz
emd_29069_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-29069
http://ftp.pdbj.org/pub/emdb/structures/EMD-29069 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29069
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29069

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_29069.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_29069.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_29069_msk_1.map
emd_29069_msk_1.map emd_29069_msk_2.map
emd_29069_msk_2.map emd_29069_msk_3.map
emd_29069_msk_3.map 試料の構成要素
試料の構成要素
 Homo sapiens (ヒト)
Homo sapiens (ヒト) non-chaperonin molecular chaperone ATPase
non-chaperonin molecular chaperone ATPase
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli BL21(DE3) (大腸菌)
Escherichia coli BL21(DE3) (大腸菌) Heat shock protein HSP 90-alpha
Heat shock protein HSP 90-alpha
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli BL21(DE3) (大腸菌)
Escherichia coli BL21(DE3) (大腸菌) 糖質コルチコイド受容体
糖質コルチコイド受容体 プロリルイソメラーゼ
プロリルイソメラーゼ
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Escherichia coli BL21(DE3) (大腸菌)
Escherichia coli BL21(DE3) (大腸菌)

 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000
Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー






































 Z
Z Y
Y X
X