+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23005 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Maltose Binding Protein in complex with Hsp90 and co-chaperone p23 Maltose-binding protein Maltose-binding protein | |||||||||
 マップデータ マップデータ | Primary map ; MBP:Hsp90:p23 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lung saccule development /  prostaglandin-E synthase / prostaglandin-E synthase /  prostaglandin-E synthase activity / prostaglandin-E synthase activity /  telomerase activity / intracellular glucocorticoid receptor signaling pathway / Aryl hydrocarbon receptor signalling / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / cyclooxygenase pathway / telomerase activity / intracellular glucocorticoid receptor signaling pathway / Aryl hydrocarbon receptor signalling / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / cyclooxygenase pathway /  telomerase holoenzyme complex / protein folding chaperone complex ...lung saccule development / telomerase holoenzyme complex / protein folding chaperone complex ...lung saccule development /  prostaglandin-E synthase / prostaglandin-E synthase /  prostaglandin-E synthase activity / prostaglandin-E synthase activity /  telomerase activity / intracellular glucocorticoid receptor signaling pathway / Aryl hydrocarbon receptor signalling / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / cyclooxygenase pathway / telomerase activity / intracellular glucocorticoid receptor signaling pathway / Aryl hydrocarbon receptor signalling / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / cyclooxygenase pathway /  telomerase holoenzyme complex / protein folding chaperone complex / glycogen biosynthetic process / prostaglandin biosynthetic process / skin development / telomerase holoenzyme complex / protein folding chaperone complex / glycogen biosynthetic process / prostaglandin biosynthetic process / skin development /  telomerase holoenzyme complex assembly / chaperone cofactor-dependent protein refolding / telomere maintenance via telomerase / HSF1 activation / chaperone-mediated protein complex assembly / Attenuation phase / telomerase holoenzyme complex assembly / chaperone cofactor-dependent protein refolding / telomere maintenance via telomerase / HSF1 activation / chaperone-mediated protein complex assembly / Attenuation phase /  DNA polymerase binding / positive regulation of phosphorylation / positive regulation of telomerase activity / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / DNA polymerase binding / positive regulation of phosphorylation / positive regulation of telomerase activity / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand /  telomere maintenance / ESR-mediated signaling / telomere maintenance / ESR-mediated signaling /  Hsp90 protein binding / unfolded protein binding / Hsp90 protein binding / unfolded protein binding /  フォールディング / fibroblast proliferation / protein-folding chaperone binding / Estrogen-dependent gene expression / Potential therapeutics for SARS / フォールディング / fibroblast proliferation / protein-folding chaperone binding / Estrogen-dependent gene expression / Potential therapeutics for SARS /  chromosome, telomeric region / protein stabilization / chromosome, telomeric region / protein stabilization /  シグナル伝達 / protein-containing complex / シグナル伝達 / protein-containing complex /  核質 / 核質 /  細胞核 / 細胞核 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
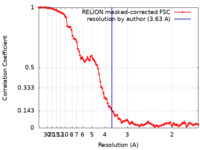
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.63 Å クライオ電子顕微鏡法 / 解像度: 3.63 Å | |||||||||
 データ登録者 データ登録者 | Noddings CM / Wang Y-R / Agard DA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structure of Hsp90-p23-GR reveals the Hsp90 client-remodelling mechanism. 著者: Chari M Noddings / Ray Yu-Ruei Wang / Jill L Johnson / David A Agard /  要旨: Hsp90 is a conserved and essential molecular chaperone responsible for the folding and activation of hundreds of 'client' proteins. The glucocorticoid receptor (GR) is a model client that constantly ...Hsp90 is a conserved and essential molecular chaperone responsible for the folding and activation of hundreds of 'client' proteins. The glucocorticoid receptor (GR) is a model client that constantly depends on Hsp90 for activity. GR ligand binding was previously shown to nr inhibited by Hsp70 and restored by Hsp90, aided by the co-chaperone p23. However, a molecular understanding of the chaperone-mediated remodelling that occurs between the inactive Hsp70-Hsp90 'client-loading complex' and an activated Hsp90-p23 'client-maturation complex' is lacking for any client, including GR. Here we present a cryo-electron microscopy (cryo-EM) structure of the human GR-maturation complex (GR-Hsp90-p23), revealing that the GR ligand-binding domain is restored to a folded, ligand-bound conformation, while being simultaneously threaded through the Hsp90 lumen. In addition, p23 directly stabilizes native GR using a C-terminal helix, resulting in enhanced ligand binding. This structure of a client bound to Hsp90 in a native conformation contrasts sharply with the unfolded kinase-Hsp90 structure. Thus, aided by direct co-chaperone-client interactions, Hsp90 can directly dictate client-specific folding outcomes. Together with the GR-loading complex structure, we present the molecular mechanism of chaperone-mediated GR remodelling, establishing the first, to our knowledge, complete chaperone cycle for any Hsp90 client. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23005.map.gz emd_23005.map.gz | 7.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23005-v30.xml emd-23005-v30.xml emd-23005.xml emd-23005.xml | 21.6 KB 21.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23005_fsc.xml emd_23005_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23005.png emd_23005.png | 60.2 KB | ||
| マスクデータ |  emd_23005_msk_1.map emd_23005_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23005_additional_1.map.gz emd_23005_additional_1.map.gz emd_23005_half_map_1.map.gz emd_23005_half_map_1.map.gz emd_23005_half_map_2.map.gz emd_23005_half_map_2.map.gz | 80.7 MB 80.9 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23005 http://ftp.pdbj.org/pub/emdb/structures/EMD-23005 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23005 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23005 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7krjC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-11028 (タイトル: Cryo-EM Structures of Glucocorticoid Receptor-Hsp90-p23 [the GR Maturation Complex], Hsp90-p23, and MBP-Hsp90-p23 EMPIAR-11028 (タイトル: Cryo-EM Structures of Glucocorticoid Receptor-Hsp90-p23 [the GR Maturation Complex], Hsp90-p23, and MBP-Hsp90-p23Data size: 494.1 Data #1: MotionCor2 aligned frames of GR-Hsp90-p23 collected on Gatan K3 [micrographs - single frame] Data #2: Processed subsets [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23005.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23005.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map ; MBP:Hsp90:p23 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23005_msk_1.map emd_23005_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Raw map ; MBP:Hsp90:p23
| ファイル | emd_23005_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map ; MBP:Hsp90:p23 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: MBP:Hsp90:p23 Half Map 1
| ファイル | emd_23005_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MBP:Hsp90:p23 Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: MBP:Hsp90:p23 Half Map 2
| ファイル | emd_23005_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MBP:Hsp90:p23 Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of the Maltose Binding Protein, Hsp90 dimer, and the co-c...
| 全体 | 名称: Complex of the Maltose Binding Protein, Hsp90 dimer, and the co-chaperone p23 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of the Maltose Binding Protein, Hsp90 dimer, and the co-c...
| 超分子 | 名称: Complex of the Maltose Binding Protein, Hsp90 dimer, and the co-chaperone p23 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 263 KDa |
-分子 #1: heat shock protein 90
| 分子 | 名称: heat shock protein 90 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MPEETQTQDQ PMEEEEVETF AFQAEIAQLM SLIINTFYSN KEIFLRELIS NSSDALDKIR YESLTDPSK LDSGKELHIN LIPNKQDRTL TIVDTGIGMT KADLINNLGT IAKSGTKAFM E ALQAGADI SMIGQFGVGF YSAYLVAEKV TVITKHNDDE QYAWESSAGG ...文字列: MPEETQTQDQ PMEEEEVETF AFQAEIAQLM SLIINTFYSN KEIFLRELIS NSSDALDKIR YESLTDPSK LDSGKELHIN LIPNKQDRTL TIVDTGIGMT KADLINNLGT IAKSGTKAFM E ALQAGADI SMIGQFGVGF YSAYLVAEKV TVITKHNDDE QYAWESSAGG SFTVRTDTGE PM GRGTKVI LHLKEDQTEY LEERRIKEIV KKHSQFIGYP ITLFVEKERD KEVSDDEAEE KED KEEEKE KEEKESEDKP EIEDVGSDEE EEKKDGDKKK KKKIKEKYID QEELNKTKPI WTRN PDDIT NEEYGEFYKS LTNDWEDHLA VKHFSVEGQL EFRALLFVPR RAPFDLFENR KKKNN IKLY VRRVFIMDNC EELIPEYLNF IRGVVDSEDL PLNISREMLQ QSKILKVIRK NLVKKC LEL FTELAEDKEN YKKFYEQFSK NIKLGIHEDS QNRKKLSELL RYYTSASGDE MVSLKDY CT RMKENQKHIY YITGETKDQV ANSAFVERLR KHGLEVIYMI EPIDEYCVQQ LKEFEGKT L VSVTKEGLEL PEDEEEKKKQ EEKKTKFENL CKIMKDILEK KVEKVVVSNR LVTSPCCIV TSTYGWTANM ERIMKAQALR DNSTMGYMAA KKHLEINPDH SIIETLRQKA EADKNDKSVK DLVILLYET ALLSSGFSLE DPQTHANRIY RMIKLGLGID EDDPTADDTS AAVTEEMPPL E GDDDTSRM EEVD |
-分子 #2: p23
| 分子 | 名称: p23 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MQPASAKWYD RRDYVFIEFC VEDSKDVNVN FEKSKLTFSC LGGSDNFKHL NEIDLFHCID PNDSKHKRTD RSILCCLRKG ESGQSWPRLT KERAKLNWLS VDFNNWKDWE DDSDEDMSNF DRFSEMMNNM GGDEDVDLPE VDGADDDSQD SDDEKMPDLE |
-分子 #3: maltose binding protein
| 分子 | 名称: maltose binding protein / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MKIEEGKLVI WINGDKGYNG LAEVGKKFEK DTGIKVTVEH PDKLEEKFPQ VAATGDGPDI IFWAHDRFGG YAQSGLLAEI TPDKAFQDKL YPFTWDAVRY NGKLIAYPIA VEALSLIYNK DLLPNPPKTW EEIPALDKEL KAKGKSALMF NLQEPYFTWP LIAADGGYAF ...文字列: MKIEEGKLVI WINGDKGYNG LAEVGKKFEK DTGIKVTVEH PDKLEEKFPQ VAATGDGPDI IFWAHDRFGG YAQSGLLAEI TPDKAFQDKL YPFTWDAVRY NGKLIAYPIA VEALSLIYNK DLLPNPPKTW EEIPALDKEL KAKGKSALMF NLQEPYFTWP LIAADGGYAF KYENGKYDIK DVGVDNAGAK AGLTFLVDLI KNKHMNADTD YSIAEAAFNK GETAMTINGP WAWSNIDTSK VNYGVTVLPT FKGQPSKPFV GVLSAGINAA SPNKELAKEF LENYLLTDEG LEAVNKDKPL GAVALKSYEE ELAKDPRIAA TMENAQKGEI MPNIPQMSAF WYAVRTAVIN AASGRQTVDE ALKDAQT |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 5608 / 平均露光時間: 5.9 sec. / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X