+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21391 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of porcine epidemic diarrhea virus (PEDV) spike protein | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / endocytosis involved in viral entry into host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated virion attachment to host cell / membrane => GO:0016020 / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion membrane エンベロープ (ウイルス) / virion membrane類似検索 - 分子機能 | |||||||||
| 生物種 |   Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Kirchdoerfer RN / Ward AB | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: Structure and immune recognition of the porcine epidemic diarrhea virus spike protein. 著者: Robert N Kirchdoerfer / Mahesh Bhandari / Olnita Martini / Leigh M Sewall / Sandhya Bangaru / Kyoung-Jin Yoon / Andrew B Ward /  要旨: Porcine epidemic diarrhea virus (PEDV) is an alphacoronavirus responsible for significant morbidity and mortality in pigs. A key determinant of viral tropism and entry, the PEDV spike protein is a ...Porcine epidemic diarrhea virus (PEDV) is an alphacoronavirus responsible for significant morbidity and mortality in pigs. A key determinant of viral tropism and entry, the PEDV spike protein is a key target for the host antibody response and a good candidate for a protein-based vaccine immunogen. We used electron microscopy to evaluate the PEDV spike structure, as well as pig polyclonal antibody responses to viral infection. The structure of the PEDV spike reveals a configuration similar to that of HuCoV-NL63. Several PEDV protein-protein interfaces are mediated by non-protein components, including a glycan at Asn264 and two bound palmitoleic acid molecules. The polyclonal antibody response to PEDV infection shows a dominance of epitopes in the S1 region. This structural and immune characterization provides insights into coronavirus spike stability determinants and explores the immune landscape of viral spike proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21391.map.gz emd_21391.map.gz | 78.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21391-v30.xml emd-21391-v30.xml emd-21391.xml emd-21391.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21391_fsc.xml emd_21391_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21391.png emd_21391.png | 182.9 KB | ||
| マスクデータ |  emd_21391_msk_1.map emd_21391_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_21391_half_map_1.map.gz emd_21391_half_map_1.map.gz emd_21391_half_map_2.map.gz emd_21391_half_map_2.map.gz | 65.4 MB 65.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21391 http://ftp.pdbj.org/pub/emdb/structures/EMD-21391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21391 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21391 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21391.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21391.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_21391_msk_1.map emd_21391_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: First half map
| ファイル | emd_21391_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Second half map
| ファイル | emd_21391_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Porcine epidemic diarrhea virus spike protein
| 全体 | 名称: Porcine epidemic diarrhea virus spike protein |
|---|---|
| 要素 |
|
-超分子 #1: Porcine epidemic diarrhea virus spike protein
| 超分子 | 名称: Porcine epidemic diarrhea virus spike protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Recombinantly expressed in insect cells using a baculovirus vector. |
|---|---|
| 由来(天然) | 生物種:   Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス)株: 13-019349 |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)組換株: Sf9 |
| 分子量 | 実験値: 600 KDa |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) |
| 分子量 | 理論値: 148.463656 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: LPQDVTRCSA NTNFRRFFSK FNVQAPAVVV LGGYLPIGEN QGVNSTWYCA GQHPTASGVH GIFVSHIRGG HGFEIGISQE PFDPSGYQL YLHKATNGNT NATARLRICQ FPSIKTLGPT ANNDVTTGRN CLFNKAIPAH MSEHSVVGIT WDNDRVTVFS D KIYYFYFK ...文字列: LPQDVTRCSA NTNFRRFFSK FNVQAPAVVV LGGYLPIGEN QGVNSTWYCA GQHPTASGVH GIFVSHIRGG HGFEIGISQE PFDPSGYQL YLHKATNGNT NATARLRICQ FPSIKTLGPT ANNDVTTGRN CLFNKAIPAH MSEHSVVGIT WDNDRVTVFS D KIYYFYFK NDWSRVATKC YNSGGCAMQY VYEPTYYMLN VTSAGEDGIS YQPCTANCIG YAANVFATEP NGHIPEGFSF NN WFLLSND STLVHGKVVS NQPLLVNCLL AIPKIYGLGQ FFSFNQTIDG VCNGAAVQRA PEALRFNIND ISVILAEGSI VLH TALGTN FSFVCSNSSN PHLATFAIPL GATQVPYYCF LKVDTYNSTV YKFLAVLPPT VREIVITKYG DVYVNGFGYL HLGL LDAVT INFTGHGTDD DVSGFWTIAS TNFVDALIEV QGTAIQRILY CDDPVSQLKC SQVAFDLDDG FYTISSRNLL SHEQP ISFV TLPSFNDHSF VNITVSASFG GHSGANLIAS DTTINGFSSF CVDTRQFTIS LFYNVTNSYG YVSKSQDSNC PFTLQS VND YLSFSKFCVS TSLLASACTI DLFGYPEFGS GVKFTSLYFQ FTKGELITGT PKPFEGVTDV SFMTLDVCTK YTIYGFK GE GIITLTNSSF LAGVYYTSDS GQLLAFKNVT SGAVYSVTPC SFSEQAAYVD DDIVGVISSL SSSTFNSTRE LPGFFYHS N DGSNCTEPVL VYSNIGVCKS GSIGYVPSQS GQVKIAPTVT GNISIPTNFS MSIRTEYLQL YNTPVSVDCA TYVCNGNSR CKQLLTQYTA ACKTIESALQ LSARLESVEV NSMLTISDEA LQLATISSFN GDGYNFTNVL GVSVYDPASG RVVQKRSFIE DLLFNKVVT NGLGTVDEDY KRCSNGRSVA DLVCAQYYSG VMVLPGVVDA EKLHMYSASL IGGMVLGGFT SAAALPFSYA V QARLNYLA LQTDVLQRNQ QLLAESFNSA IGNITSAFES VKEAISQTSK GLNTVAHALT KVQEVVNSQG AALTQLTVQL QH NFQAISS SIDDIYSRLD ILSADAQVDR LITGRLSALN AFVAQTLTKY TEVQASRKLA QQKVNECVKS QSQRYGFCGG DGE HIFSLV QAAPQGLLFL HTVLVPSDFV DVIAIAGLCV NDEIALTLRE PGLVLFTHEL QNHTATEYFV SSRRMFEPRK PTVS DFVQI ESCVVTYVNL TRDQLPDVIP DYIDVNKTLD EILASLPNRT GPSLPLDVFN ATYLNLTGEI ADLEQRSESL RNTTE ELQS LIYNINNTLV DLEWLNRVET GSGYIPEAPR DGQAYVRKDG EWVLLSTFLE NLYFQGGHHH HHHAWSHPQF EK |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 15 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #6: PALMITOLEIC ACID
| 分子 | 名称: PALMITOLEIC ACID / タイプ: ligand / ID: 6 / コピー数: 6 / 式: PAM |
|---|---|
| 分子量 | 理論値: 254.408 Da |
| Chemical component information | 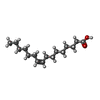 ChemComp-PAM: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.3 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 1.4000000000000001 µm 最小 デフォーカス(公称値): 0.6 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最高: 100.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-64 / 撮影したグリッド数: 1 / 平均露光時間: 12.8 sec. / 平均電子線量: 54.9 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X