+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21390 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of nevanimibe-bound human tetrameric ACAT1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cholesterol (コレステロール) / cholesterol (コレステロール) /  cholesteryl ester / cholesteryl ester /  acyltransferase (アシルトランスフェラーゼ) / acyltransferase (アシルトランスフェラーゼ) /  MBOAT / MBOAT /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 sterol O-acyltransferase / sterol O-acyltransferase /  sterol O-acyltransferase activity / cholesterol O-acyltransferase activity / cholesterol storage / positive regulation of amyloid precursor protein biosynthetic process / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / LDL clearance / cholesterol efflux ... sterol O-acyltransferase activity / cholesterol O-acyltransferase activity / cholesterol storage / positive regulation of amyloid precursor protein biosynthetic process / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / LDL clearance / cholesterol efflux ... sterol O-acyltransferase / sterol O-acyltransferase /  sterol O-acyltransferase activity / cholesterol O-acyltransferase activity / cholesterol storage / positive regulation of amyloid precursor protein biosynthetic process / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / LDL clearance / cholesterol efflux / sterol O-acyltransferase activity / cholesterol O-acyltransferase activity / cholesterol storage / positive regulation of amyloid precursor protein biosynthetic process / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / LDL clearance / cholesterol efflux /  cholesterol binding / macrophage derived foam cell differentiation / cholesterol metabolic process / cholesterol homeostasis / endoplasmic reticulum membrane / cholesterol binding / macrophage derived foam cell differentiation / cholesterol metabolic process / cholesterol homeostasis / endoplasmic reticulum membrane /  小胞体 / 小胞体 /  生体膜 / identical protein binding 生体膜 / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.67 Å クライオ電子顕微鏡法 / 解像度: 3.67 Å | |||||||||
 データ登録者 データ登録者 | Li X / Long T | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structure of nevanimibe-bound tetrameric human ACAT1. 著者: Tao Long / Yingyuan Sun / Abdirahman Hassan / Xiaofeng Qi / Xiaochun Li /  要旨: Cholesterol is an essential component of mammalian cell membranes, constituting up to 50% of plasma membrane lipids. By contrast, it accounts for only 5% of lipids in the endoplasmic reticulum (ER). ...Cholesterol is an essential component of mammalian cell membranes, constituting up to 50% of plasma membrane lipids. By contrast, it accounts for only 5% of lipids in the endoplasmic reticulum (ER). The ER enzyme sterol O-acyltransferase 1 (also named acyl-coenzyme A:cholesterol acyltransferase, ACAT1) transfers a long-chain fatty acid to cholesterol to form cholesteryl esters that coalesce into cytosolic lipid droplets. Under conditions of cholesterol overload, ACAT1 maintains the low cholesterol concentration of the ER and thereby has an essential role in cholesterol homeostasis. ACAT1 has also been implicated in Alzheimer's disease, atherosclerosis and cancers. Here we report a cryo-electron microscopy structure of human ACAT1 in complex with nevanimibe, an inhibitor that is in clinical trials for the treatment of congenital adrenal hyperplasia. The ACAT1 holoenzyme is a tetramer that consists of two homodimers. Each monomer contains nine transmembrane helices (TMs), six of which (TM4-TM9) form a cavity that accommodates nevanimibe and an endogenous acyl-coenzyme A. This cavity also contains a histidine that has previously been identified as essential for catalytic activity. Our structural data and biochemical analyses provide a physical model to explain the process of cholesterol esterification, as well as details of the interaction between nevanimibe and ACAT1, which may help to accelerate the development of ACAT1 inhibitors to treat related diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21390.map.gz emd_21390.map.gz | 11.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21390-v30.xml emd-21390-v30.xml emd-21390.xml emd-21390.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21390.png emd_21390.png | 41.4 KB | ||
| Filedesc metadata |  emd-21390.cif.gz emd-21390.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21390 http://ftp.pdbj.org/pub/emdb/structures/EMD-21390 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21390 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21390 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21390.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21390.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.833 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ACAT1
| 全体 | 名称: ACAT1 |
|---|---|
| 要素 |
|
-超分子 #1: ACAT1
| 超分子 | 名称: ACAT1 / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Sterol O-acyltransferase 1
| 分子 | 名称: Sterol O-acyltransferase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  sterol O-acyltransferase sterol O-acyltransferase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 65.80125 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MVGEEKMSLR NRLSKSRENP EEDEDQRNPA KESLETPSNG RIDIKQLIAK KIKLTAEAEE LKPFFMKEVG SHFDDFVTNL IEKSASLDN GGCALTTFSV LEGEKNNHRA KDLRAPPEQG KIFIARRSLL DELLEVDHIR TIYHMFIALL ILFILSTLVV D YIDEGRLV ...文字列: MVGEEKMSLR NRLSKSRENP EEDEDQRNPA KESLETPSNG RIDIKQLIAK KIKLTAEAEE LKPFFMKEVG SHFDDFVTNL IEKSASLDN GGCALTTFSV LEGEKNNHRA KDLRAPPEQG KIFIARRSLL DELLEVDHIR TIYHMFIALL ILFILSTLVV D YIDEGRLV LEFSLLSYAF GKFPTVVWTW WIMFLSTFSV PYFLFQHWAT GYSKSSHPLI RSLFHGFLFM IFQIGVLGFG PT YVVLAYT LPPASRFIII FEQIRFVMKA HSFVRENVPR VLNSAKEKSS TVPIPTVNQY LYFLFAPTLI YRDSYPRNPT VRW GYVAMK FAQVFGCFFY VYYIFERLCA PLFRNIKQEP FSARVLVLCV FNSILPGVLI LFLTFFAFLH CWLNAFAEML RFGD RMFYK DWWNSTSYSN YYRTWNVVVH DWLYYYAYKD FLWFFSKRFK SAAMLAVFAV SAVVHEYALA VCLSFFYPVL FVLFM FFGM AFNFIVNDSR KKPIWNVLMW TSLFLGNGVL LCFYSQEWYA RQHCPLKNPT FLDYVRPRSW TCRYVFDYKD DDDK UniProtKB:  Sterol O-acyltransferase 1 Sterol O-acyltransferase 1 |
-分子 #2: OLEIC ACID
| 分子 | 名称: OLEIC ACID / タイプ: ligand / ID: 2 / コピー数: 4 / 式: OLA |
|---|---|
| 分子量 | 理論値: 282.461 Da |
| Chemical component information |  ChemComp-OLA: |
-分子 #3: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 3 / コピー数: 10 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #4: nevanimibe
| 分子 | 名称: nevanimibe / タイプ: ligand / ID: 4 / コピー数: 4 / 式: ROV |
|---|---|
| 分子量 | 理論値: 421.618 Da |
| Chemical component information | 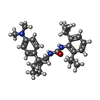 ChemComp-ROV: |
-分子 #5: COENZYME A
| 分子 | 名称: COENZYME A / タイプ: ligand / ID: 5 / コピー数: 2 / 式: COA |
|---|---|
| 分子量 | 理論値: 767.534 Da |
| Chemical component information |  ChemComp-COA: |
-分子 #6: S-{(3R,5R,9R)-1-[(2R,3S,4R,5R)-5-(6-amino-9H-purin-9-yl)-4-hydrox...
| 分子 | 名称: S-{(3R,5R,9R)-1-[(2R,3S,4R,5R)-5-(6-amino-9H-purin-9-yl)-4-hydroxy-3-(phosphonooxy)tetrahydrofuran-2-yl]-3,5,9-trihydroxy-8,8-dimethyl-3,5-dioxido-10,14-dioxo-2,4,6-trioxa-11,15-diaza- ...名称: S-{(3R,5R,9R)-1-[(2R,3S,4R,5R)-5-(6-amino-9H-purin-9-yl)-4-hydroxy-3-(phosphonooxy)tetrahydrofuran-2-yl]-3,5,9-trihydroxy-8,8-dimethyl-3,5-dioxido-10,14-dioxo-2,4,6-trioxa-11,15-diaza-3lambda~5~,5lambda~5~-diphosphaheptadecan-17-yl} (9Z)-octadec-9-enethioate (non-preferred name) タイプ: ligand / ID: 6 / コピー数: 2 / 式: 3VV |
|---|---|
| 分子量 | 理論値: 1.03198 KDa |
| Chemical component information |  ChemComp-3VV: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 初期 角度割当 | タイプ: OTHER |
| 最終 角度割当 | タイプ: OTHER |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.67 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 263839 |
 ムービー
ムービー コントローラー
コントローラー














