+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1701 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The molecular mechanism of the multi-tasking kinesin-8 motor | |||||||||
 マップデータ マップデータ | 10A reconstruction of a 15pf microtubule decorated with the motor domain of Kif18A (kinesin 8) in the Apo-state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Molecular motor (分子モーター) / Molecular motor (分子モーター) /  kinesin 8 / ruby-helix kinesin 8 / ruby-helix | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Bos taurus (ウシ) Bos taurus (ウシ) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 10.1 Å クライオ電子顕微鏡法 / 解像度: 10.1 Å | |||||||||
 データ登録者 データ登録者 | Peters C / Brejc K / Belmont L / Bodey A / Lee Y / Yu M / Ramchandani S / Guo J / Lichtsteiner S / Wood KW ...Peters C / Brejc K / Belmont L / Bodey A / Lee Y / Yu M / Ramchandani S / Guo J / Lichtsteiner S / Wood KW / Sakowicz R / Hartman J / Moores C | |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2010 ジャーナル: EMBO J / 年: 2010タイトル: Insight into the molecular mechanism of the multitasking kinesin-8 motor. 著者: Carsten Peters / Katjuša Brejc / Lisa Belmont / Andrew J Bodey / Yan Lee / Ming Yu / Jun Guo / Roman Sakowicz / James Hartman / Carolyn A Moores /  要旨: Members of the kinesin-8 motor class have the remarkable ability to both walk towards microtubule plus-ends and depolymerise these ends on arrival, thereby regulating microtubule length. To analyse ...Members of the kinesin-8 motor class have the remarkable ability to both walk towards microtubule plus-ends and depolymerise these ends on arrival, thereby regulating microtubule length. To analyse how kinesin-8 multitasks, we studied the structure and function of the kinesin-8 motor domain. We determined the first crystal structure of a kinesin-8 and used cryo-electron microscopy to calculate the structure of the microtubule-bound motor. Microtubule-bound kinesin-8 reveals a new conformation compared with the crystal structure, including a bent conformation of the α4 relay helix and ordering of functionally important loops. The kinesin-8 motor domain does not depolymerise stabilised microtubules with ATP but does form tubulin rings in the presence of a non-hydrolysable ATP analogue. This shows that, by collaborating, kinesin-8 motor domain molecules can release tubulin from microtubules, and that they have a similar mechanical effect on microtubule ends as kinesin-13, which enables depolymerisation. Our data reveal aspects of the molecular mechanism of kinesin-8 motors that contribute to their unique dual motile and depolymerising functions, which are adapted to control microtubule length. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1701.map.gz emd_1701.map.gz | 47.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1701-v30.xml emd-1701-v30.xml emd-1701.xml emd-1701.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  4db-apyrase_500.tif 4db-apyrase_500.tif | 732.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1701 http://ftp.pdbj.org/pub/emdb/structures/EMD-1701 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1701 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1701 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1701.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1701.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 10A reconstruction of a 15pf microtubule decorated with the motor domain of Kif18A (kinesin 8) in the Apo-state | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
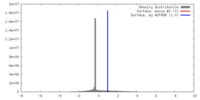
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Microtubule complexed with motor domain of Kif18A (no nucleotide ...
| 全体 | 名称: Microtubule complexed with motor domain of Kif18A (no nucleotide state) |
|---|---|
| 要素 |
|
-超分子 #1000: Microtubule complexed with motor domain of Kif18A (no nucleotide ...
| 超分子 | 名称: Microtubule complexed with motor domain of Kif18A (no nucleotide state) タイプ: sample / ID: 1000 / Number unique components: 3 |
|---|
-分子 #1: Motor domain of Kif18A
| 分子 | 名称: Motor domain of Kif18A / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Motor domain of Kif18A / 詳細: Contains the N-terminal 355 aa / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Cytoplasm Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Cytoplasm |
| 組換発現 | 生物種:  Escherichia coli Rosetta DE3 (大腸菌) Escherichia coli Rosetta DE3 (大腸菌) |
-分子 #2: Alpha tubulin
| 分子 | 名称: Alpha tubulin / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Alpha tubulin / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) / 組織: brain / 細胞中の位置: Cytoplasm Bos taurus (ウシ) / 組織: brain / 細胞中の位置: Cytoplasm |
-分子 #3: Beta tubulin
| 分子 | 名称: Beta tubulin / タイプ: protein_or_peptide / ID: 3 / Name.synonym: Beta tubulin / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) / 組織: Brain / 細胞中の位置: Cytoplasm Bos taurus (ウシ) / 組織: Brain / 細胞中の位置: Cytoplasm |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 詳細: 80mM PIPES, 150mM NaCl, 7mM MgCl2, 1mM EGTA, 1mM beta-mercaptoethanol |
|---|---|
| グリッド | 詳細: 400 mesh copper grid |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Manual plunger Timed resolved state: Vitrified after incubating with Apyrase 手法: Blotted for 1 sec before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: OTHER / Cs: 2.0 mm / 最大 デフォーカス(公称値): 3.3 µm / 最小 デフォーカス(公称値): 0.92 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: cryo-holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| アライメント法 | Legacy - 非点収差: Objective lense stigmatism was corrected at 150,000 times magnification |
| 詳細 | Low dose |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 50 / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 12 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Phase flipping, Wiener |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 10.1 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Ruby-Helix 詳細: Fourier Bessel Synthesis using data from 25 microtubules (36000 asymmetric units) |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)