+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1534 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EcoKI type I RM methyltransferase with DNA mimic Ocr. Negative stain 3D. | |||||||||
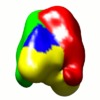
 マップデータ マップデータ | Negative stain EM reconstruction of M.EcoKI-Ocr (Two different models submitted to PDB: 2Y7C, 2Y7H) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | EcoKI / methyltransferase / type I restriction / Ocr / HsdS / HsdM / T7 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報type I site-specific deoxyribonuclease complex / protein binding / symbiont-mediated evasion of host restriction-modification system / N-methyltransferase activity / site-specific DNA-methyltransferase (adenine-specific) / site-specific DNA-methyltransferase (adenine-specific) activity / DNA restriction-modification system / methylation / symbiont-mediated suppression of host innate immune response / virus-mediated perturbation of host defense response ...type I site-specific deoxyribonuclease complex / protein binding / symbiont-mediated evasion of host restriction-modification system / N-methyltransferase activity / site-specific DNA-methyltransferase (adenine-specific) / site-specific DNA-methyltransferase (adenine-specific) activity / DNA restriction-modification system / methylation / symbiont-mediated suppression of host innate immune response / virus-mediated perturbation of host defense response / DNA binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |    Enterobacteria phage T7 (ファージ) Enterobacteria phage T7 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 18.0 Å | |||||||||
 データ登録者 データ登録者 | Kennaway CK / Obarska-Kosinska A / White JH / Tuszynska I / Cooper LP / Bujnicki JM / Trinick J / Dryden DTF | |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2009 ジャーナル: Nucleic Acids Res / 年: 2009タイトル: The structure of M.EcoKI Type I DNA methyltransferase with a DNA mimic antirestriction protein. 著者: Christopher K Kennaway / Agnieszka Obarska-Kosinska / John H White / Irina Tuszynska / Laurie P Cooper / Janusz M Bujnicki / John Trinick / David T F Dryden /  要旨: Type-I DNA restriction-modification (R/M) systems are important agents in limiting the transmission of mobile genetic elements responsible for spreading bacterial resistance to antibiotics. EcoKI, a ...Type-I DNA restriction-modification (R/M) systems are important agents in limiting the transmission of mobile genetic elements responsible for spreading bacterial resistance to antibiotics. EcoKI, a Type I R/M enzyme from Escherichia coli, acts by methylation- and sequence-specific recognition, leading to either methylation of DNA or translocation and cutting at a random site, often hundreds of base pairs away. Consisting of one specificity subunit, two modification subunits, and two DNA translocase/endonuclease subunits, EcoKI is inhibited by the T7 phage antirestriction protein ocr, a DNA mimic. We present a 3D density map generated by negative-stain electron microscopy and single particle analysis of the central core of the restriction complex, the M.EcoKI M(2)S(1) methyltransferase, bound to ocr. We also present complete atomic models of M.EcoKI in complex with ocr and its cognate DNA giving a clear picture of the overall clamp-like operation of the enzyme. The model is consistent with a large body of experimental data on EcoKI published over 40 years. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1534.map.gz emd_1534.map.gz | 329.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1534-v30.xml emd-1534-v30.xml emd-1534.xml emd-1534.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1534.gif 1534.gif | 38.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1534 http://ftp.pdbj.org/pub/emdb/structures/EMD-1534 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1534 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1534 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1534_validation.pdf.gz emd_1534_validation.pdf.gz | 205.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1534_full_validation.pdf.gz emd_1534_full_validation.pdf.gz | 204.2 KB | 表示 | |
| XML形式データ |  emd_1534_validation.xml.gz emd_1534_validation.xml.gz | 4.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1534 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1534 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1534 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1534 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1534.map.gz / 形式: CCP4 / 大きさ: 422.9 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1534.map.gz / 形式: CCP4 / 大きさ: 422.9 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain EM reconstruction of M.EcoKI-Ocr (Two different models submitted to PDB: 2Y7C, 2Y7H) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.125 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli M.EcoKI (M2S1) bound to dimeric ocr from T7 phage
| 全体 | 名称: E. coli M.EcoKI (M2S1) bound to dimeric ocr from T7 phage |
|---|---|
| 要素 |
|
-超分子 #1000: E. coli M.EcoKI (M2S1) bound to dimeric ocr from T7 phage
| 超分子 | 名称: E. coli M.EcoKI (M2S1) bound to dimeric ocr from T7 phage タイプ: sample / ID: 1000 / 詳細: stained with 1pc uranyl acetate / 集合状態: HsdS-(HsdM)2-(ocr)2 / Number unique components: 5 |
|---|---|
| 分子量 | 理論値: 200 KDa |
-分子 #1: HsdS
| 分子 | 名称: HsdS / タイプ: protein_or_peptide / ID: 1 / Name.synonym: EcoKI specificity subunit / コピー数: 1 / 集合状態: Monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 51.4 KDa |
| 配列 | GO: protein binding InterPro: Type I restriction modification DNA specificity domain |
-分子 #2: HsdM
| 分子 | 名称: HsdM / タイプ: protein_or_peptide / ID: 2 / Name.synonym: EcoKI methylase subunit / コピー数: 2 / 集合状態: Dimer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.3 KDa |
| 配列 | GO: protein binding / InterPro: DNA methylase, adenine-specific |
-分子 #3: 0.3 gene
| 分子 | 名称: 0.3 gene / タイプ: protein_or_peptide / ID: 3 / Name.synonym: ocr, 0.3 gene, from T7 phage / コピー数: 2 / 集合状態: Dimer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T7 (ファージ) / 別称: phage T7 Enterobacteria phage T7 (ファージ) / 別称: phage T7 |
| 分子量 | 理論値: 13.8 KDa |
| 配列 | InterPro: Protein Ocr |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.050 mg/mL |
|---|---|
| 緩衝液 | pH: 4.7 / 詳細: 20mM Tris-Cl, 100 mM NaCl, |
| 染色 | タイプ: NEGATIVE 詳細: Protein was adsorbed onto UV treated carbon for 2 mins, blotted, then 1% uranyl acetate solution was applied for 1 min then blotted, twice. |
| グリッド | 詳細: 400 mesh copper |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | JEOL 1200EX |
| 温度 | 平均: 294 K |
| アライメント法 | Legacy - 非点収差: Corrected at 80,000x |
| 詳細 | Customised JEOL 1200 EX microscope, low dose mode. |
| 日付 | 2008年2月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.25 µm / 実像数: 12 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 14 |
| 電子線 | 加速電圧: 80 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 0.87 µm / 最小 デフォーカス(公称値): 0.275 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry / 試料ホルダーモデル: OTHER |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| Microscopy ID | 2 |
|---|---|
| 顕微鏡 | JEOL 1200EX |
| 温度 | 平均: 294 K |
| アライメント法 | Legacy - 非点収差: Corrected at 80,000x |
| 詳細 | Customised JEOL 1200 EX microscope, low dose mode. |
| 日付 | 2008年2月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.25 µm / 実像数: 12 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 14 |
| 電子線 | 加速電圧: 80 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 0.87 µm / 最小 デフォーカス(公称値): 0.275 µm / 倍率(公称値): 40000 |
| 試料ステージ | 試料ホルダー: Side entry / 試料ホルダーモデル: OTHER |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: Situs |
| 詳細 | Protocol: Rigid body. atomic model of whole complex (M2,S1,ocr2) fitted as single rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-2y7c:  PDB-2y7h: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称: Situs |
| 詳細 | Protocol: Rigid body. HsdS homology model based on 1yf2 used. Atomic model of whole complex (M2,S1,ocr2) fitted as single rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-2y7c:  PDB-2y7h: |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F / Chain - #6 - Chain ID: G / Chain - #7 - Chain ID: H |
|---|---|
| ソフトウェア | 名称: Situs |
| 詳細 | Protocol: Rigid body. Modified version of hsdM pdb used with alternative chain trace. Atomic model of whole complex (M2,S1,ocr2) fitted as single rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-2y7c:  PDB-2y7h: |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera