+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11201 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | S-shaped FtsH dodecamer of A. aeolicus with intertwined N-terminal domains in LMNG micelle | |||||||||
 マップデータ マップデータ | S-shaped FtsH dodecamer of A. aeolicus with intertwined N-terminal domains | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
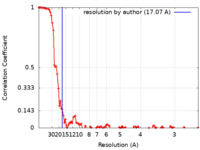
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 17.07 Å クライオ電子顕微鏡法 / 解像度: 17.07 Å | |||||||||
 データ登録者 データ登録者 | Carvalho V / Prabudiansyah I / Kovacik L / Chami M / Kieffer R / van der Valk R / de Lange N / Engel A / Aubin-Tam M-E | |||||||||
| 資金援助 |  オランダ, オランダ,  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2021 ジャーナル: J Biol Chem / 年: 2021タイトル: The cytoplasmic domain of the AAA+ protease FtsH is tilted with respect to the membrane to facilitate substrate entry. 著者: Vanessa Carvalho / Irfan Prabudiansyah / Lubomir Kovacik / Mohamed Chami / Roland Kieffer / Ramon van der Valk / Nick de Lange / Andreas Engel / Marie-Eve Aubin-Tam /   要旨: AAA+ proteases are degradation machines that use ATP hydrolysis to unfold protein substrates and translocate them through a central pore toward a degradation chamber. FtsH, a bacterial membrane- ...AAA+ proteases are degradation machines that use ATP hydrolysis to unfold protein substrates and translocate them through a central pore toward a degradation chamber. FtsH, a bacterial membrane-anchored AAA+ protease, plays a vital role in membrane protein quality control. How substrates reach the FtsH central pore is an open key question that is not resolved by the available atomic structures of cytoplasmic and periplasmic domains. In this work, we used both negative stain TEM and cryo-EM to determine 3D maps of the full-length Aquifex aeolicus FtsH protease. Unexpectedly, we observed that detergent solubilization induces the formation of fully active FtsH dodecamers, which consist of two FtsH hexamers in a single detergent micelle. The striking tilted conformation of the cytosolic domain in the FtsH dodecamer visualized by negative stain TEM suggests a lateral substrate entrance between the membrane and cytosolic domain. Such a substrate path was then resolved in the cryo-EM structure of the FtsH hexamer. By mapping the available structural information and structure predictions for the transmembrane helices to the amino acid sequence we identified a linker of ∼20 residues between the second transmembrane helix and the cytosolic domain. This unique polypeptide appears to be highly flexible and turned out to be essential for proper functioning of FtsH as its deletion fully eliminated the proteolytic activity of FtsH. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11201.map.gz emd_11201.map.gz | 8.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11201-v30.xml emd-11201-v30.xml emd-11201.xml emd-11201.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11201_fsc.xml emd_11201_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11201.png emd_11201.png | 69.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11201 http://ftp.pdbj.org/pub/emdb/structures/EMD-11201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11201 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11201.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11201.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | S-shaped FtsH dodecamer of A. aeolicus with intertwined N-terminal domains | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.28 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : FtsH protease of A. aeolicus
| 全体 | 名称: FtsH protease of A. aeolicus |
|---|---|
| 要素 |
|
-超分子 #1: FtsH protease of A. aeolicus
| 超分子 | 名称: FtsH protease of A. aeolicus / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換プラスミド: pET22a Escherichia coli (大腸菌) / 組換プラスミド: pET22a |
| 分子量 | 実験値: 427 KDa |
-分子 #1: A. aeolicus FtsH protease
| 分子 | 名称: A. aeolicus FtsH protease / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MNALKNFFIW AIIIGAAIVA FNLFEGKREF TTKVSLNEVV KLVEEGKVSY AEVRGNTAII QTKDGQKLE VTLPPNTNLV DKMVEKGVRV EVANPEPPGG WLVNVFLSWL PILFFIGIWI F LLRQMSGG GNVNRAFNFG KSRAKVYIEE KPKVTFKDVA GIEEVKEEVK ...文字列: MNALKNFFIW AIIIGAAIVA FNLFEGKREF TTKVSLNEVV KLVEEGKVSY AEVRGNTAII QTKDGQKLE VTLPPNTNLV DKMVEKGVRV EVANPEPPGG WLVNVFLSWL PILFFIGIWI F LLRQMSGG GNVNRAFNFG KSRAKVYIEE KPKVTFKDVA GIEEVKEEVK EIIEYLKDPV KF QKLGGRP PKGVLLYGEP GVGKTLLAKA IAGEAHVPFI SVSGSDFVEM FVGVGAARVR DLF ETAKKH APCIIFIDEI DAVGRARGAI PVGGGHDERE QTLNQLLVEM DGFDTSDGII VIAA TNRPD ILDPALLRPG RFDRQIFIPK PDVRGRYEIL KVHARNKKLA KDVDLEFVAR ATPGF TGAD LENLLNEAAL LAARKGKEEI TMEEIEEALD RITMGLERKG MTISPKEKEK IAIHEA GHA LMGLVSDDDD KVHKISIIPR GMALGVTQQL PIEDKHIYDK KDLYNKILVL LGGRAAE EV FFGKDGITTG AENDLQRATD LAYRMVSMWG MSDKVGPIAI RRVANPFLGG MTTAVDTS P DLLREIDEEV KRIITEQYEK AKAIVEEYKE PLKAVVKKLL EKETITCEEF VEVFKLYGI ELKDKCKKEE LFDKDRKSEE NKELKSEEVK EEVV |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.005 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.5 µm 最小 デフォーカス(補正後): 0.7000000000000001 µm 倍率(補正後): 78247 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD  Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 215000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 90.0 K / 最高: 100.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3760 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-35 / 撮影したグリッド数: 1 / 実像数: 3993 / 平均露光時間: 16.0 sec. / 平均電子線量: 53.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー